【论文标题】3D-Transformer: Molecular Representation with Transformer in 3D Space
【作者团队】Fang Wu, Qiang Zhang, Dragomir Radev, Jiyu Cui, Wen Zhang, Huabin Xing, Ningyu Zhang, Huajun Chen
【发表时间】2021/10/04
【机 构】哥伦比亚大学、浙大、耶鲁
【论文链接】https://arxiv.org/abs/2110.01191v1
【代码链接】 https://github.com/smiles724/3D-Transformer
三维空间结构对于确定分子的特性非常重要。最近的论文使用几何深度学习来表征分子和预测特性。然而,这些论文在捕捉输入原子的长距离依赖性方面计算成本很高,而且没有考虑到原子间距离的不均匀性,因此无法学习不同尺度上的上下文依赖性表征。为了解决这些问题,本文引入了3D-Transformer,这是分子表征的Transformer的一个包含了三维空间信息的变种。3D-Transformer在原子间有直接联系的全连接图上运行。为了应对原子间距离的不均匀性,本文开发了一个多尺度的自注意力模块,利用局部细粒度的模式来增加背景尺度。由于不同大小的分子依赖于不同种类的空间特征,本文设计了一个自适应位置编码模块,对小分子和大分子采用不同的位置编码方法。最后,为了从原子嵌入中获得分子表征,本文提出了一种关注最远点采样算法,该算法在关注分数的帮助下选择一部分原子,克服了虚拟节点和以前距离主导的下采样方法的障碍。本文在三个重要的科学领域验证了3D-Transformer:量子化学、材料科学和蛋白质组学。本文的实验表明,在晶体性质预测任务和蛋白质-配体结合亲和力预测任务中,3D-Transformer比最先进的模型有明显的改进,并在量子化学分子数据集中表现出更好或有竞争力的性能。这项工作提供了明确的证据,表明生化任务可以从三维分子表征中获益,不同的任务需要不同的位置编码方法。
本文的主要内容为:
- 据本文所知,3D-Transformer是第一个将三维空间信息纳入分子表征学习的Transformer变体。
- 所提出的多尺度自注意力是一种新颖的自注意力的实现方式,具有不断递增的上下文尺度,可以从非均匀距离的原子中提取局部模式。
- 自适应位置编码(ATPE)方法根据新设计的结构复杂性测量指标,采用不同的位置编码策略。对于小而简单的分子,本文在ATPE中开发了一种旋转不变的卷积位置编码方法,以线性计算时间成本来编码每个原子和它们的邻居的相对距离,并使用绝对位置编码方法来描绘大而复杂的分子的三维构象。
- 为了获得基于原子特征的分子表征,本文设计了一个简单而有效的下采样算法,名为注意力最远点采样,该算法同时考虑了每个原子的空间距离和重要性。
- 本文在量子化学、材料科学和蛋白质组学等领域进行了广泛的实验,并在一些基准上显示出明显的改进。本文还表明,3D-Transformer对多种分子构象具有鲁棒性。
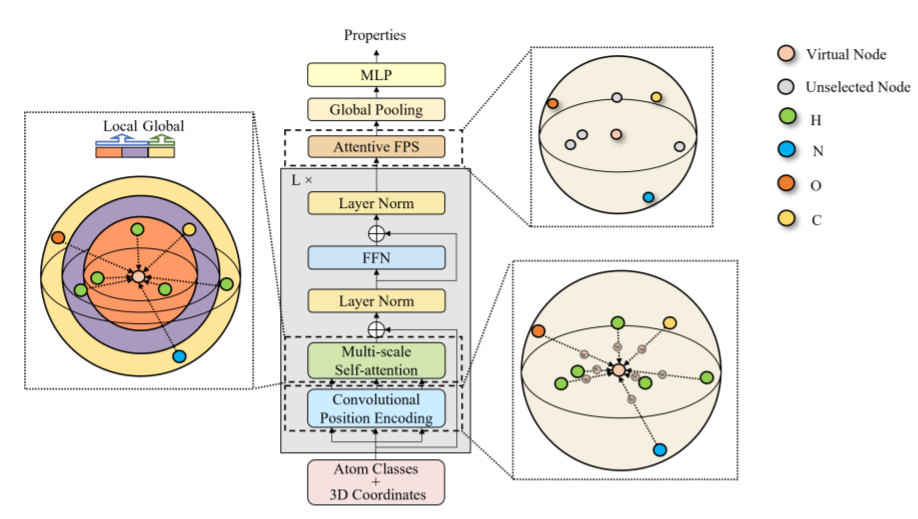
上图展示了分子3DTransformer的整体架构,局部特征显示为紫色和橙色;黄色对应的是全局特征。为了将Transformer架构定制为具有三维空间信息的分子表征,本文开发了三个主要的改进:一个自适应位置编码模块、一个多尺度自注意力模块和一个注意力最远点采样模块
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢