【论文标题】Geometric Transformers for Protein Interface Contact Prediction
【作者团队】Alex Morehead, Chen Chen, Jianlin Cheng
【发表时间】2021/10/06
【机 构】密苏里大学
【论文链接】https://arxiv.org/abs/2110.02423v1
【代码链接】https://github.com/BioinfoMachineLearning/DeepInteract
预测蛋白质之间界面接触的计算方法在药物发现中很重要,因为它们可以显著提高相关方法的准确性,如蛋白质-蛋白质对接、蛋白质功能分析和其他蛋白质生物信息学的计算方法。在这项工作中,本文提出了几何Transformer,一种新型的几何进化图Transformer,用于旋转变换不变的蛋白质界面接触预测,被整合在DeepInteract,一个端到端的预测管道中。DeepInteract以两个蛋白质的三级结构为输入,预测特定的蛋白质界面接触。在严格的基准测试中,DeepInteract对来自新的互动蛋白质结构增强数据库(DIPS-Plus)和第13和第14届CASP-CAPRI的蛋白质复合物任务上分别达到17%和13%的最高L/5精度。在此过程中,DeepInteract以几何Transformer作为其基于图的架构,除了与DeepInteract兼容的其他基于图的神经网络架构外,还优于现有的界面接触预测方法,从而验证了几何Transformer在学习丰富的几何特征方面的有效性,可用于三维蛋白质结构的下游任务。
本文的主要内容为:
- 本文提供了第一个应用于蛋白质界面接触预测的图自注意力的例子,展示了其在学习蛋白质几何结构表征方面的有效应用,以便在下游任务中加以利用。
- 本文提出了新的几何Transformer,可用于三维蛋白质结构和其他三维图形的任务。对于界面接触预测的问题,本文训练几何Transformer,使其与蛋白质序列和协同进化特征同时演化出蛋白质结构的几何表征,用于预测链间残基-残基的接触。在此过程中,本文还展示了最近发布的互动蛋白质结构增强型数据库(DIPS-Plus)在界面预测方面的优点。
- 本文对具有挑战性的蛋白质复合体目标的实验表明,本文提出的方法DeepInteract在界面接触预测方面取得了最先进的结果。通过这些实验,本文还强调需要更透明和适合问题的指标,以及对数据集交叉验证分区进行更严格的基于序列的过滤,以评估蛋白质的机器学习模型的实际性能。
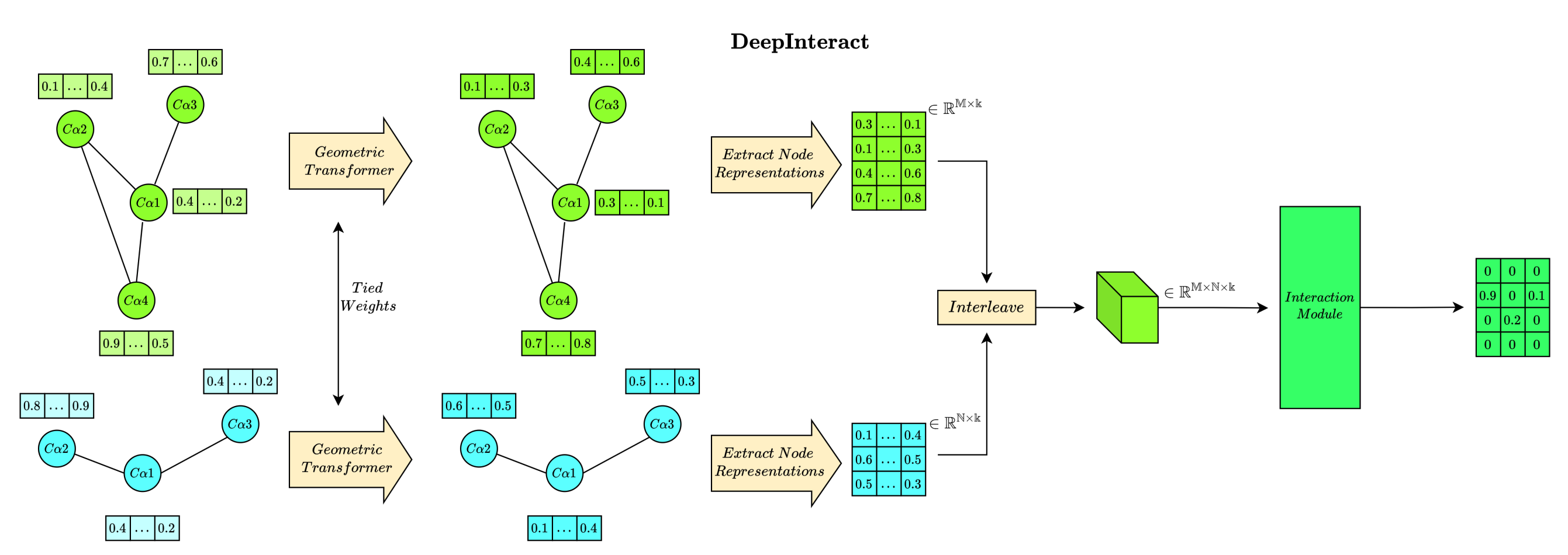
上图显示了DeepInteract的流程,将界面接触预测分成了两个任务。(1) 从成对的残基蛋白图中归纳学习新的节点表征;(2) 对新的节点表征进行卷积,以预测成对的接触概率。
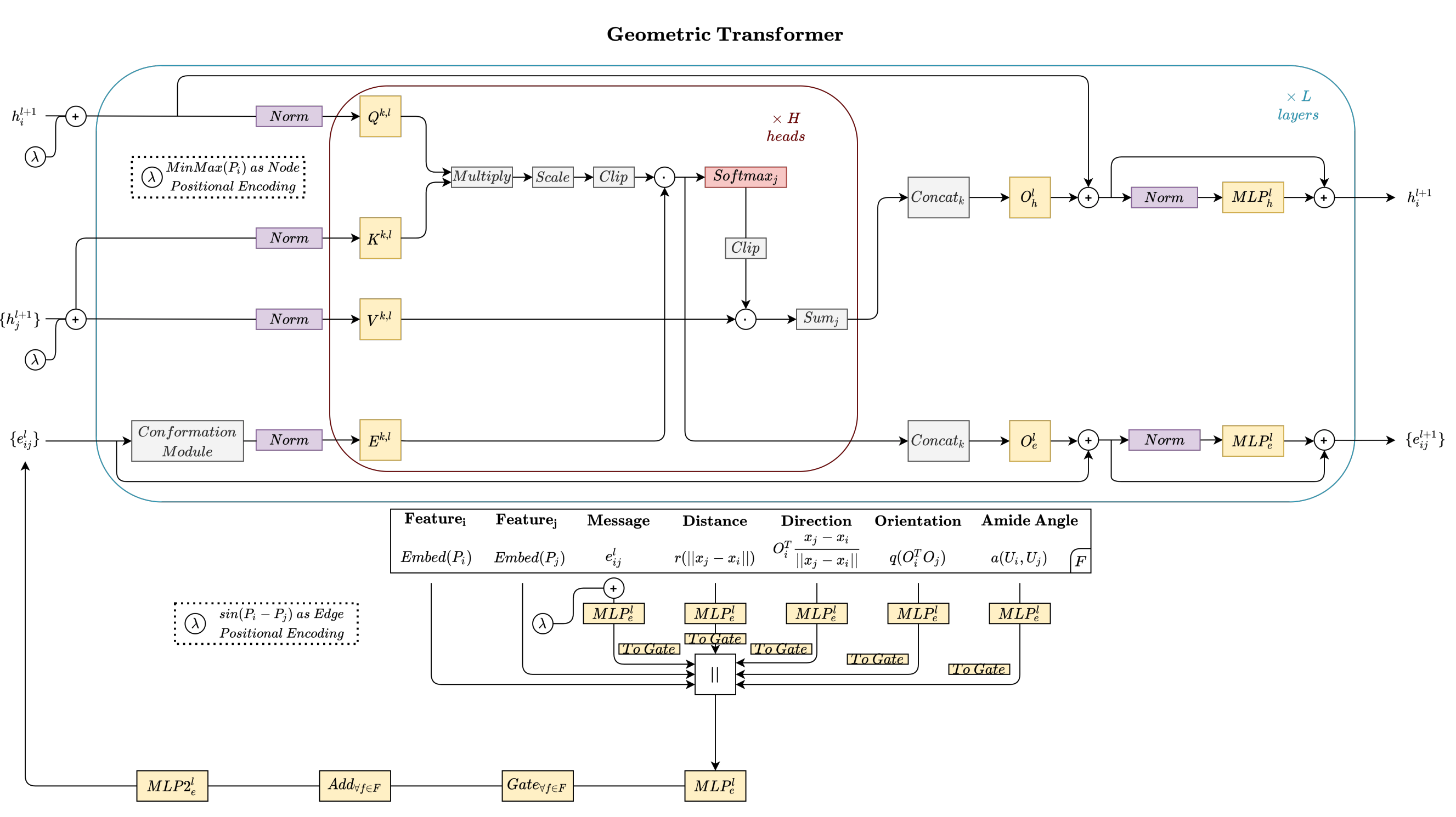
上图展示了几何Transformer架构,其使用图Transformer作为其基础架构,明确设计用于捕获和迭代演化蛋白质几何特征的图神经网络,包括一个边缘初始化模块和几何演变构象模块,以及更微妙的架构改进,如将网络的第一个归一化层移到任何亲和力分数计算之前,以提高训练稳定性。Geometric Transformer是第一个将多头注意力应用于partner-specific蛋白接触界面预测任务的深度学习模型。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢