论文链接:https://doi.org/10.1101/2021.10.17.464750
本文介绍由哈佛医学院Pinello实验室Luca Pinello等人发表于bioRxiv的论文:SIMBA: SIngle-cell eMBedding Along with features. 该论文提出了 SIMBA,这是一种单细胞嵌入方法,可将单细胞及其特征(例如基因、染色质可及区域和转录因子结合序列)嵌入到一个共同的潜在空间中。通过利用细胞和特征的共嵌入,SIMBA 可用于细胞异质性研究、无聚类标记发现、基因调控推断、去除批次效应和多组学数据集成。SIMBA 可应用于 scRNA-seq、scATAC-seq 和多组学数据分析。
SIMBA首先将不同类型的实体如细胞、基因、开放染色质区域和转录因子编码成一个单独的图(如下图),其中节点代表不同的实体,边表示它们之间的关系。然后使用无监督图嵌入方法计算节点的低维表示。这个图嵌入过程利用了PyTorch-BigGraph框架,该框架允许SIMBA扩展到数百万个细胞。由此产生的细胞和特征的联合嵌入不仅重建了细胞的异质性,而且允许以无聚类的方式发现每个单细胞的特征,将细胞类型的特征与非信息特征分离。事实上,实体嵌入之间的接近程度可以提供信息,以了解特征对细胞的潜在重要性,以及发现特征之间的相互作用。当多种类型的特征(如转录组和表观遗传特征)同时嵌入时,SIMBA为研究基因调控以及细胞分化和细胞类型形成的调控机制提供了一种直观的方法。
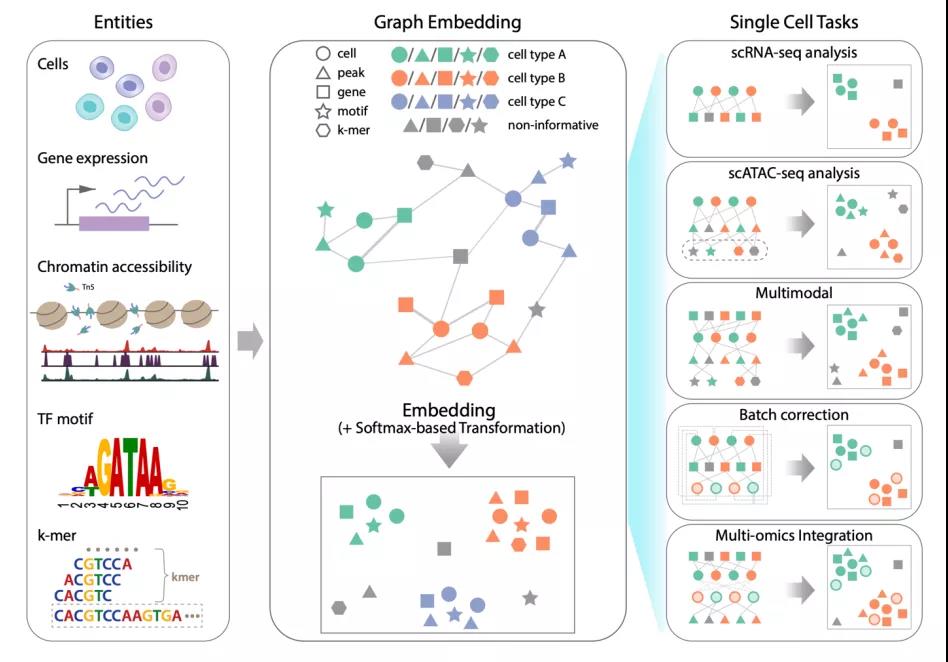 SIMBA模型概述
SIMBA模型概述
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢