1、【Scientific Reports 2020】SkipGNN: Predicting Molecular Interactions
with Skip-Graph Networks
作者:Huang Kexin,Xiao Cao,Glass Lucas,Zitnik Marinka,Sun Jimeng Harvard University
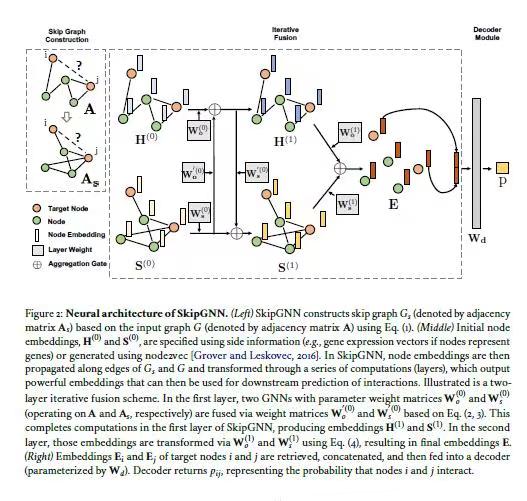
文章导读:目前的图神经网络(GNN)方法都是基于交互节点之间的直接相似性进行优化预测。然而在生物网络中,没有直接互动的节点之间的相似性被证明是非常有用的。SkipGNN不仅通过聚合直接相互作用的信息,还通过聚合二阶相互作用的信息来预测分子相互作用,我们称之为跳跃相似性。与现有的gnn相比, SkipGNN接收来自交互网络中2-hop邻居和直接邻居的消息,并对消息进行非线性变换,获得有用的预测信息。
链接:https://arxiv.org/abs/2004.14949
2、[arxiv1810]Co-Learning Feature Fusion Maps from PET-CT Images of Lung Cancer
author: Ashnil Kumar, Michael Fulham, Dagan Feng, Jinman Kim
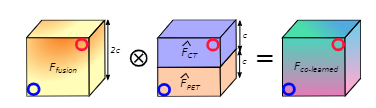
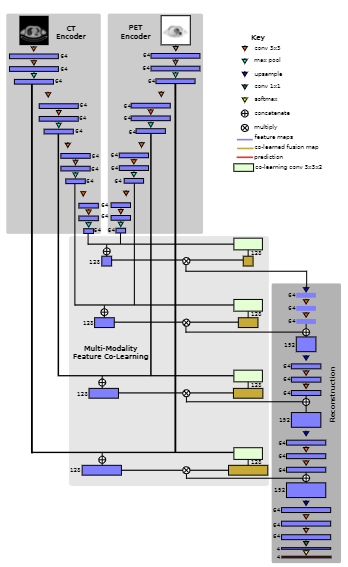
导读:用于计算机辅助诊断应用的多模态正电子发射断层扫描和计算机断层扫描 (PET-CT) 图像的分析需要将 PET 检测异常区域的敏感性与 CT 的解剖定位相结合。当前用于 PET-CT 图像分析的方法要么单独处理模态,要么基于有关图像分析任务的知识融合来自每个模态的信息。这些方法通常不考虑空间变化的视觉特征,这些特征在不同的模态中编码不同的信息,在不同的位置具有不同的优先级。例如,肺部的高异常 PET 摄取对肿瘤检测比心脏中的生理 PET 摄取更有意义。我们的目标是改进多模态 PET-CT 中的互补信息与新的监督卷积神经网络 (CNN) 的融合,该网络学习融合互补信息以进行多模态医学图像分析。我们的 CNN 首先编码特定于模态的特征,然后使用它们来导出空间变化的融合图,该图量化了每个模态特征在不同空间位置的相对重要性。然后将这些融合图与特定于模态的特征图相乘,以获得不同位置的互补多模态信息的表示,然后可用于图像分析。我们使用肺癌的 PET-CT 图像数据集评估了我们的 CNN 检测和分割具有不同融合要求的多个区域的能力。我们将我们的方法与多模态图像融合和分割的基线技术进行了比较。我们的研究结果表明,与最近的 PET-CT 肿瘤分割方法相比,我们的 CNN 具有比融合基线显着更高的前景检测准确度(99.29%,p < 0.05)和显着更高的 Dice 得分(63.85%)。
链接:https://arxiv.org/pdf/1810.02492.pdf
3、论文标题:CuCo:Graph Representation with Curriculum Contrastive Learning(IJCAI2021)
作者:Guanyi Chu,Xiao Wang,chuan Shi,Beijing University of Posts and Telecommunications
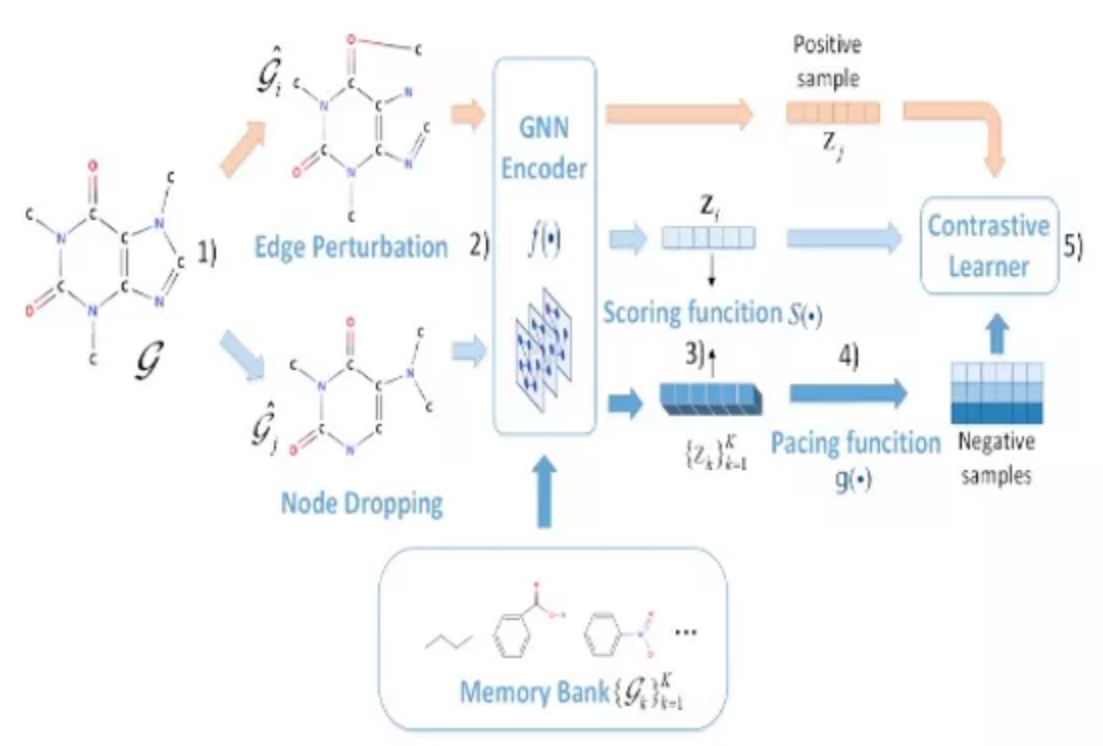
文章导读:图级的表示学习旨在学习整个图的低维表示,在实际应用有着很大影响。本文研究了负样本对图级表示学习的影响,提出了一种新的基于自监督图级表示的课程对比学习框架CuCo。具体来说,我们引入了四种图增强技术来获得图的正样本和负样本,并利用图神经网络来学习它们的表示。然后,我们提出了一个评分函数,将负样本从易到难进行排序,并在每个训练过程中自动选择负样本。我们在15个图分类的真实世界数据集上的大量实验以及参数分析充分表明,我们提出的CuCo在分类和收敛性能方面取得了令人鼓舞的结果。
论文链接:http://www.shichuan.org/doc/111.pdf
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢