AlphaFold2 时代已经到来,彻底改变了结构生物学的工作流程。然而,该算法并没有考虑到影响蛋白质结构和功能的基本修饰。
在这里,约克大学(University of York)和梅努斯大学(Maynooth University)的研究人员讨论如何以相对简单的方式解决这种遗漏,从而对复杂的生物分子系统进行完整的结构预测。研究人员通过将蛋白质糖基化数据库移植到 AlphaFold 模型上,开发了解析蛋白质糖基化修饰的新工具。
该研究以「The case for post-predictional modifications in the AlphaFold Protein Structure Database」为题,于 2021 年 10 月 29 日发布在《Nature Structural & Molecular Biology》。
DeepMind 和 EMBL-EBI 最近发布的 AlphaFold 蛋白质结构数据库标志着结构生物学的重大突破,因为它为全球科学界提供了对来自人类的 20,000 种蛋白质和来自 20 种其他生物相关生物体的蛋白质的高度准确的结构预测。许多从事大分子结构研究的科学家对这一发展感到非常兴奋,但研究人员认为许多人对当前的内容有不可忽视的误解。
特别是,AlphaFold 数据库中的仅蛋白质预测,意味着辅助因子以及最重要的翻译和翻译后修饰被排除在外;这也是可以理解的——由于技术的限制——被排除在外。最相关的共翻译和翻译后修饰之一是蛋白质糖基化——相关性非常明显,正如最近对完全糖基化 SARS-CoV-2 刺突蛋白动力学的研究所表明的那样。
事实上,在这预测的 20,000 种人类蛋白质中,有 50% 到 70% 被认为是糖基化的,但这些都没有在数据库中明显突出显示。通过 AlphaFolds 与 Uniprot 的链接,可以轻松获得关于修改可能性的详细信息,因此研究人员建议用户在下载模型之前检查 Uniprot 上的可用信息。
Uniprot:https://www.uniprot.org
在这个框架内,研究人员认为 AlphaFold 蛋白质结构数据库的模型中没有辅因子和共翻译或翻译后修饰,后续可以通过使用基于序列和结构的比较研究来修复。事实上,在糖基化的特定情况下,DeepMind 实施的算法已经消化了来自蛋白质数据库(PDB)的残基间距离,其中糖基化蛋白质通常表现出完整或部分聚糖结构;因此,AlphaFold 模型以某种方式保留了未建模的修饰(例如蛋白质糖基化)应该出现的空间,这允许将这些结构特征直接移植到模型上。
为了证明这种方法的潜力,研究人员开发了概念验证功能,将蛋白质糖基化从分子动力学获得的结构平衡聚糖块库移植到 AlphaFold 模型上。此任务已自动化并集成到特定于碳水化合物的 Privateer 软件的新 Python 界面中,并且在其 GitHub 存储库上可供所有人使用。
Privateer 软件:https://github.com/glycojones/privateer.git
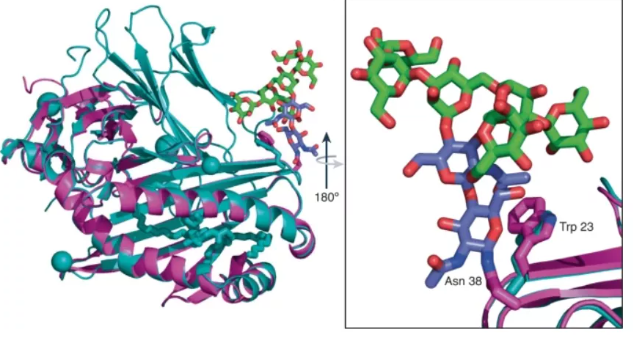
下图显示了人类 T 细胞表面糖蛋白 Cd1b 的 AlphaFold 模型 P29016(以洋红色描绘),叠加在蛋白质的晶体结构 PDB 5WL1 上。后者在昆虫细胞系中表达,它显示了 N-聚糖的特征双核岩藻糖基化。
新工具嫁接到 AlphaFold 模型上的 N-聚糖不仅与可用空间兼容,而且显示出与蛋白质表面的高度互补性,其中 Man6 核心在 CH-π 相互作用中与 Trp 23 相关,如图所示晶体结构。
图示:将 N-聚糖嫁接到 AlphaFold 模型上。(来源:论文)
需要强调的是,这种方法也可能有助于完成数据库中具有其他类型修改的 AlphaFold 模型。例如,AlphaFold 模型 P6887,一种血红蛋白亚基β,包含一个血红素结合位点,其空间刚好容纳一个血红素辅因子。某些结构完成只有通过针对可用结构信息的自动比较分析才可行——例如,共翻译修饰,如豆蔻酰化或 O-GlcNAcylation——而其他如 N-糖基化或色氨酸甘露糖基化,依赖于共有序列,预测将更方便。由于比较研究必须依赖于实验结构信息,因此可以通过将放置的坐标与可用结构信息的叠加进行比较来估计位置不确定性(例如,类似 pLDDT 的分数)。
然而,在蛋白质糖基化的特殊情况下,会有更多的组成问题。事实上,最大的难题是对与每个序列相关的糖型进行很好的估计。由于迁移率和微观异质性等限制因素,实验结构仅提供部分信息,因此应使用其他知识来源(例如,糖组学和分子动力学模拟),尤其是在尝试对全长聚糖进行建模时。目前该团队正在扩展 Privateer 软件以解决这些情况,通过利用糖组学数据库中可用的丰富信息。
总而言之,研究人员认为这些早期结果非常令人鼓舞,可以作为开发者社区的一个集结点来完成和丰富预测的蛋白质模型,并进行可能的修改,使它们发挥最大潜力,并将引领下一代结构生物学研究。
数据链接:https://zenodo.org/record/5564681
论文链接:https://www.nature.com/articles/s41594-021-00680-9
相关报道:https://phys.org/news/2021-11-scientists-ai-protein-sugar.html
内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢