介绍一篇来自中国矿业大学的李政伟副教授、聂茹副教授和中国科学院新疆理化技术研究所的尤著宏教授在2021年合作发表在《Briefings in Bioinformatics》上的一篇文章《A graph auto-encoder model for miRNA-disease associations prediction》。本文提出了一种基于图自动编码器的miRNA-disease关联预测方法。

文章地址:
https://doi.org/10.1093/bib/bbaa240
代码地址:
摘要
动机:有新的证据表明,miRNA的异常表达参与了人类各种复杂疾病的进化和进展。将疾病相关的miRNA作为新的生物标志物,可以促进疾病病理学和临床医学的发展。然而,设计生物学实验来验证疾病相关的miRNAs通常是费时和昂贵的。因此,迫切需要设计有效的计算方法来预测潜在的miRNA与疾病的关联。
结果:受图神经网络在链路预测方面的巨大进展的启发,作者提出了一种新的图自动编码模型,命名为GAEMDA,以端到端方式预测潜在的miRNA与疾病的关联。具体地说,GAEMDA模型采用基于图神经网络的编码器,包含聚合器函数和用于聚合节点邻域信息的多层感知器,来生成miRNA与疾病节点的低维嵌入,实现异构信息的有效融合。然后,将miRNA和疾病的节点嵌入送入到一个双线性解码器中,以预测miRNA和疾病节点之间的潜在联系。各种实验结果表明,GAEMDA模型可以作为一种可靠的工具,指导后续miRNA调控作用的研究。
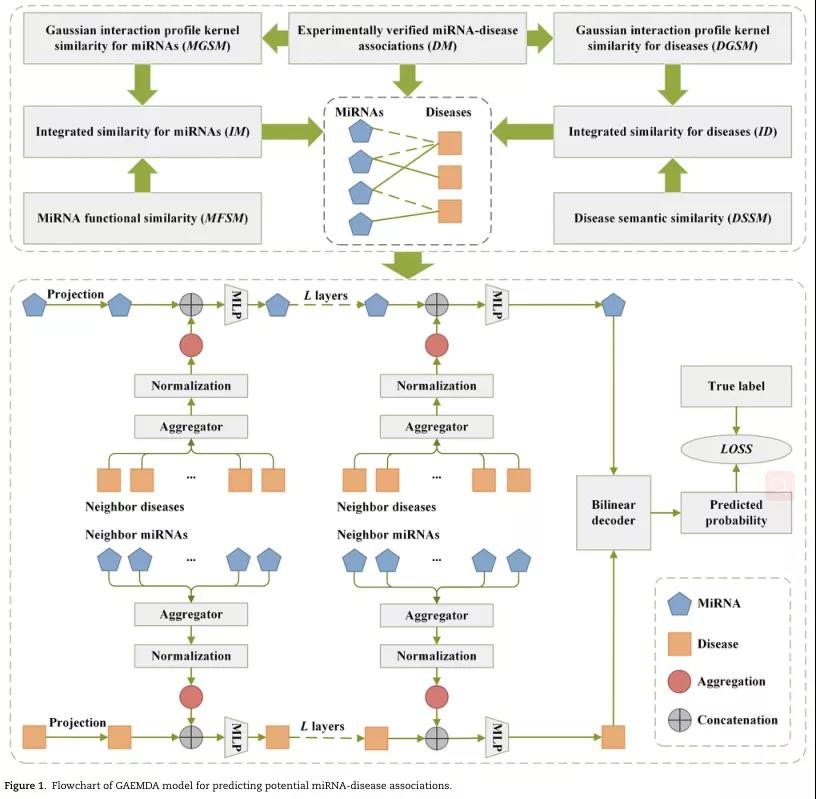
模型的结构图
GAEMDA可以描述为五个步骤:(i)构建miRNA-疾病二部图,(ii)将miRNA和疾病节点投影到同一个向量空间,(iii)应用基于图神经网络的编码器生成miRNA和疾病节点的嵌入,(iv)采用双线性解码器重构二部图中的链路;(v)采用交叉熵损失函数端到端训练整个模型。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢