本文介绍的是由荷兰莱顿药物研究学术中心、西安交通大学电子与信息工程学院和莱顿高级计算机科学研究所联合发表在Journal of Cheminformatics上的研究成果。作者在之前的一项研究中提出了一种名为DrugEx的药物分子生成方法,将探索策略集成到基于RNN的强化学习中,以提高生成分子的多样性。在本文中,作者通过多目标优化扩展DrugEx算法,以生成针对多个靶标或一个特定靶标的类药物分子,同时避免脱靶(本研究中的两个腺苷受体,A1AR和A2AAR,以及钾离子通道hERG)。该模型使用RNN作为智能体(agent),机器学习预测器作为环境,agent和环境都被预先训练,然后在强化学习框架下交互。作者将进化算法的概念融合到模型中,交叉和变异操作由与agent相同的深度学习模型实现。训练期间,agent生成一批SMILES形式的分子。随后,环境提供的所有靶标的亲和力分数将用于构建生成的分子的帕累托排名,该排序采用了非支配排序算法和拥挤距离算法。作者证明了生成的化合物可以对多种靶标进行作用,并具有高效低毒的潜力。

论文链接:
https://doi.org/10.1186/s13321-021-00561-9
代码:
https://github.com/XuhanLiu/DrugEx
为了预测每个生成分子对给定靶标的pChEMBL的平均值(pX,包括pKi、pKd、pIC50或pEC50),作者使用4种不同的机器学习算法构建了QSAR回归模型,即随机森林(RF)、支持向量机(SVM)、偏最小二乘回归(PLS)和多任务深度神经网络(MT-DNN)。
作者将数据集中的所有分子拆分为一系列标记,以构成一个SMILES词汇表。生成模型使用RNN进行构建,包含一层输入层、一层嵌入层、三层循环层和一层输出层。
对生成器进行预训练后,强化学习(RL)训练流程分为四步(如下图):(1)根据生成器计算出的概率,通过逐步采样标记生成一批SMILES;(2)有效的SMILES被解析为分子并编码为描述符,以得到预测的pXs;(3)基于Pareto优化将预测的pXs转化为单个值作为每个分子的奖励;(4)将SMILES序列及其奖励送回生成器,用策略梯度方法进行训练。
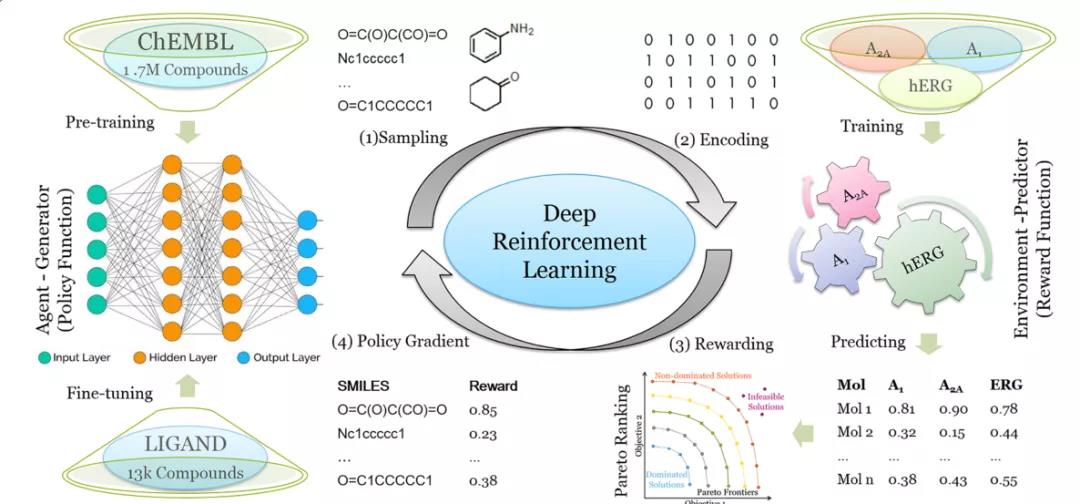
DrugEx2利用强化学习的训练流程
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢