【论文标题】Keeping it Simple: Language Models can learn Complex Molecular Distributions
【作者团队】Daniel Flam-Shepherd, Kevin Zhu, Alán Aspuru-Guzik
【发表时间】2021/12/06
【机 构】多伦多大学
【论文链接】https://arxiv.org/abs/2112.03041v1
分子的预训练深度生成模型已经得到了极大的普及,在相关的数据集上进行训练,这些模型被用来搜索化学空间。生成模型在新型功能化合物的逆向设计中的下游效用取决于它们学习分子训练分布的能力。最简单的例子是语言模型,它采用了循环神经网络的形式,用字符串表征生成分子。更复杂的是图生成模型,它按顺序构建分子图,通常能取得最先进的结果。然而,最近的工作表明,语言模型的能力比曾经认为的更强,特别是在低数据体系下。在这项工作中,作者研究了简单语言模型学习分子分布的能力。为此,本文引入了几个具有挑战性的需要编译特别复杂的分子分布的生成性建模任务。在每个任务中,本文将语言模型的能力与两个广泛使用的图生成模型相比较进行评估。结果表明,语言模型是强大的生成模型,能够熟练地学习复杂的分子分布,并产生比图模型更好的性能。
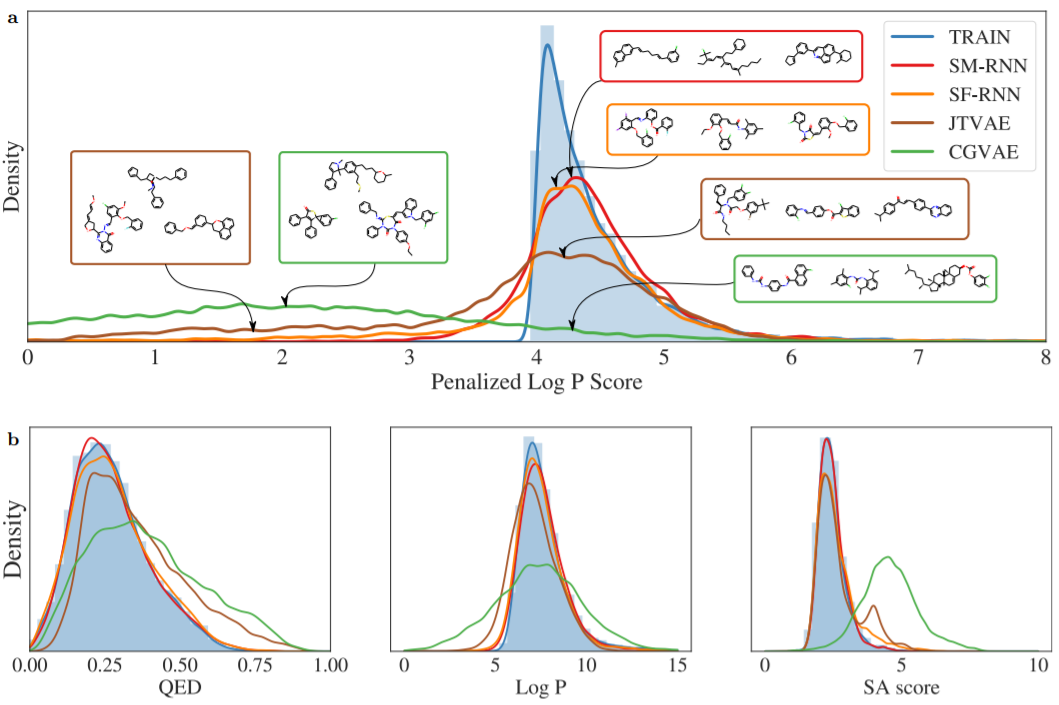
第一项任务为惩罚性LogP任务,最广泛使用的搜索化学空间的基准评估之一,寻找具有高LogP的分子,并以可合成性和不现实的环作为惩罚。作者把这个任务变成一个生成性建模任务,目标是学习具有高惩罚性LogP分数的分子的分布。找到一个具有良好得分的单一分子可能具有挑战性,但是学习从这部分化学空间直接生成,从而使模型产生的每个分子都具有高惩罚性LogP,为标准任务增加了另一层复杂性。
从结果上来看语言模型的表现明显好于图形模型,SELFIES RNN产生的结果与图a中的训练分布稍微接近。CGVAE和JTVAE学习产生了大量的分子,其惩罚后的LogP分数大大低于最低的训练分数。值得注意的是,从图a中显示的这些例子来看,这些得分较低的分子与训练分布的主要模式的分子相当相似,这突出了学习这种分布的难度。在图b中,作者看到JTVAE和CGVAE学习产生了更多比训练数据更大的SA得分的分子。对于LogP,作者看到所有的模型都学习了主要模式,但是RNNs产生了更接近的分布,对于QED也可以看到类似的结果。这些结果延续到了定量指标上。
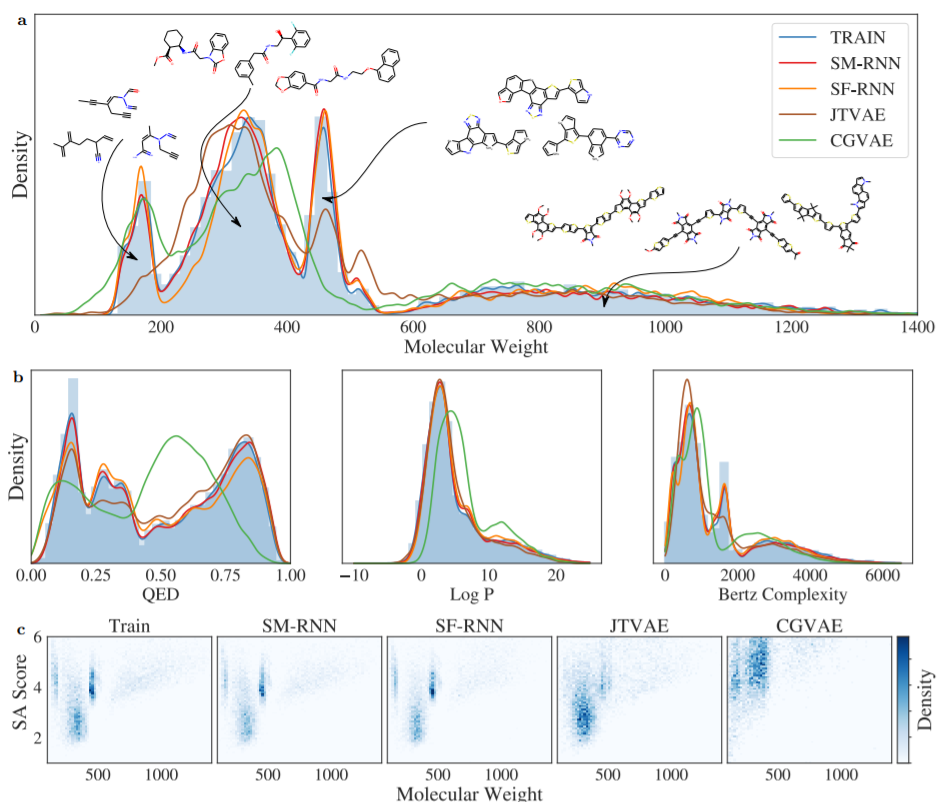
第二个任务为多分布任务,测试模型是否能学习到复合分布中的各个分布,作者通过结合以下子集创建了一个数据集。
1)分子量(MW)≤185的GDB13分子
2)分子量为185≤MW≤425的ZINC分子
3) 哈佛清洁能源项目(CEP)分子,分子量为460≤MW≤600
4)MW≥600的POLYMERS分子。
训练分布中CEP和GDB13占1/3,ZINC和POLYMERS各占1/3,总共200K的训练分子。
在多分布任务中,两个RNN模型都能很好地捕获数据分布,并学习训练分布中的每个模式,。另一方面,JTVAE完全错过了GDB13的第一个模式,然后对ZINC和CEP的学习很差。同样,CGVAE也学习了GDB13,但低估了ZINC,并完全错过了CEP的模式。更多的证据表明,RNN模型更紧密地学习了训练分布,在图c中,CGVAE和JTVAE几乎没有区分出主要模式。此外,RNN模型产生的分子与训练数据更相似。
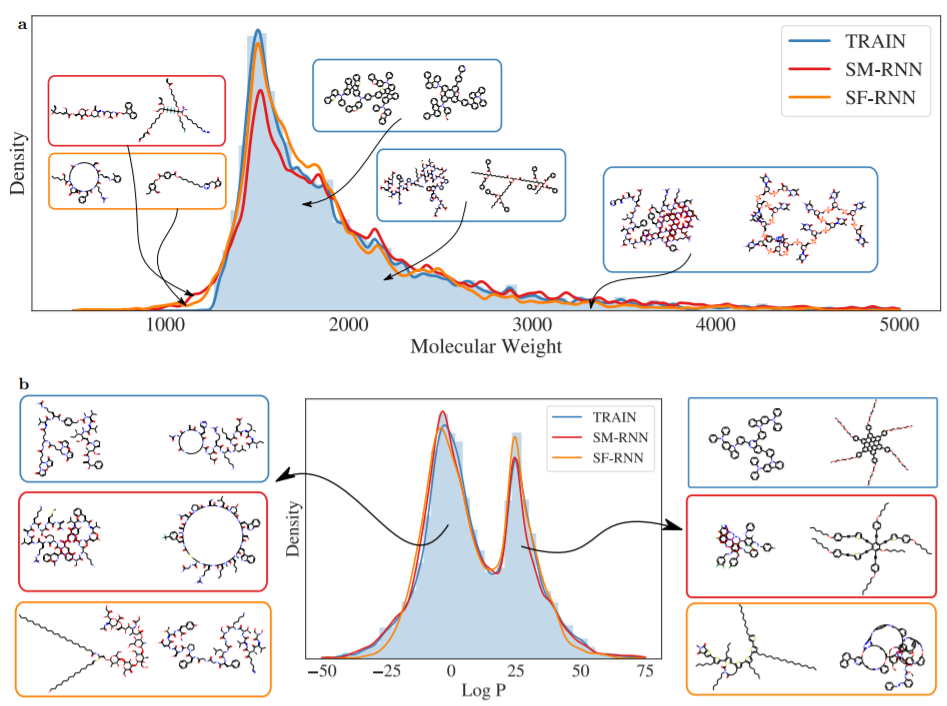
最后一项生成模型任务为大型任务,即测试分子生成模型生成的最大可能分子。作者为此转向PubChem,筛选出有100多个重原子的较大分子,产生了30万个分子。这些是小型生物分子及其他,分子量范围也很广,从1500到5000,但大多数分子属于1500-2000范围。
这项任务对图模型来说是最具挑战性的,两者都没能训练,完全无法学习训练数据。特别是,JTVAE的树状分解算法应用于训练数据产生了一个固定的11000个子结构的词汇。然而,两个RNN模型都能够学习生成与训练数据一样大、一样多的分子。训练分子对应于很长的SMILES和SELFIES字符串表示,在这种情况下,SELFIES字符串提供了额外的优势,SELFIES RNN可以更接近于数据分布(图a)。特别是,用SMILES语法学习有效的分子大大增加了难度,因为这些分子有更多的字符需要生成,模型犯错并产生无效字符串的概率更高。相比之下,生成的SELFIES字符串永远不会是无效的。有趣的是,即使RNN模型生成的分子不在分布范围内,并且比训练分子小得多,它们仍然有类似的子结构,并且与训练分子相似(图a)。此外,训练分子似乎分为两种模式,即LogP值较低和较高的分子(图b):LogP较低的生物大分子,和LogP较高的具有更多环和更长碳链的分子,而RNN都可以学到这两种模型。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢