【论文标题】Multimodal Pre-Training Model for Sequence-based Prediction of Protein-Protein Interaction
【作者团队】Yang Xue, Zijing Liu, Xiaomin Fang, Fan Wang
【发表时间】2021/12/09
【机 构】百度
【论文链接】https://arxiv.org/abs/2112.04814v1
蛋白质-蛋白质相互作用(PPI)是许多生物过程的关键,在这些过程中,两个或多个蛋白质物理地结合在一起以实现其功能。建立PPI模型对许多生物医学应用是有用的,如疫苗设计、抗体治疗和多肽类药物发现。预训练蛋白质模型以学习有效的表征对PPIs来说至关重要,大多数PPI的预训练模型是基于序列的,采用自然语言处理中使用的语言模型来处理氨基酸序列。更先进的工作是利用结构感知的预训练技术,利用已知蛋白质结构的接触图。然而,无论是序列还是接触图都不能完全描述蛋白质的结构和功能,这与PPI问题密切相关。受此启发,我们提出了一个多模态的蛋白质预训练模型,包括三个模态:序列、结构和功能(S2F)。值得注意的是,我们没有使用接触图来学习氨基酸水平的刚性结构,而是用重原子的点云的拓扑复合物来编码结构特征。这使得我们的模型不仅可以学习骨架的结构信息,还可以学习侧链的结构信息。此外,我们的模型还结合了从文献或人工注释中提取的蛋白质功能描述的知识。我们的实验表明,S2F学习的蛋白质嵌入在各种PPI任务中取得了良好的表现,包括跨物种PPI、抗体-抗原亲和力预测、SARS-CoV-2的抗体中和预测,以及突变驱动的结合亲和力变化预测。
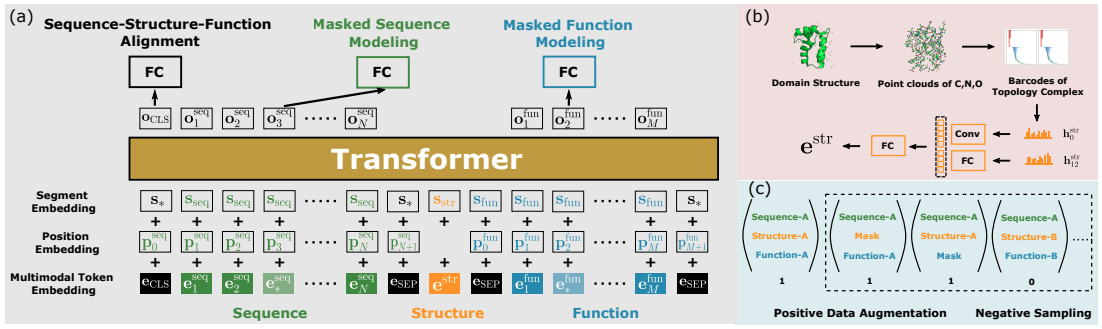
序列-结构-功能(S2F)蛋白质预训练模型的概述。绿色、橙色和蓝色元素分别代表序列、结构和功能模式。a图表示了单流多模态转化器模型的结构和三个预训练任务:序列-结构-功能排列、屏蔽序列建模和屏蔽功能建模。b图表示了从域结构中提取拓扑结构标记e^str的拓扑编码器的管道。c图表示了预训练任务的正向数据增强和负向采样过程的说明。
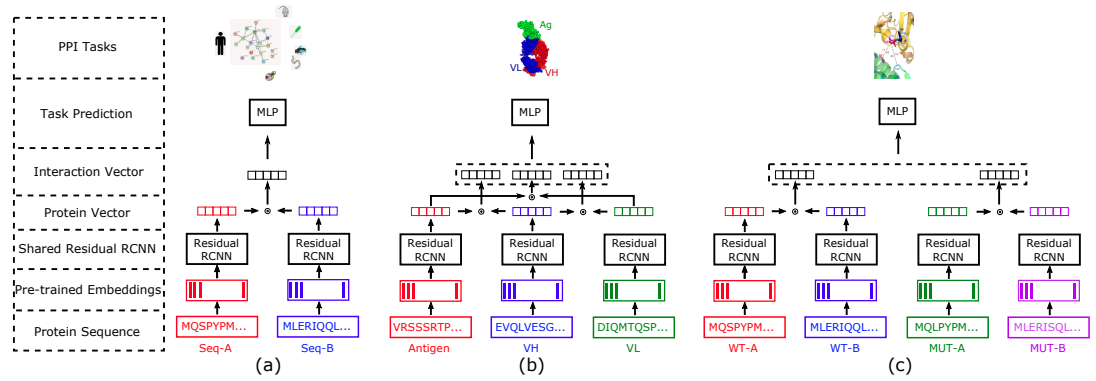
上图展示了用于下游PPI任务的网络的变体。跨物种PPI任务的网络;抗体-抗原相互作用任务的网络;突变驱动的亲和力变化预测任务的网络,Residual RCNN模块的权重在每个具体任务中都是共享的。
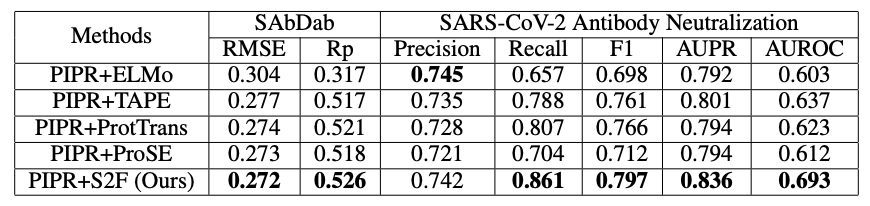
上图展示了部分任务的结果。任务之一抗体-抗原亲和力预测任务的结果。该数据集来自SAbDab数据库,有494个样本。每个例子都有一个抗原序列,一个可变重链,一个可变轻链,以及亲和力标签。任务之二SARS-CoV-2抗体中和任务,预测给定的抗体是否能中和S蛋白的作用,阻碍SARS-CoV-2的感染。该数据集来自CoV-AbDab数据库,有747个阳性样本和330个阴性样本。
对于抗体-抗原亲和力预测任务,SAbDab,我们使用均方根误差(RMSE)和皮尔逊相关度(Rp)来评估性能。对于分类任务,SARS-CoV-2抗体中和,我们使用UROC、AUPR、F1得分、精度和召回率来评估模型。上图中报告了10倍交叉验证平均值。不难看出,S2F模型在回归和分类任务中都击败了其他预训练的嵌入。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢