本文介绍的是由美国加州理工学院生物与生物工程系的David Van Valen、斯坦福大学病理学系的Michael Angelo等研究人员在《Nature Biotechnology》上发表的研究成果。组织成像数据的分析中一个主要挑战是细胞分割,即识别图像中每个细胞的精确边界的任务。在这篇文章中,为了解决这个问题,作者首先构建了一个用于训练分割模型的数据集TissueNet,这其中包括了一百多万个手动标记的细胞。然后作者用TisseNet训练了一种基于深度学习的分割算法Mesmer。通过实验表明,Mesmer比以往的方法更加准确,它能够概括TissueNet中组织类型和成像平台的全部多样性,并且达到了人类水平的表现。Mesmer还能够自动提取关键的细胞特征,如蛋白质信号的亚细胞定位。作者之后对Mesmer进行调整从而使其能够在高度复用的数据集中利用细胞谱系信息,并且还利用这个增强的版本量化了人类妊娠期间细胞形态的变化。
论文链接:
https://www.nature.com/articles/s41587-021-01094-0
在这篇文章中,作者试图为细胞核和全细胞的分割创建一个自动化、简单且可扩展的算法,该算法可以在各种组织类型和成像平台上精确执行。作者认为开发这种算法需要两个创新:
(1)能生成大量像素级别训练数据的可扩展方法。
(2)一个集成的深度学习算法,它使用这些数据来达到人类水平的性能。
为了解决第一个挑战,如图1所示,该团队开发了一种众包的、“人在环路”的方法来分割细胞,其中人和算法协同工作以产生准确的标注,并就此算法创建了 TissueNet,这是一个包含超过 100万对全细胞与核注释的综合分割数据集。
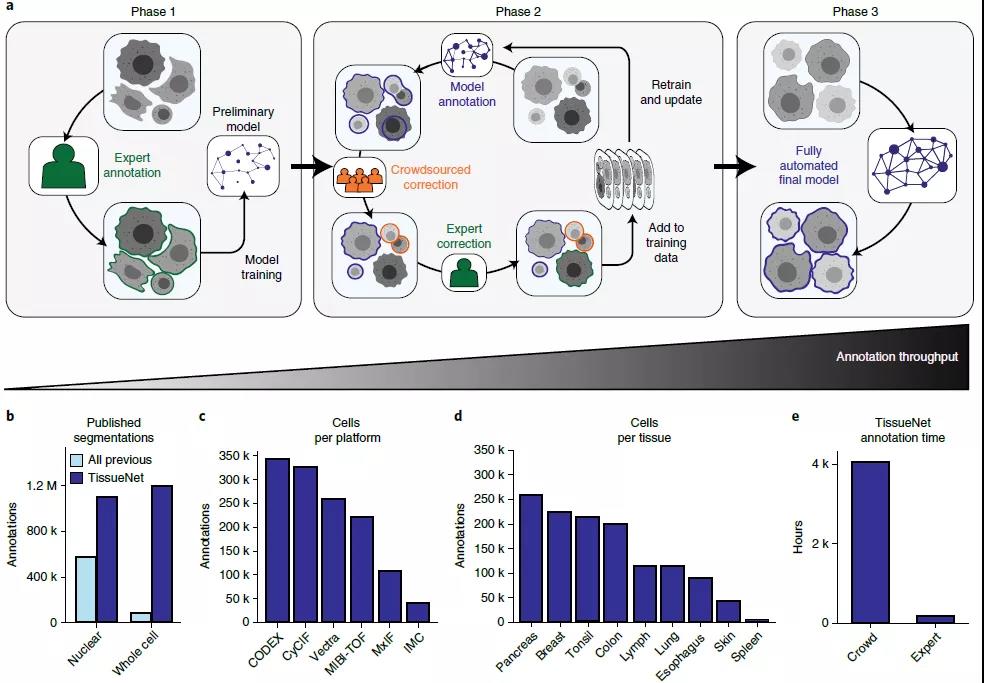 图1 | 一种“人在回路”的方法能够对大型图像集合进行可伸缩的像素级别的标注。
图1 | 一种“人在回路”的方法能够对大型图像集合进行可伸缩的像素级别的标注。
为了满足细胞分割对准确性和速度的要求,团队创建了 Mesmer,这是一种基于深度学习的组织数据核和全细胞分割算法。如图2a所示,Mesmer 的模型由一个 ResNet50 主干和一个特征金字塔网络组成,该网络有四个预测头(两个用于核分割,两个用于全细胞分割),它们与金字塔顶部相连接。Mesmer 的输入是用于定义每个细胞核的核图像和胞膜或胞质图像,这些输入被标准化,平铺成固定大小的块后,被送入深度学习模型直到模型输出产生对图像中每个细胞核和细胞的质心和边界的预测。然后将质心和边界的预测作为分水岭算法的输入,为图像中的每个细胞核和每个细胞创建最终实例分割掩码。
将其与现有的 FeatureNet 和 Cellpose进行比较,Mesmer 可以更有效地捕获图像中每个细胞的真实大小(图2b)。该团队还检查了 Mesmer 对一系列组织类型的分割预测(图2f),可以对细胞进行均一分割,不会出现过大或过小的细胞。
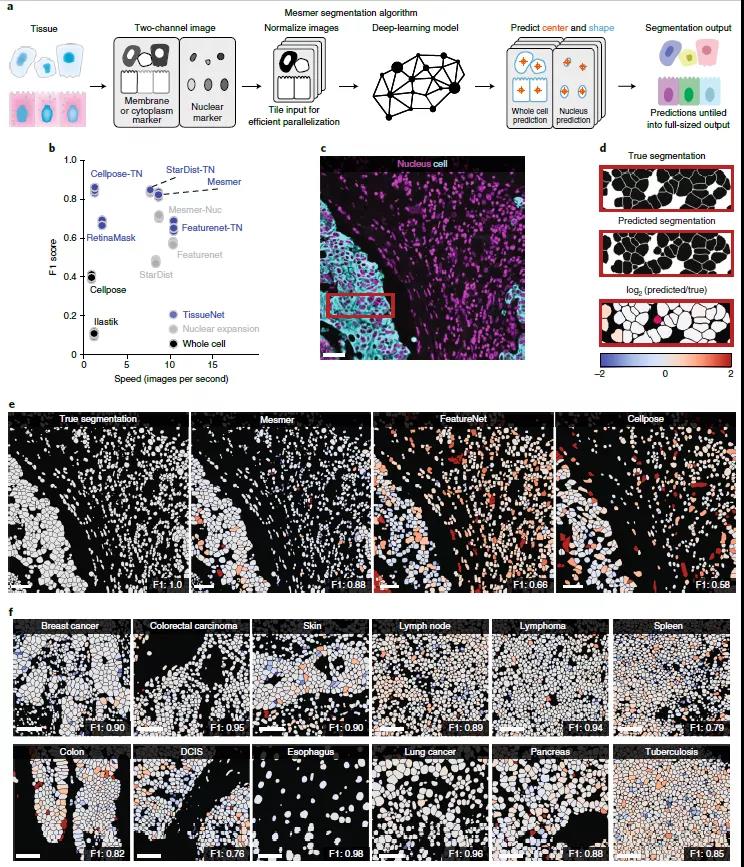 图2 | Mesmer在组织的多重图像中提供精确的细胞核和全细胞分割
图2 | Mesmer在组织的多重图像中提供精确的细胞核和全细胞分割
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢