AlphaFold2引爆科研圈的热度还未退散,又一蛋白质设计相关巨作荣登《Nature》。12月1日,蛋白质设计大师David Baker带领的团队在《Nature》上发表了《De novo protein design by deep network hallucination》一文,共同参与创作的还有华盛顿大学、伦斯勒理工学院和哈佛大学的研究人员。
 论文链接:
论文链接:
https://www.nature.com/articles/s41586-021-04184-w
David Baker之前的工作trRosetta已经可以做到根据氨基酸序列精准地预测其结构,在此基础上,如果输入到trRosetta中的序列本身是具有我们需要的蛋白特征的,那么最后折叠出来的蛋白也将具有理想特征。而现在关键的问题是:如何使序列本身具有理想特征呢?DeepDream带给了David Baker灵感,如果能够像DeepDream一样,训练过程中不断“强化”随机氨基酸序列的理想特征,那么最后就将生成具有理想特征的“想象蛋白”。
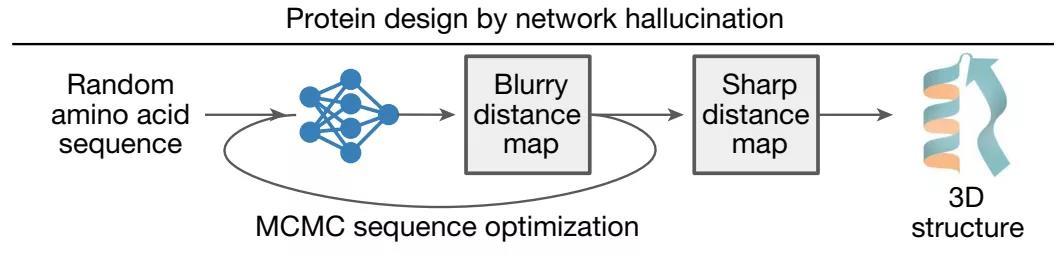
“想象蛋白”生成模型图
在大量天然蛋白质基础上进行结构预测训练之后的trRosetta,已具备能够捕获蛋白序列和结构信息的能力。因此作者选择将其作为背景网络进而“诱导”输入的随机序列生成新的蛋白质序列。通过实验发现,最初输入到网络中的随机序列生成的残基对距离分布是扩散的,难以折叠为合理的蛋白质,但随着迭代的进行,被优化的序列逐渐合理。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢