【论文标题】Pipeline for retrieval of COVID-19 immune signatures
【作者团队】Adam J H Newton, David Chartash, Steven H Kleinstein, Robert A McDougal
【发表时间】2021/12/30
【机 构】耶鲁等
【论文链接】https://doi.org/10.1101/2021.12.29.474353
【资源链接】covid-signatures.org
生物医学文献出版速度的加快使得检索论文和提取特定的综合科学信息成为一个关键的挑战。一个恰当的例子是检索报告免疫特征,即连贯的生物标志物集,的论文子集,以了解驱动不同的SARS-CoV-2感染结果的免疫反应机制。这需要一个系统的和可扩展的方法来识别和提取结构化和机器可读的COVID-19免疫特征。本文使用SPECTER嵌入,一种基于预训练的生物文本嵌入,配合SVM分类器来自动识别含有免疫特征的论文。由此而成的分类器可以检索出带有人类COVID-19免疫特征的论文,阳性预测值为86%。本文进一步对这些论文的相应作者进行半自动查询流程构建,检索特征信息,取得了31%的回应率。这证明了即使这些信息很少出现在摘要中,使用SVM分类器与摘要和标题的文档嵌入仍然可以检索具有科学意义的信息的论文。此外,基于嵌入的分类也可确定免疫特征的类型,比如基因表达与其他类型的分析。将基于文献嵌入的分类器与作者的直接参与结合起来,为建立科学相关信息的半结构化表示提供了一个很好的途径。通过这种方法,部分自动化的文献挖掘可以帮助快速创建半结构化的知识库,用于自动分析新出现的健康威胁。
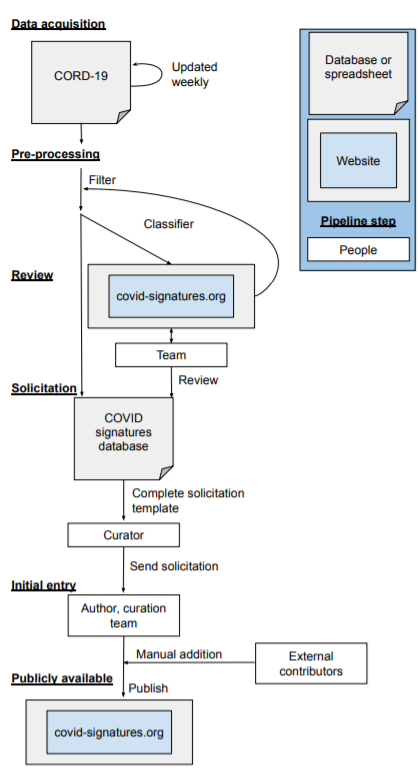
上图展示了半自动整理COVID-19免疫特征的流程。该流程的步骤与计算神经科学模型库ModelDB开发的通用网络服务流程的步骤一致。数据采集利用CORD-19,它需要预处理;包括对数据集进行实质性的过滤和删除重复的条目。筛选将文章分为3个相关或2个不相关的类别之一,然后用来建立一个分类器,为筛选提供进一步的文章,以及直接用于征集。公开的,征集后的信息归于covid-signatures.org。
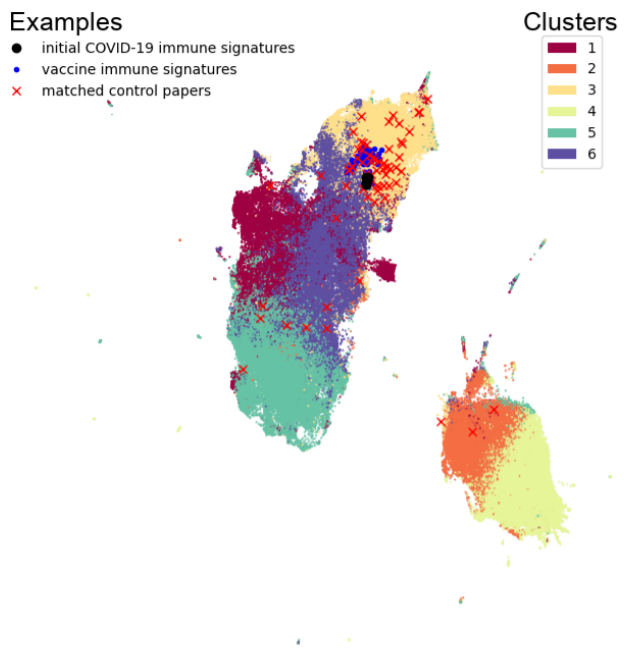
上图展示了根据其SPECTER预训练嵌入,对CORD-19数据集中的170,000以上的论文进行UMAP投影。人工选择的具有COVID-19免疫特征的论文(n = 5,大圆点)、疫苗反应免疫特征(n = 69,*标记)和匹配的对照论文(n = 68,x标记)被标记。每篇论文通过k-means聚类被分配到一个群组,根据Akaike Information Criterion判断,k=6。
几乎所有具有免疫特征的论文都被归入一个群组,即群组3。有COVID-19特征的5篇论文中的4篇和有疫苗接种特征的69篇论文中的68篇都在第3组中。其余具有免疫特征的论文属于第二个群组,即第6群组,表现为5篇具有COVID-19特征的论文中的1篇,以及69篇具有疫苗接种特征的论文中的1篇。相比之下,对照组的论文在嵌入空间中的分布更加广泛,与具有疫苗接种特征的论文相比,分布明显不同。
这种显著的聚类表明,SPECTER嵌入有效地保留了一篇论文是否含有免疫特征的信息。这些结果表明,我们可以使用SPECTER嵌入作为基础,构建一个分类器来识别具有COVID-19免疫特征的论文。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢