基于深度学习做医学图像数据分析,例如病灶检测、肿瘤或者器官分割等任务,第一步就是要对数据有一个大概的认识。但是我刚刚入门医学图像分割的时候,很迷茫不知道自己该干啥,不知道需要准备哪些知识,慢慢到现在才建立了一个简陋的知识体系。个人认为,比如说医学图像分割这个方向,再具体一点比如腹部器官分割或者肝脏肿瘤分割,需要掌握两方面的知识:(1)医学图像预处理方法;(2)深度学习知识。而第一点是进行第二点的必要条件,因为你需要了解输入到DL网络中的到底是长啥样的数据。
这篇文章主要介绍常见的医学图像读取方式和预处理方法。
1. 医学图像数据读取
1.1 ITK-SNAP软件
首先介绍一下医学图像可视化软件ITK-SNAP,可以作为直观感受医学图像3D结构的工具,也可以用来做为分割和检测框标注工具,免费,很好用,安利一下:ITK-SNAP官方下载地址。此外,mango是另一个非常轻量的可视化软件,也可以试试。我一般用ITK-SNAP。
ITK-SNAP的使用方法可以参考大佬的这篇博文,讲的很简洁:
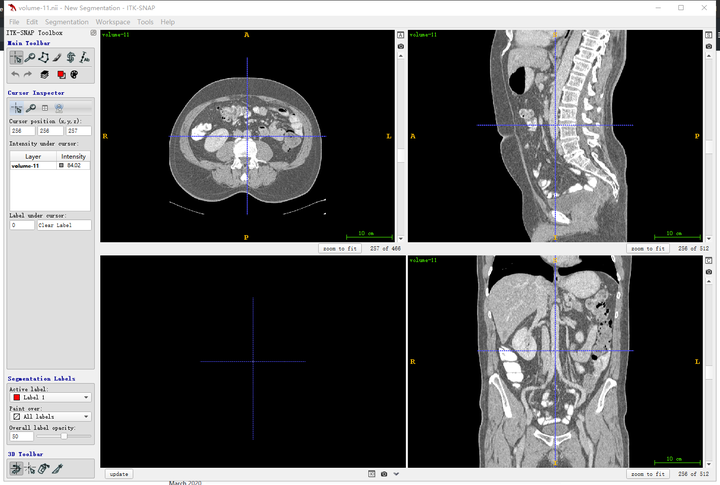
首先要明确一下和人体对应的方向,其中三个窗口对应三个切面,对应关系如下图所示,按照字母索引即可。例如,左上图对应R-A-L-P这个面,是从脚底往头部方向看的切面(即z方向),另外两张类似。

也可以同时将分割结果导入,对比观察。
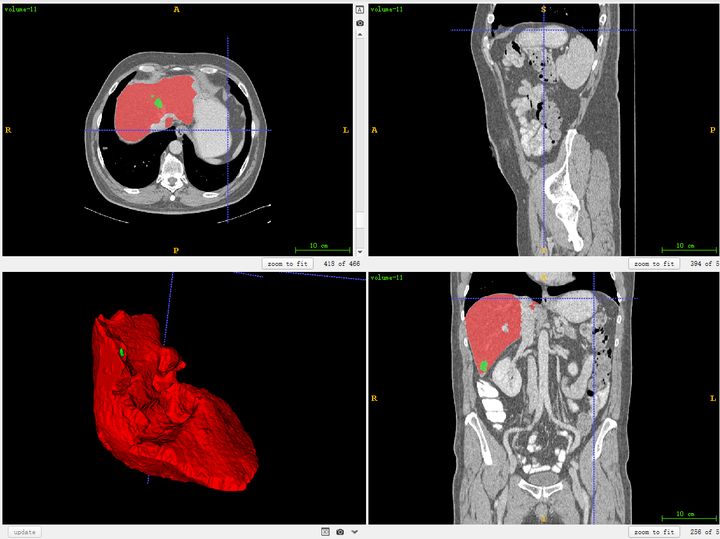
对于标注不太严谨的地方也可以精细化修改。当然公开集的话,绝大多数都挺好的。自己标注也是类似。(如果显示不太清晰,对比度太低,需要在软件中调节窗宽和窗位)
1.2 SimpleITK
我们知道,最常见的医学图像有CT和MRI,这都是三维数据,相比于二维数据要难一些。而且保存下来的数据也有很多格式,常见的有.dcm .nii(.gz) .mha .mhd(+raw)。这些类型的数据都可以用Python的SimpleITK来处理,此外pydicom可以对.dcm文件进行读取和修改。
读取操作的目的是从每一个病人数据中抽取tensor数据,用Simpleitk读取上面的.nii数据为例:
import numpy as np
import os
import glob
import SimpleITK as sitk
from scipy import ndimage
import matplotlib.pyplot as plt # 载入需要的库
# 指定数据root路径,其中data目录下是volume数据,label下是segmentation数据,都是.nii格式
data_path = r'F:\LiTS_dataset\data'
label_path = r'F:\LiTS_dataset\label'
dataname_list = os.listdir(data_path)
dataname_list.sort()
ori_data = sitk.ReadImage(os.path.join(data_path,dataname_list[3])) # 读取其中一个volume数据
data1 = sitk.GetArrayFromImage(ori_data) # 提取数据中心的array
print(dataname_list[3],data1.shape,data1[100,255,255]) #打印数据name、shape和某一个元素的值
plt.imshow(data1[100,:,:]) # 对第100张slice可视化
plt.show()输出结果:
['volume-0.nii', 'volume-1.nii', 'volume-10.nii', 'volume-11.nii',...
volume-11.nii (466, 512, 512) 232.0表明该数据shape为(466,512,512),注意对应的顺序是z,x,y。z其实是slice的索引。x和y是某一个slice的宽和高。
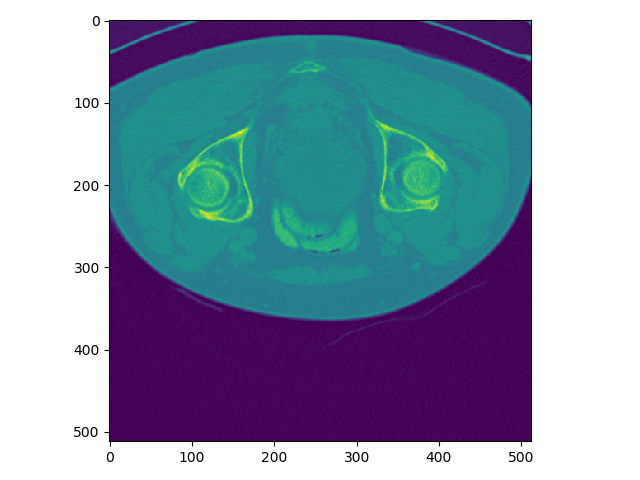
z索引为100的plot结果:
同一个slice在ITK-SNAP可视化结果(注意这里(x,y,z=(256,256,101)),因为itk-snap默认从1开始索引):
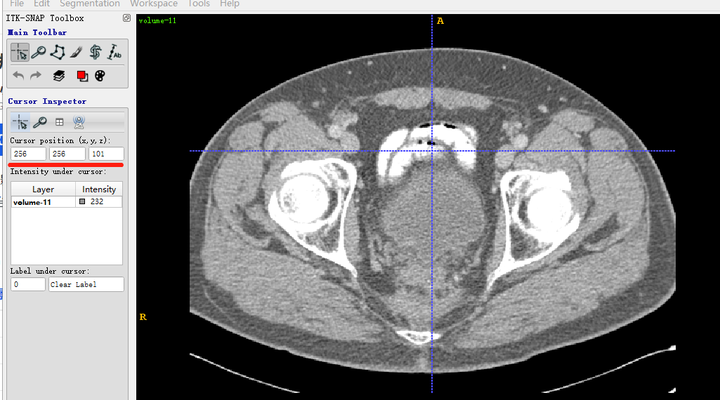
可以发现,上下两张x轴一样但y轴方向上下翻转了,这是由于matplotlib显示方式不同,但是不会出现读取数据对不齐的问题。
对于dicom和mhd的处理方式,可以参考这篇博文:
2.医学图像预处理
这部分内容比较杂乱。因为不同的任务、不同的数据集,通常数据预处理的方法有很大不同。但基本思路是要让处理后的数据更有利于网络训练。那么二维图像预处理的一些方法都是可以借鉴的,如对比度增强、去噪、裁剪等等。此外还有医学图像本身的一些先验知识也可以利用,比如CT图像中不同仿射剂量(单位:HU) 会对应人体不同的组织器官。
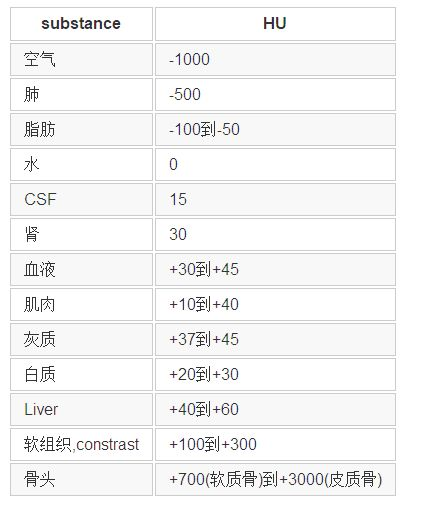
基于上表,可以对原始数据进行归一化处理:
MIN_BOUND = -1000.0
MAX_BOUND = 400.0
def norm_img(image): # 归一化像素值到(0,1)之间,且将溢出值取边界值
image = (image - MIN_BOUND) / (MAX_BOUND - MIN_BOUND)
image[image > 1] = 1.
image[image < 0] = 0.
return image也可以将其标准化/0均值化,将数据中心转移到原点处:
image = image-meam上述归一化处理适用于绝大多数数据集,其他一些都是可有可无的针对于具体数据的操作,这些操作包括上面的MIN_BOUND 和MAX_BOUND 都最好参考优秀论文的开源代码的处理方式。
预处理后的数据集建议保存在本地,这样可以减少训练时的部分资源消耗。此外,如随机裁剪、线性变换等数据增强处理步骤,还是需要在训练时进行。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢