【论文标题】Geometry-enhanced molecular representation learning for property prediction
【作者团队】Xiaomin Fang, Lihang Liu, Jieqiong Lei, Donglong He, Shanzhuo Zhang, Jingbo Zhou, Fan Wang, Hua Wu & Haifeng Wang
【发表时间】2022/02/07
【机 构】百度
【论文链接】https://www.nature.com/articles/s42256-021-00438-4
【代码链接】https://github.com/PaddlePaddle/PaddleHelix/tree/dev/apps/pretrained_compound/ChemRL/GEM
有效的分子表征学习对于促进分子特性预测具有重要意义。最近分子表征学习的进展在应用图神经网络来建立分子模型方面显示出巨大的前景。此外,最近的一些研究为分子表征设计了预训练自监督的学习方法,以解决标记分子不足的问题;然而,这些自监督的框架将分子视为拓扑图,而没有充分地利用分子几何信息。分子几何,也被称为分子的三维空间结构,对于确定分子特性至关重要。为此,我们提出了一种新的几何学增强的分子表示学习方法(GEM)。所提出的GEM有一个专门设计的基于几何的图形神经网络结构,以及几个专门的几何层面的自监督学习策略来学习分子的几何知识。我们在不同的基准上将GEM与各种最先进的基线进行了比较,结果表明它可以大大超过它们,证明了所提方法的优越性。
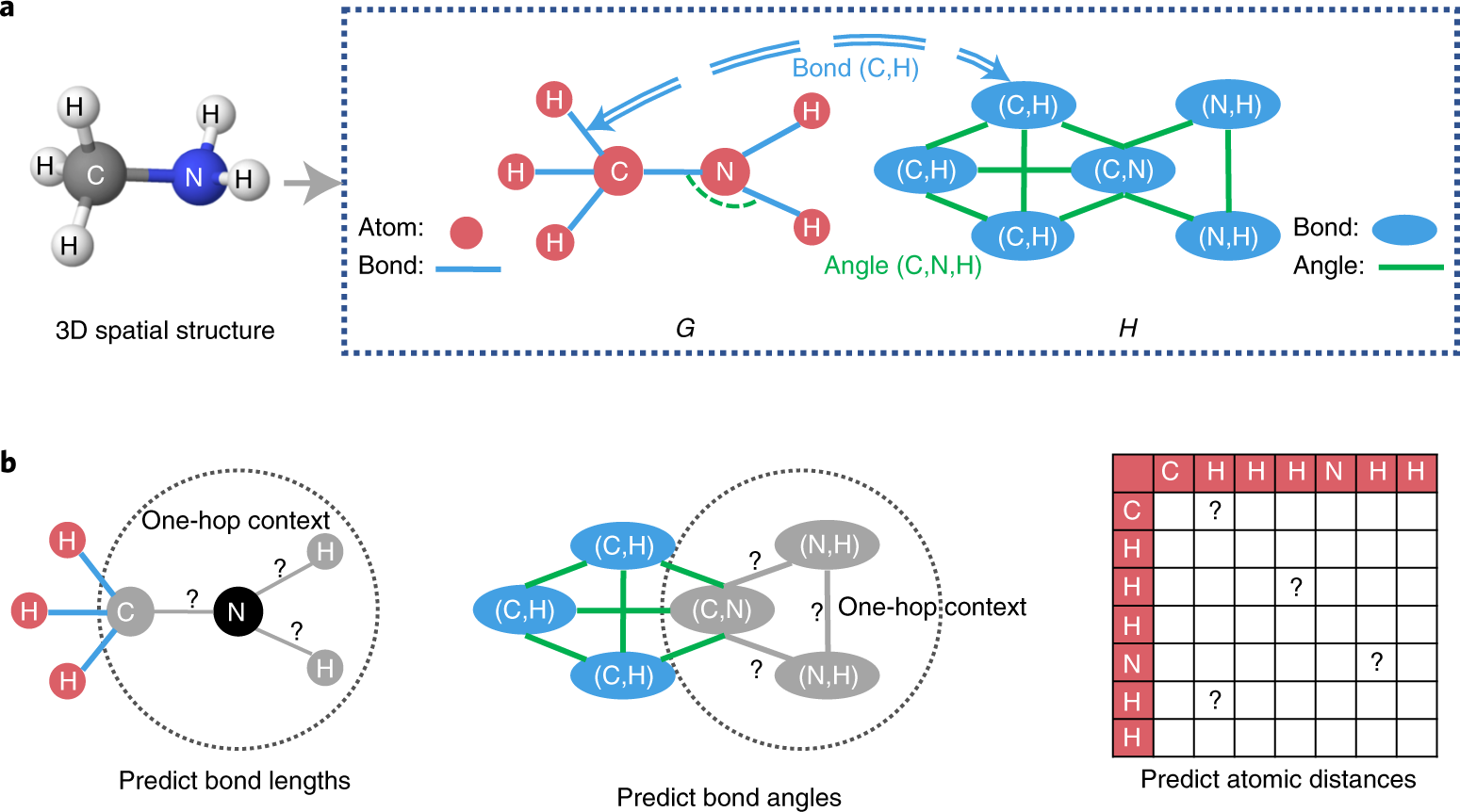
a图,在原子-键图G中,化学键被看作是边,连接原子。在键角图H中,键角被视为边,一个键角连接着两个化学键和三个原子。双线弧表示两个图中元素之间的对应关系。
b图,几何层面的自我监督学习任务演示。黑色圆圈代表被选择的原子,而图G中的灰色圆圈代表相邻的屏蔽原子,图G中的灰色线和图H中的灰色椭圆代表相邻的屏蔽键,图H中的灰色线代表相邻的屏蔽键角。
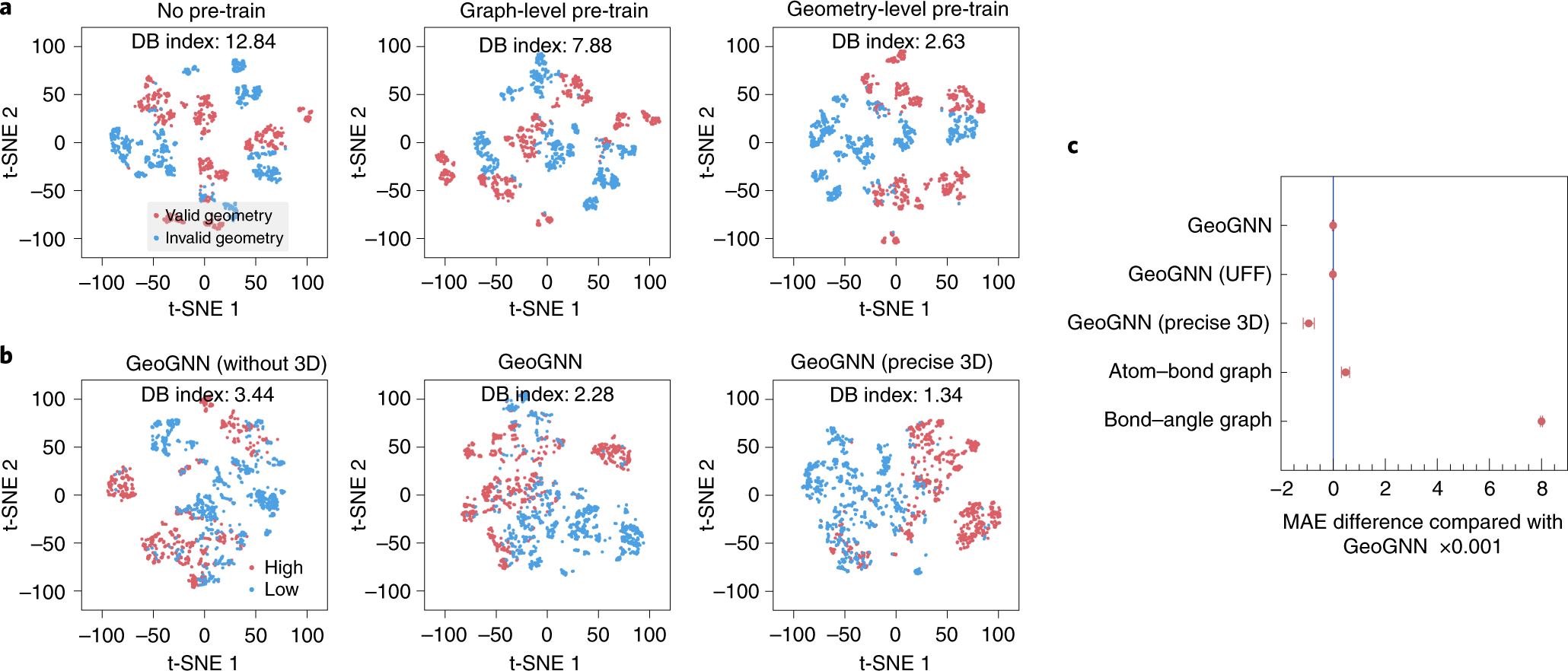
a图, 预训练的表征可视化,比较了不同的自监督方法。有效的几何图形集群包含由RDCit生成的几何图形的分子,而无效的几何图形集群包含那些随机生成的几何图形。
b图, 表征可视化,比较不同质量的几何图形。高集群包含具有高HOMO-LUMO间隙的分子,而低集群包含具有低HOMO-LUMO间隙的分子。
c图, 基线GeoGNN和其他GeoGNN变体在QM9上的MAE差异(越低越好)。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢