【论文标题】Prompt-Guided Injection of Conformation to Pre-trained Protein Model
【作者团队】Qiang Zhang, Zeyuan Wang, Yuqiang Han, Haoran Yu, Xurui Jin, Huajun Chen
【发表时间】2022/02/07
【机 构】浙大、阿里、德睿
【论文链接】https://arxiv.org/pdf/2202.02944v1.pdf
在各种生物过程中,蛋白质结构可以在几种构象之间转变,即蛋白质的折叠,而预训练的蛋白质模型(PTPMs)代表了具有一个固定嵌入的蛋白质,可能不能胜任各种任务。为了使PTPMs能够产生任务导向的表征,本文建议学习可解释的、可插拔的和可扩展的蛋白质prompt,作为向PTPMs注入任务相关知识的一种方式。先前的PTPM优化与被遮蔽的语言建模任务可以解释为学习一个序列prompt,使PTPM能够捕捉到氨基酸之间的顺序依赖。为了将构象知识纳入PTPM,本文提出了一个相互作用-构象prompt,它通过在蛋白质-蛋白质相互作用任务的反向传播学习的。作者作为一个实例提出了一个构象感知的预训练的蛋白质模型,该模型在多任务环境下学习序列和相互作用形态prompt,并在九个蛋白质数据集上进行了综合实验,结果证实了作者的预期,即使用序列prompt不会损害PTPMs在序列相关任务上的表现,而纳入相互作用形态prompt则显著提高PTPMs在构象知识方面的表现。本文还表明,学到的prompt可以被组合和扩展以处理新的复杂任务。
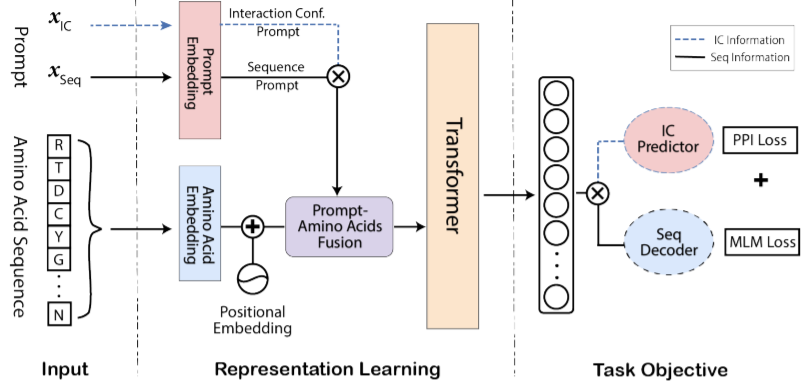
上图显示了模型的架构,prompt即一个符号化的模式串,可以手动设计或自动学习,将与任务相关的知识注入到预训练好的模型中,从而产生信息性的表征。作者通过ConfProtein来实例化所提出的prompt学习方法。本文使用之前的预训练MLM任务,即根据上下文恢复被替换的氨基酸,来优化PTPM和Seq prompt,所得到的表征应该捕捉到氨基酸之间的化学特性,并有助于预测蛋白质的二级结构。此外,另一个目标是通过学习相互作用-形态prompt将蛋白质构象知识注入PTPM中,为此进行了新的任务--预测第p个和第q个蛋白质是否能相互作用。
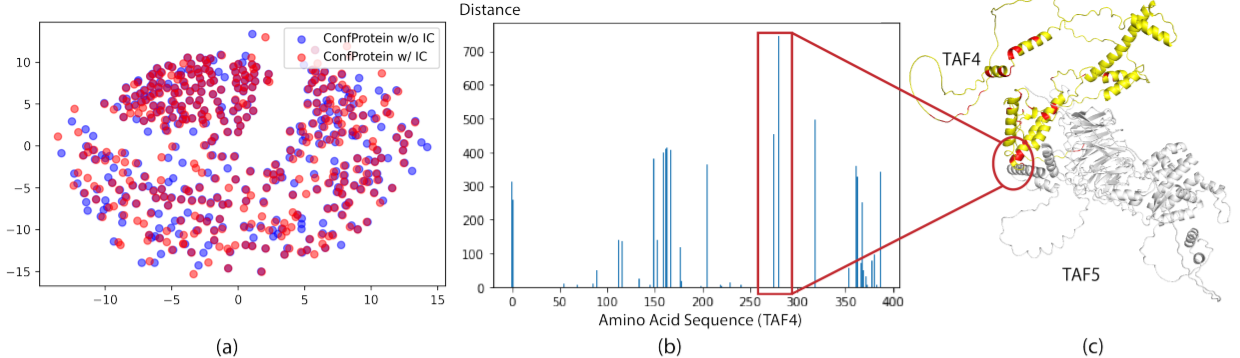 上图显示了两个蛋白质是转录起始因子TFIID亚单位4(TAF4)和转录起始因子TFIID亚单位5(TAF5)交互构象信息的可视化。左图:通过MDS可视化有构象信息和无构象信息的氨基酸(TAF4)的嵌入。中间。可视化有构象信息和无构象信息的相应氨基酸的距离。右图。可视化距离大于100的氨基酸(红色)。可以发现被标记的嵌入物都是蛋白质表面的氨基酸,这与与PPI有关的氨基酸几乎都位于蛋白质的表面,而不是核心部位的事实相一致。
上图显示了两个蛋白质是转录起始因子TFIID亚单位4(TAF4)和转录起始因子TFIID亚单位5(TAF5)交互构象信息的可视化。左图:通过MDS可视化有构象信息和无构象信息的氨基酸(TAF4)的嵌入。中间。可视化有构象信息和无构象信息的相应氨基酸的距离。右图。可视化距离大于100的氨基酸(红色)。可以发现被标记的嵌入物都是蛋白质表面的氨基酸,这与与PPI有关的氨基酸几乎都位于蛋白质的表面,而不是核心部位的事实相一致。
总结:
- 本文学习可插拔、可解释和可扩展的prompt,将任务相关的知识注入预训练的蛋白质模型中。
- 作为一个实例,本文设计了ConfProtein模型,在多任务环境下,将顺序和构象知识注入预训练的蛋白质模型。
- 本文创建了一个新的数据集,其中包含用于接触预测的交互构象信息。
- 对蛋白质功能和结构预测任务的综合评估表明,适当的prompt能显著提高预训练模型的性能。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢