【论文标题】A deep-learning system bridging molecule structure and biomedical text with comprehension comparable to human professionals
【作者团队】Zheni Zeng, Yuan Yao, Zhiyuan Liu & Maosong Sun
【发表时间】2022/02/14
【机 构】清华
【论文链接】https://www.nature.com/articles/s41467-022-28494-3
【代码链接】https://github.com/thunlp/KV-PLM
为了加速生物医学研究进程,研究者开始通过阅读大规模的生物医学数据来自动获取分子实体的知识。受人类从分子结构和生物医学文本信息的多功能阅读中学习深度分子知识的启发,本文提出了一个知识型机器阅读系统,在一个统一的深度学习和预训练框架中衔接这两类信息,为生物医学研究提供全面帮助。本文解决了现有的机器阅读模型只能分别处理不同类型的数据的问题,从而实现对分子实体的全面彻底的理解。通过在不同信息源内和不同信息源之间以预训练无监督的方式掌握元知识,本文的系统可以促进各种现实世界的生物医学应用,包括分子特性预测、生物医学关系提取等。实验结果表明,本文的系统在分子特性的理解能力上甚至超过了人类专业人士,同时也揭示了其在促进未来自动药物发现和记录方面的巨大潜力。
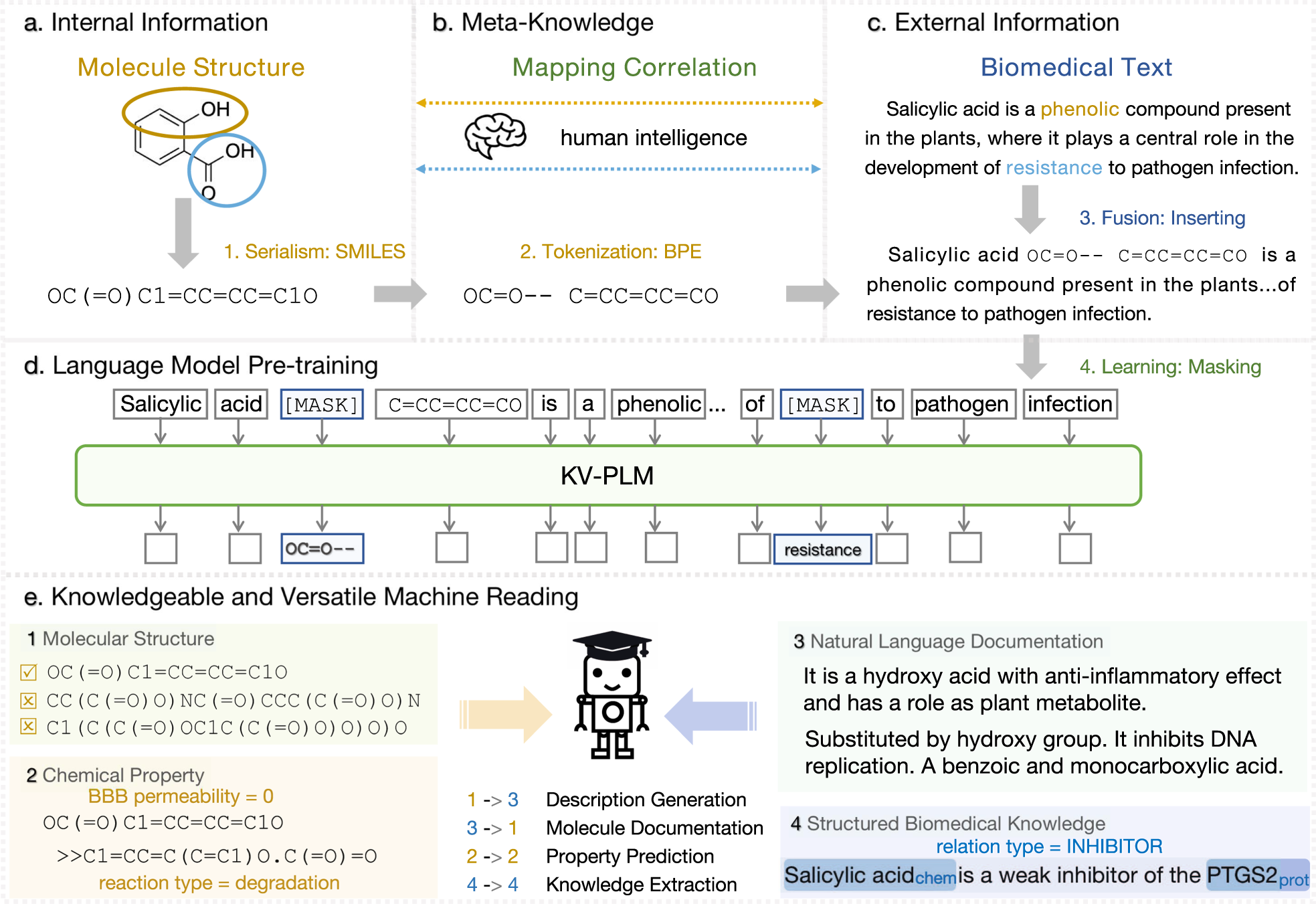
这里本文以水杨酸为例。受人类在不同信息内和不同信息间通用学习元知识的启发,本文的机器阅读系统首先通过BPE对SMILES字符串进行序列化,然后将子字符串插入大规模语料库,并通过掩码语言建模学习不同语义单元间的细粒度映射。通过这种方式,该系统可以进行知识性和多功能性阅读,在单信息下游任务和多功能性阅读任务上都取得了良好的表现。

对于给定的陌生分子实体,我们得到一个包括结构和描述的多功能材料,从中知道正确的句子,并从池中随机挑选错误的句子,形成四个选择。 分子结构和选择的文本被送入KV-PLM,并得到它们的表示,在此基础上,通过余弦相似度计算选择的置信度分数,结构和生物医学文本的标记器不同。在这个例子中,KV-PLM成功地找出了给定物质的正确描述句。

上图显示了由KV-PLM预测的Tuberin的属性描述。分子结构首先被序列化为SMILES字符串。随着提供更多的SMILES基团(紫),模型可以更精确地预测属性。对于描述检索,系统会找到适当的描述性句子,并为给定的SMILE字符串生成一段自然语言描述。句子和物质都是从PCdes测试集中随机选择的。醚键一开始被预测为醇,在得到输入的苯环模式后,成功地被识别为芳香族醚。该模型甚至预测,主要由于双键,Tuberin具有抗氧化剂的作用,这在PubChem中没有记录。结晶也是一个正确预测的属性。
综上,
1.本文提出了一个知识渊博、用途广泛的机器阅读系统,将分子结构和生物医学文本连接起来。
2.本文的主要贡献在于将所提出的模型应用于协助药物发现和生物医学研究的文件。
3.综合实验表明了所提模型的有效性。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢