AlphaFold 虽好,但耗时且成本高,现在首个用于蛋白质结构预测模型的性能优化方案来了。

论文:https://arxiv.org/pdf/2203.00854.pdf
代码:http://github.com/hpcaitech/FastFold
实验结果表明,FastFold 将整体训练时间从 11 天减少到 67 小时,并实现了 7.5 ∼ 9.5 倍的长序列推理加速。此外,研究者还将 FastFold 扩展到 512 个 A100 GPU 的超算集群上,聚合峰值性能达到了 6.02PetaFLOPs,扩展效率达到 90.1%。
不同于一般的 Transformer 模型,AlphaFold 在 GPU 平台上的计算效率较低,主要面临两个挑战:1) 有限的全局批大小限制了使用数据并行性将训练扩展到更多节点,更大的批大小会导致准确率更低。即使使用 128 个谷歌 TPUv3 训练 AlphaFold 也需要约 11 天; 2) 巨大的内存消耗超出了当前 GPU 的处理能力。在推理过程中,较长的序列对 GPU 内存的需求要大得多,对于 AlphaFold 模型,一个长序列的推理时间甚至可以达到几个小时。
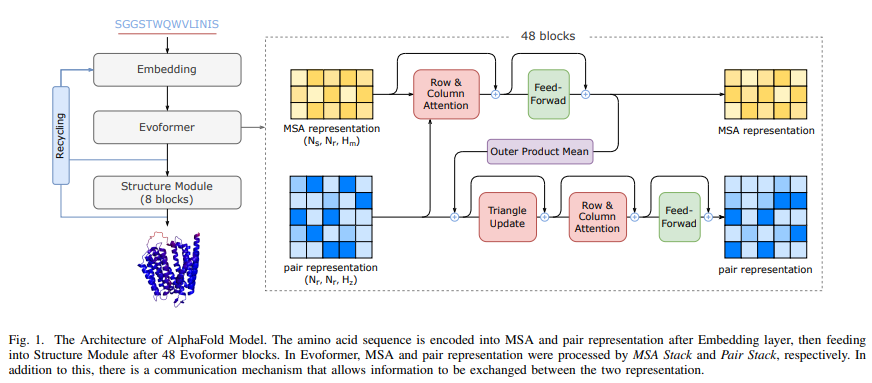
作为首个用于蛋白质结构预测模型训练和推理的性能优化工作,FastFold 成功引入了大型模型训练技术,显著降低了 AlphaFold 模型训练和推理的时间和经济成本。

本文的作者之一尤洋现为新加坡国立大学计算机系任助理教授。2020 年,尤洋在加州大学伯克利分校计算机系获得博士学位。
尤洋的主要研究方向是高性能计算与机器学习的交叉领域,当前研究重点为大规模深度学习训练算法的分布式优化。他曾其以一作作者的身份发表研究论文《Large Batch Optimization for Deep Learning :Training BERT in 76 Minutes》,提出了一种 LAMB 优化器(Layer-wise Adaptive Moments optimizer for Batch training),将超大模型 BERT 的预训练时间由 3 天缩短到了 76 分钟,刷新世界记录。到目前为止,LAMB 仍为机器学习领域的主流优化器,成果被 Google、Facebook、腾讯等科技巨头在实际中使用。
2021 年 7 月,尤洋在北京中关村创办了高性能计算公司「潞晨科技」。不久后,潞晨科技即宣布完成超千万元种子轮融资,该公司创业目标是「最大化人工智能开发速度的同时,最小化人工智能模型部署成本」。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢