【论文标题】Drug design and repurposing with a sequence-to-drug paradigm
【作者团队】 Lifan Chen Chen, Zisheng Fan, Jie Chang, Ruirui Yang, Hao Guo, Yinghui Zhang, Tianbiao Yang, Chenmao Zhou, Zhengyang Chen, Chen Zheng, Xinyue Hao, Keke Zhang, Rongrong Cui, Yiluan Ding, Naixia Zhang, Xiaomin Luo, Hualiang Jiang, Sulin Zhang, Mingyue Zheng
【发表时间】2022/03/26
【机 构】中科院、中科大等
【论文链接】https://doi.org/10.1101/2022.03.26.485909
【代码链接】https://github.com/lifanchensimm/transformerCPI
近几十年来,基于靶点蛋白的药物开发一直是一种成功的方法。传统的基于结构的药物设计管道是一个复杂的、由人设计的管道,有多个独立的优化步骤。端到端差异化学习的进展表明,类似的重新制定药物设计的潜在好处。本文提出了一个新的序列到药物的范式,直接从蛋白质序列中发现类似药物的小分子调节剂,并首次在三个阶段验证了这个概念。首先,本文设计了基于预训练与蛋白序列和分子图的TransformerCPI2.0作为序列到药物范式的核心工具,它表现出与传统的基于结构的药物设计方法相竞争的性能。第二,本文验证了TransformerCPI2.0学到的结合知识。第三,本文应用序列到药物的范式发现了E3泛素蛋白连接酶的新hits:斑点型POZ蛋白(SPOP)和没有三维结构的RNF130。本文表明,序列到药物的模式是一个很有前途的药物开发方向。
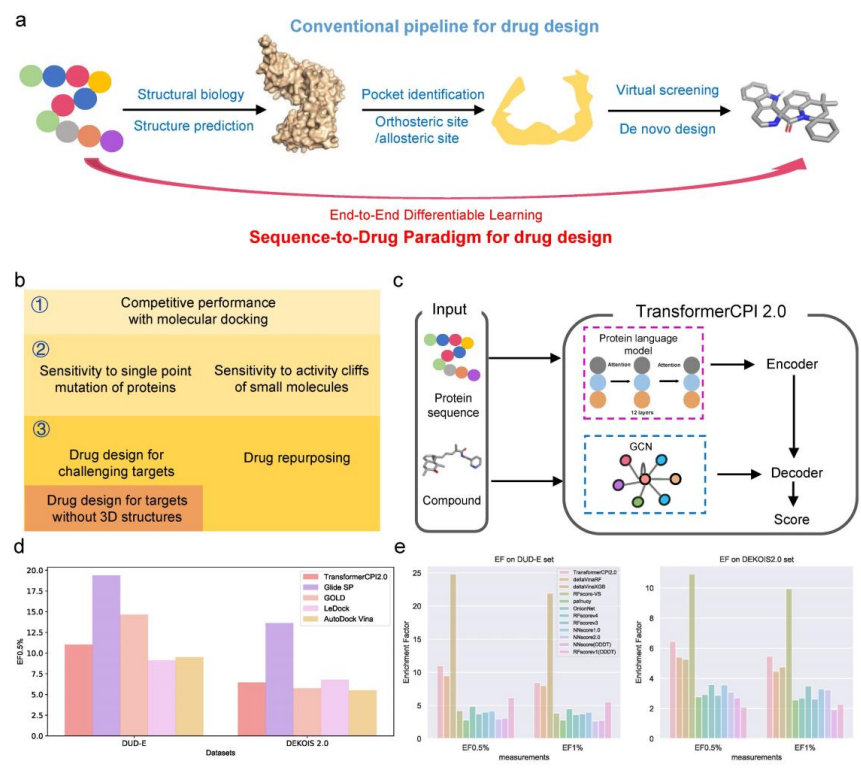
A图展示了传统的基于靶点的药物设计流程和从序列到药物的范式。基于蛋白质结构的药物设计(SBDD)从蛋白质序列开始,通过结构生物学或结构预测建立一个三维结构,确定结合口袋 ,并最终通过虚拟筛选或从头设计发现活性调节剂
B图展示了从序列到药物范式的概念验证的三个阶段,每个阶段用不同的颜色标示。
C图展示了TransformerCPI2.0的计算流程。该模型通过预训练的蛋白质语言模型TAPE-BERT计算蛋白质序列表征,用基于自注意的Transformer编码器并在原子序列中引入基于GNN的原子向量,携带分子水平的交互信息。
D图展示了TransformerCPI2.0和docking方法在DUD-E和DEKOIS 2.0 2个docking数据集上的EF(enrichment factor)结果。
E图展示了TransformerCPI2.0和机器学习打分函数在DUD-E集和DEKOIS 2.0集上的EF0.5%和EF1%的结果。
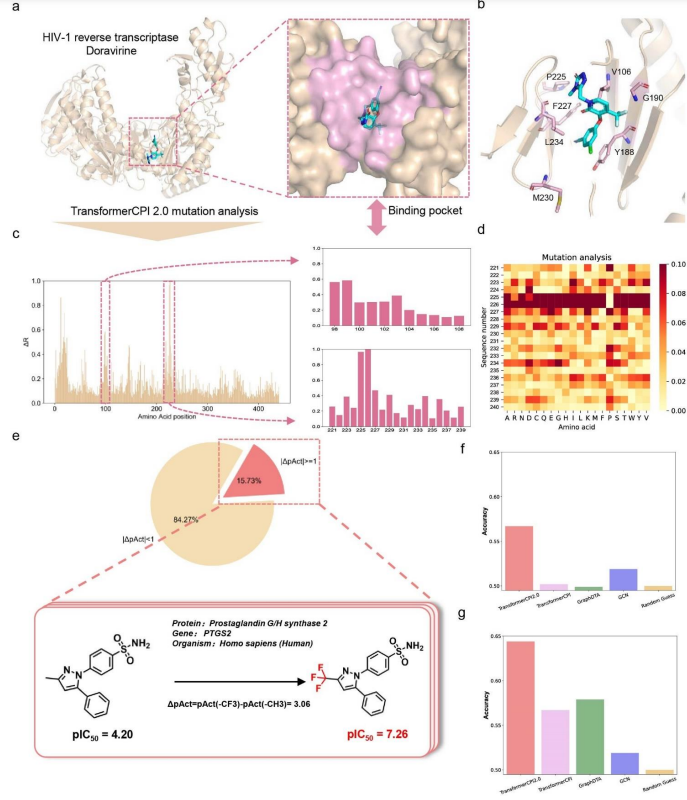
a, HIV-1逆转录酶和多拉韦林的共晶体结构(PDB: 4NCG)。
b, 多拉韦林的结合模式。
c, TransformerCPI2.0计算的每个氨基酸位置的相对活性变化分数(∆𝑅)。
d, 热力图绘制了221到240位的活性变化得分,其中每个位置都突变为20个氨基酸(包括其本身)。颜色越深代表突变引起的活性变化得分越高。
e, 左三氟甲基替代数据集的数据分布。只有15.73%的-CH3被-CF3取代后可以使生物活性增加或减少至少一个数量级。本文对这部分数据进行了取代效应分析。
f, TransformerCPI2.0和基线模型在整个数据集上的准确性。
g, TransformerCPI2.0和基线模型在-CH3被-CF3替代后,生物活性至少增加或减少三个数量级的子集上的准确性。
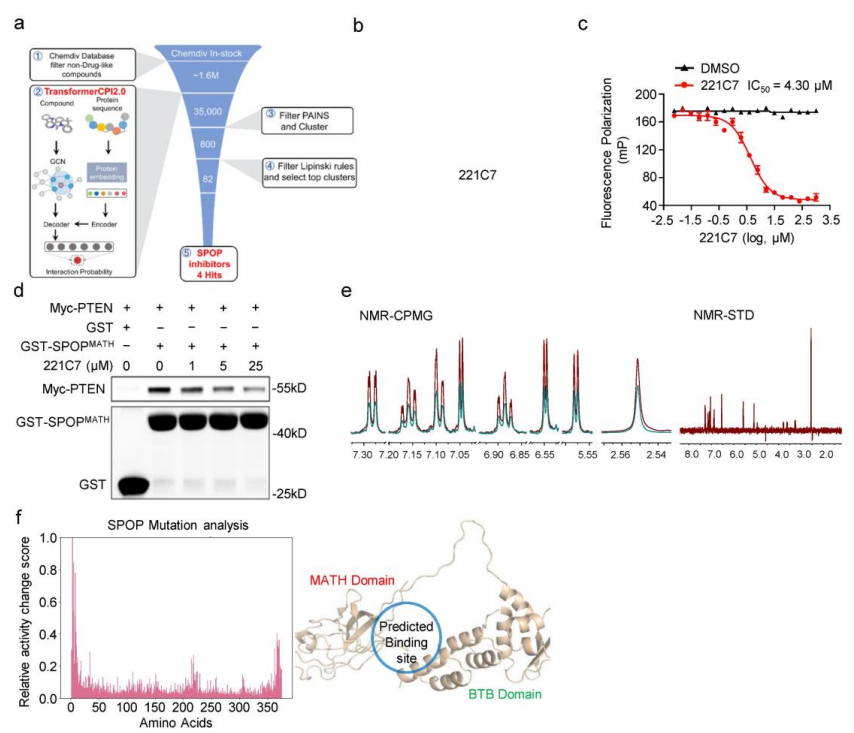
上图展示了发现新的SPOP的骨架hits的流程。SPOP的小分子抑制剂的虚拟筛选方案如a图。
首先,从ChemDiv库中筛选出非药物类化合物,其中包含约160万个库存化合物,然后应用TransformerCPI2.0对这些化合物进行评分,通过筛选选出前35000个分子(~前2%,确保化合物多样性)。第二,本文过滤了panassay干扰化合物(PAINS),并根据这些分子的扩展连接性指纹(ECFP)自动进行聚类,得到了大约800个聚类。第三,本文通过Lipinski规则过滤这些化合物,并从每个聚类中手动选择有代表性的化合物。最后,总共选择了82个候选化合物,其中4个经过荧光偏振(FP)测定为hits,221C7 IC50为4.30微摩,具体如图所示
c, 221C7竞争性地抑制puc_SBC1肽与SPOPMATH的结合,这是通过FP测定的。
d, 221C7破坏了SPOPMATH和PTEN之间的蛋白质结合,通过体外下拉试验进行测量。
e,核磁共振测量221C7和SPOPMATH之间的直接结合。221C7的CPMG核磁共振谱(红色),221C7在5μM SPOPMATH存在下的谱(绿色)。221C7的STD光谱是在5μM SPOPMATH存在下记录的。
f,耐药性突变分析解释了TransformerCPI2.0的预测,重要的残基被映射回全长SPOP的结构(PDB:3HQI)。预测的结合位点位于MATH结构域上和BTB结构域附近。
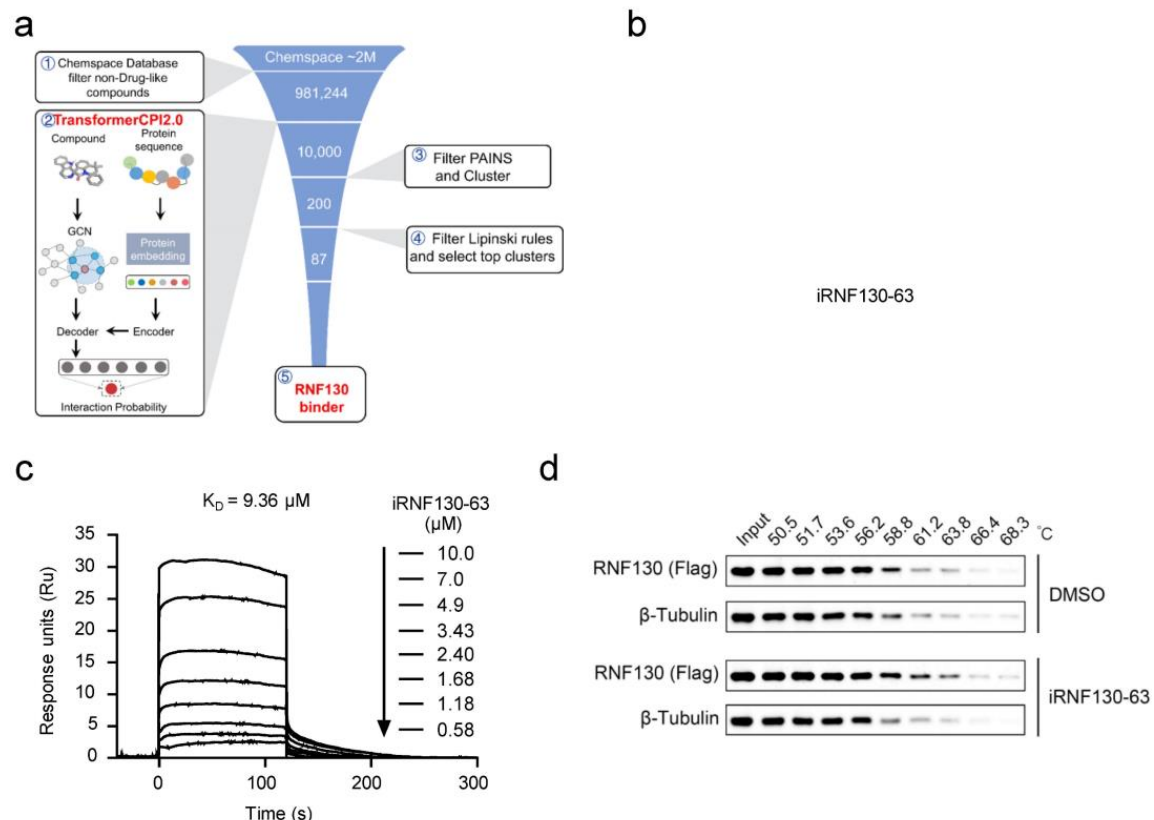
另一个案例为RNF130的虚拟筛选。首先,从Chemspace Library 中过滤出非药物类化合物,其中包含大约200万个库存化合物,然后应用TransformerCPI2.0对这些化合物进行评分,选出前10000个分子(~前0.5%,确保化合物多样性)。第二,本文过滤了panassay干扰化合物(PAINS),并根据这些分子的扩展连接性指纹(ECFP)自动进行聚类,得到了大约200个聚类。第三,本文通过Lipinski规则过滤这些化合物,并从每个聚类中手动选择有代表性的化合物。最后,总共选择了87个候选化合物,其中iRNF130-63经过SPR测定kd值为9.63微摩,并经过细胞热位移测定具备热稳定性。
总体来说,本文的方法不依赖于蛋白质的三维结构,却能达到与基于三维结构的分子对接相媲美的性能,可用于解决虚拟筛选和目标识别任务。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢