【论文标题】Accelerating Bayesian Optimization for Biological Sequence Design with Denoising Autoencoders
【作者团队】Samuel Stanton, Wesley Maddox, Nate Gruver, Phillip Maffettone, Emily Delaney, Peyton Greenside, Andrew Gordon Wilson
【发表时间】2022/03/23
【机 构】NYU
【论文链接】https://arxiv.org/abs/2203.12742v1
【代码链接】https://github.com/ samuelstanton/lambo
贝叶斯优化是查询有效的连续优化的一个黄金标准。本文开发了一种新的方法LaMBO,它将去噪自编码器与多任务高斯过程联合训练,使自编码器的隐空间中的多目标函数基于梯度优化,这使LaMBO能够在多轮设计中平衡探索-开发的权衡,并通过在帕累托边界的许多不同点上优化序列。本文在基于ZINC数据集的小分子任务和一个针对荧光蛋白的大分子任务上评估了LaMBO,在比较中该方法优于各种基线优化方法,这表明贝叶斯优化对于生物序列设计是实用和有效的。值得一提的是本文讨论了基于无监督预训练的优化方法,研究大型预训练编码器与序列模型技术相结合进行蛋白质设计将是值得期待的。
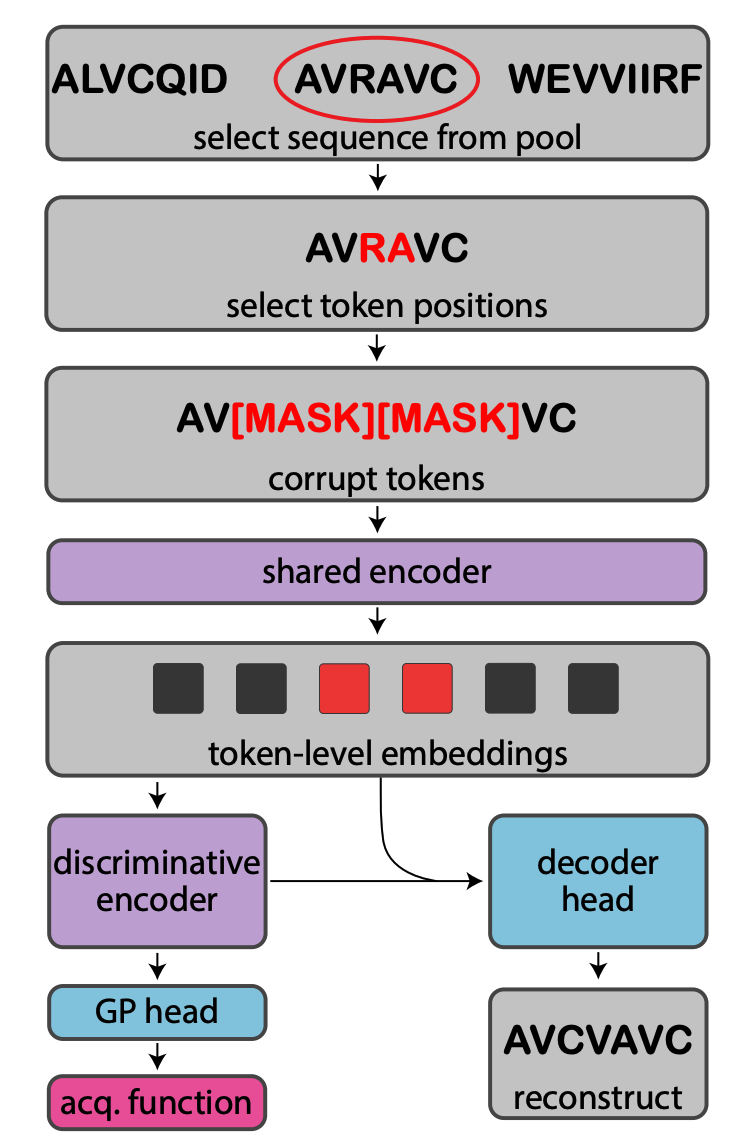
本文使用非自回归去噪自动编码器将离散序列映射到连续标记级表征的序列中,并从该序列中映射出一个多任务GP头,对集合的序列级表征进行操作。一个共享的、双向的编码器从遮蔽输入序列中产生连续的标记级嵌入Z,这些嵌入被传递给一个鉴别器以产生特定任务的标记级嵌入Z1,生成式解码器头接收Z和Z1作为输入,鉴别式GP头汇集Z1以预测目标值。GP头允许使用原则性的获取函数来管理探索-开发的权衡,而解码器头允许LaMBO在选择新的查询时遵循潜在空间中的获取梯度。
与以往将GP与深度生成模型相结合的工作不同,本文的架构不需要预训练,也不需要在接收新数据后完全重新初始化代理。LaMBO与LatentSpace BayesOpt(LSBO)最为相似,因为这两种方法都是在生成模型解码的隐表征之上训练GP头,并利用隐表征进行内循环优化。LSBO使用大型预训练数据集和VAE来解决单目标小分子设计任务,分别训练VAE和GP头。除了LaMBO使用DAE而不是VAE之外,本文在多个方面对LSBO进行了实质性的改进,例如能够使用通用的序列模型架构,取消了预训练的要求,为用共享编码器联合训练生成式和GP头提供了一个可靠的架构,并引入了多目标函数功能。
此外,本文证明随机突变和精确推理都可以使用,缓解了对计算可扩展性或对非高斯目标的适用性的担忧。
创新点
1. 本文提出LaMBO架构,通过深度核学习与非自回归去噪自编码器(DAEs)的结合,解决了蛋白质设计的高维、多目标、数据稀缺的问题,用于有监督的GP推理。
2. LaMBO内循环优化程序,包括一个可靠的初始化程序,解决了多目标获取函数的缺陷。
3. 引入了一个新的、动机良好的多目标基准任务,使fpBase中的红色荧光蛋白的SASA和稳定性最大化。
4. 在一个常用的小分子设计任务以及本文新提出的任务上取得了有力的结果,并与遗传和潜在的优化基线进行了比较。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢