【论文标题】Few Shot Protein Generation
【作者团队】Soumya Ram, Tristan Bepler
【发表时间】2022/04/03
【机 构】MIT
【论文链接】https://arxiv.org/pdf/2204.01168v1.pdf
本文提出了MSA2Prot Transformer,一个蛋白质序列的生成模型,其条件是由多序列比对(MSA)代表的蛋白质家族。与现有的学习蛋白质家族生成模型的方法不同,MSA2Prot Transformer将序列生成的条件直接建立在对多序列比对的学习编码上,避免了对特定家族模型的拟合。通过对Pfam中大量整理的MSA进行训练,我们的MSA2Prot Transformer对未经训练的蛋白质家族有很好的概括性,并且优于传统的家族建模方法,特别是当MSA较小时。我们的生成方法准确地模拟了上位性和插入删除,并允许精确推断和高效采样。本文在案例中展示了MSA-蛋白质Transformer的蛋白质序列建模能力,并在综合基准实验中与其他序列建模方法进行比较。
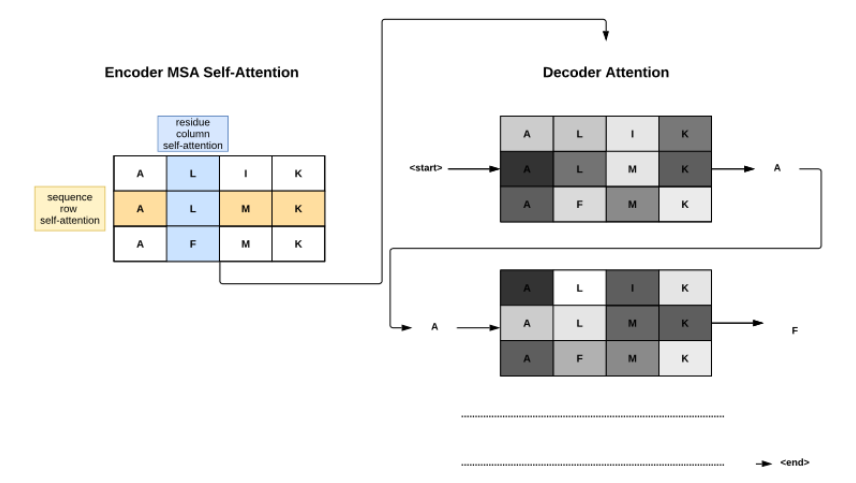
上图展示了 MSA2Prot架构。该模型由Encoder和Decoder两部分构成,Encoder部分类似MSA transformer,由行和列分别组成的轴向多头自注意力与全连接层编码;Decoder层包括3部分,因果自注意力,交叉注意力MSA表征和全连接层。其中的因果自注意力掩码旨在使模型只可见单向的序列和MSA表征,交叉自注意力MSA则旨在加速模型,获得沿着行和列的维度扁平化的MSA表征,最终在解码器完成基于目标序列ak对k+1位置token的解码。
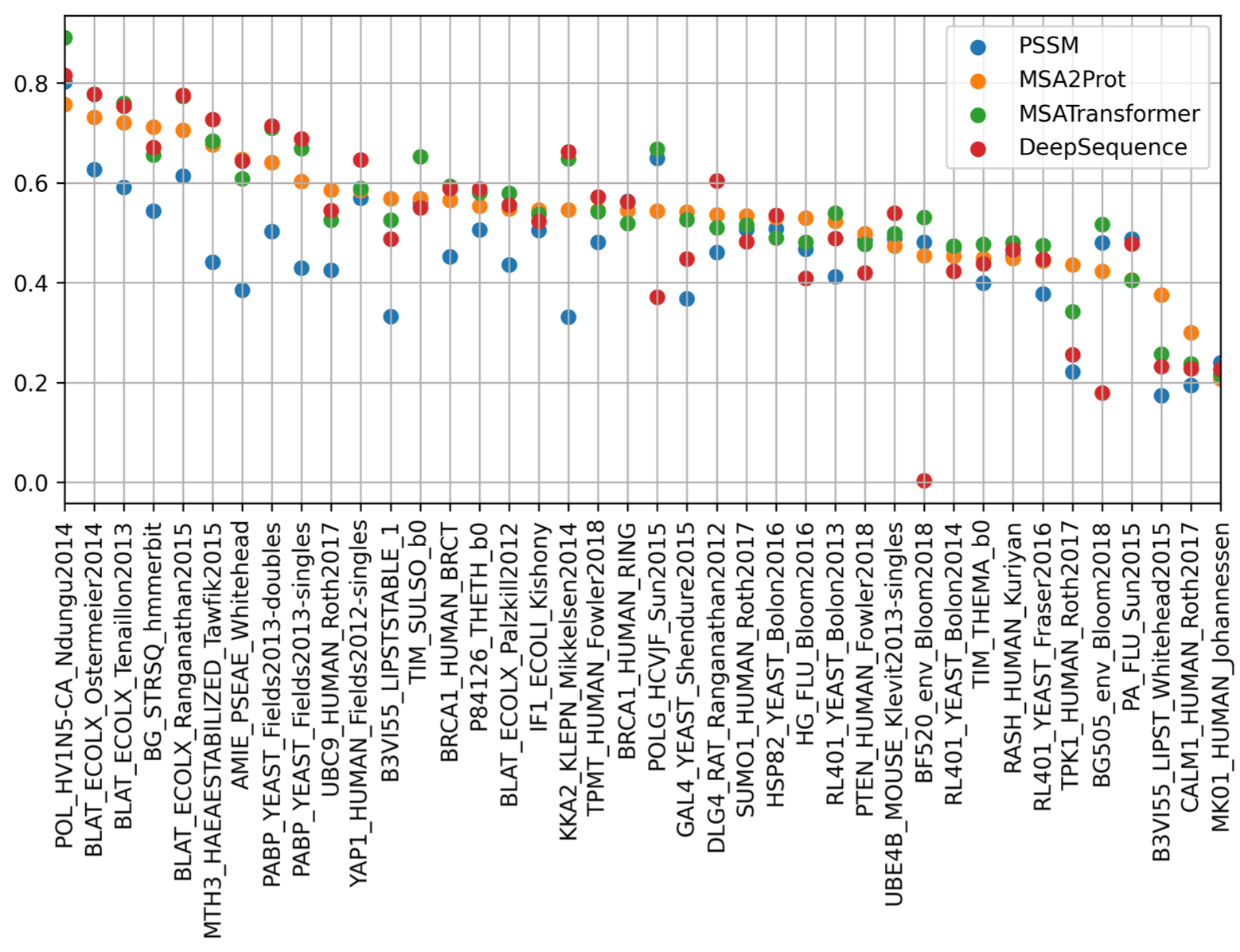
上图展示了MSA2Prot在Deepsequence上的单突/双突DMS数据的表现,可以看到MSA2Prot效果领先。指的注意的是,对于多样化的突变数据集,比如chorismate突变酶数据集,MSA-transformer的表现只有相关系数0.12,远远低于NSA2Prot或者其他对比方法,这可能是因为MSATransformer使用加法评分来评估高阶突变体的可能性,而MSA2Prot的解码器能够准确模拟上位性。
另一方面,本文在Courtney et al提供的262个删除和4422个插入数据上进行了评价,尽管MSA2Prot不需要训练,但它依然与HMM,Deepsequence等方法取得了类似的效果。
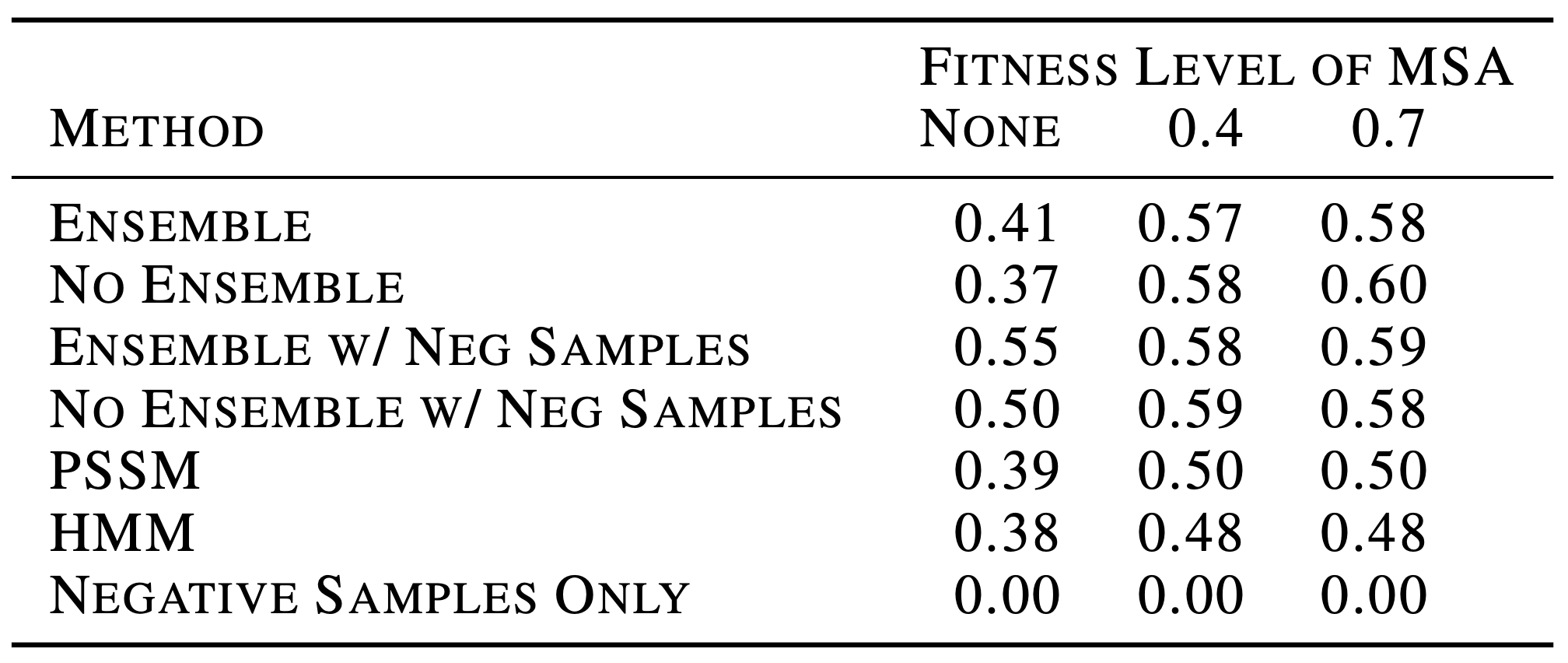
本文对MSA2Prot对于功能蛋白的泛化进行了评价。该实验是基于如下假设,蛋白空间极其庞大,而其中大部分为非功能突变体,因此只关注功能蛋白质突变体空间对有效可行地搜索序列空间至关重要。基于此,本文发现MSA2Prot能够从高表现的突变体的分布中进行归纳。
具体而言,在训练MSA被不同fitness阈值0.4,0.7过滤到只包括几百个高表现序列后,测试集上的Spearman相关系数获得大幅度改善。另外本文发现MSA2Prot能够利用低表现的变体来显著提高准确性。尽管这些低性能变体本身没有预测价值,但作者通过从标准MSA的序列的可能性中减去低性能MSA的序列的可能性,将它们作为负样本使用。
这契合了通常情况,对一个给定的蛋白质的文献整理往往会得到不同的高表现和低表现的突变体。然而,鉴于实验设置的不同,可能没有一致的fitness度量。MSA2Prot是这种情况的理想候选者,因为它有能力利用高表现和低表现的突变体,而无需明确的功能测量。
另外,MSA2Prot还可以进行条件生成。给定一个蛋白质序列,下一个残基的概率分布可以通过增加和重新归一化两个不同属性的MSA的边际来获得。
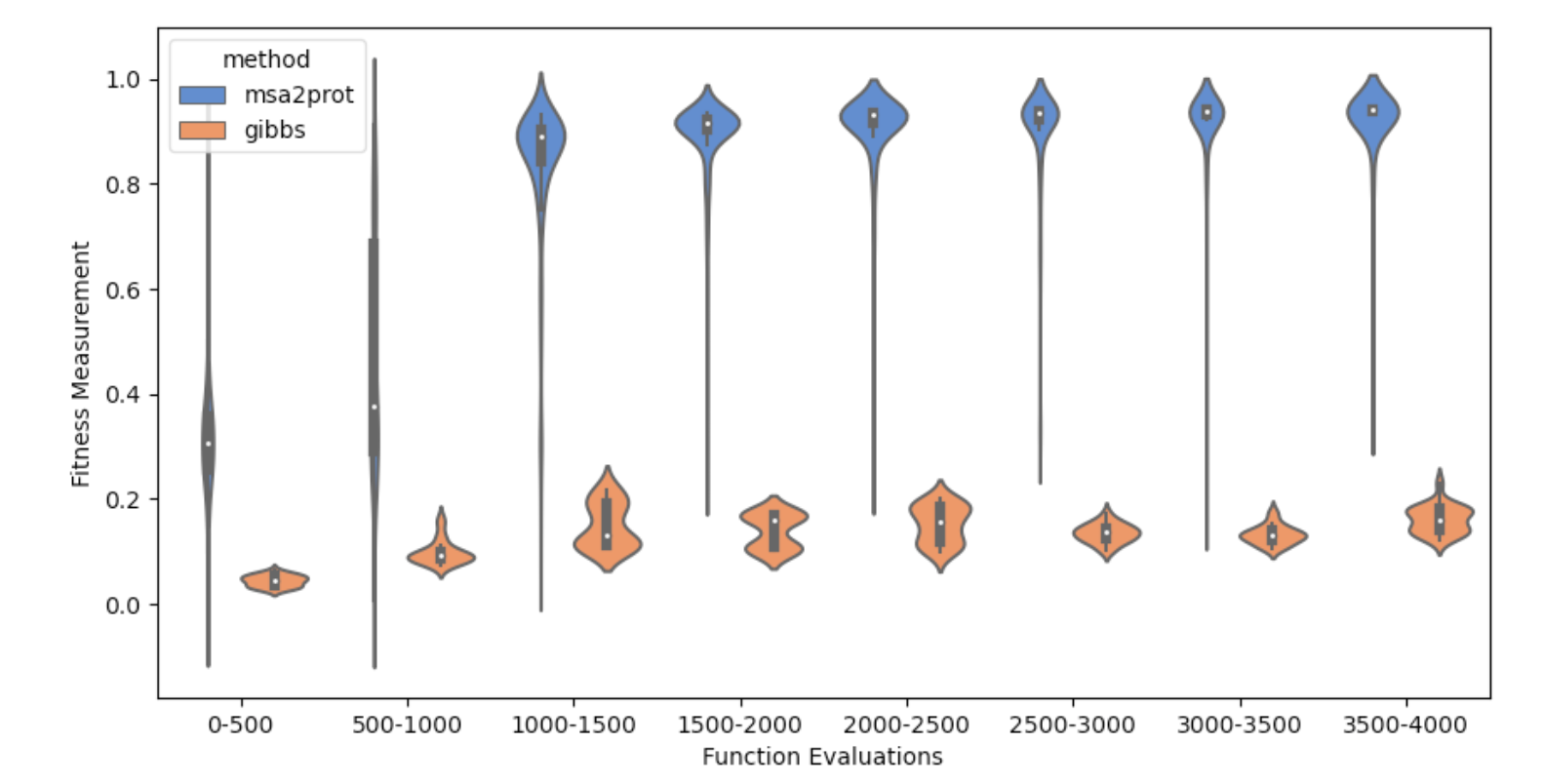
最后MSA2Prot除去根据其对数似然进行预测外,可以从高fitness的突变体中进行生成性推断。上图显示了通过自适应抽样在chorismate突变酶数据集上获得的生成样本fitness变化,横轴代表的以500条序列为间隔的更新次数。
具体而言,我们从训练的MSA中随机抽取100个序列,形成一个初始MSA。我们对序列进行抽样,如果回归器预测抽样的序列比MSA中的最小fitness序列具有更高的fitness,则更新MSA。上图结果表明,MSA2Prot能够有效地产生强突变体。MSA2Prot最初生成的序列的fitness为0.3,这是训练分布的第56个百分点。经过几千次更新,MSA2Prot产生的序列的fitness为0.9,处于训练分布的第89百分位。作为比较,吉布斯采样最初产生的序列的fitness为0.03,处于训练分布的第39百分位。在相同数量的函数评估之后,吉布斯抽样产生了一个fitness为0.3的序列,它位于训练分布的第56百分位。因此,MSA2Prot显示了相当强的性能。与标准的自适应抽样方法相比,MSA2Prot的好处是不需要训练。相反,MSA2Prot依赖于它的小样本泛化能力。因此,MSA2Prot为蛋白质序列设计提供了一种计算效率高的替代方法。
创新点
1.本文提出了MSA2Prot,一个基于Transformer的直接以MSA为条件的蛋白质序列生成模型。该模型允许对包括插入和删除以及替换在内的序列进行有效的采样和精确的对数似然评估,而不需要逐个在MSA上进行训练或微调。
2.MSA2Prot模型对未见过的家族有很好的泛化性,并且比其他家族生成模型更容易生成未见过的家族,特别是当MSA较小时。
3.本文证明了MSA2Prot在不同的突变体数据集上实现了最先进的突变体功能预测性能。
4.MSA2Prot不需要昂贵的重训练,通过只关注功能突变体的自适应采样,展示了序列生成的新能力。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢