摘要
蛋白质结构预测是计算生物学中一个长达半个世纪的重大挑战,最近,深度学习在这方面取得了前所未有的进展。4月1日,一项发表在 Nature Communications 上的最新论文从蛋白质结构预测、蛋白质功能预测、基因工程、系统生物学和数据集成以及系统发育推断五个方面探讨了深度学习的最新进展、局限性和对未来的展望。文章讨论了每个应用领域,并讨论了深度学习方法的主要瓶颈,如训练数据、问题范围,以及在新环境中利用现有深度学习架构的能力,最后总结了深度学习在生物科学领域面临的学科相关和一般性挑战。
研究领域:深度学习,结构生物学,蛋白质结构
郭瑞东 | 作者
转自:集智俱乐部

论文题目:
Current progress and open challenges for applying deep learning across the biosciences
论文链接:
AlphaFold2 成功地预测蛋白质3D结构问题,已成为深度学习在计算生物学领域的典型范例。Nature Communication 的综述“在生物学中应用深度学习的当前进展和开放挑战”,系统性介绍了在生物学中应用深度学习,当前取得的成果以及有待解决的开放性问题。
综述先介绍了在生物领域广泛应用的模型架构,指出所使用的模型包含有监督、无监督和强化学习三个范式。图神经网络、图像识别(CNN)以及自然预言处理(RNN,transformer)中的常见模型,都已在生物领域被成功应用。
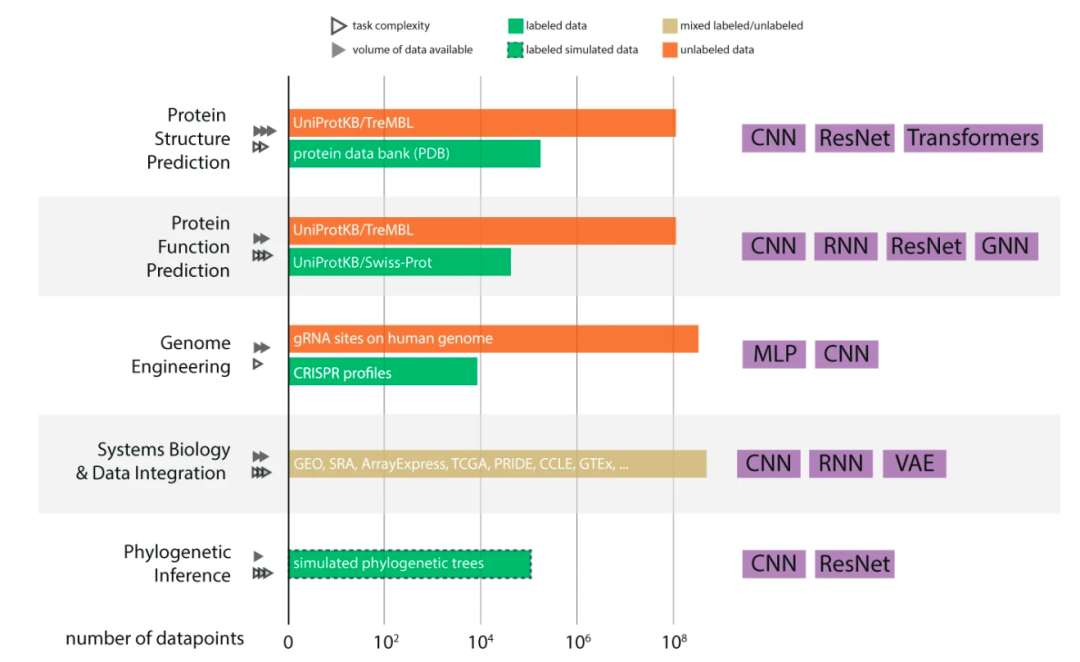
图1. 深度学习所需的数据集的数量级及常用模型
文中将取得的成就分为四种:范式革命、显著、中等和较小范围内的成功。按照应用场景,指出五类问题当前的进展、公开数据集的大小、常用的模型架构。

图2. 深度学习在各个领域取得的进展程度
综述指出,尽管深度学习在例如蛋白质结构预测取得了范式转移级的成功,改变了该领域的默认选项。在更多的领域,如蛋白质功能预测、基因工程(例如基因编辑)和多组学数据集成中也取得了长足的进步。然而相对于传统的方法,对于其他领域,例如系统发育推断、经典的计算方法在这些领域仍然占据上风。并不是所有深度学习的应用在计算生物学都同样成功。成功的领域高度依赖于具有多样性、无偏采样且贴近实际应用场景的大量有标注及无标准的标准数据集。
除了概述各领域的进展,该综述的亮点在于指出了在生物领域应用深度学习面对的一般性挑战和可能的解决方案:
1. 有偏的结论:即模型给出的结论和真实情况不符。面对这一问题,需要的是改进实验设计,避免训练数据和实际数据的分布有差异,还需要识别出模型有偏的原因,在针对性的使用更公平的模型。
2. 较高的计算成本:可解决方案是并行运算及优化代码,或选用部分(核心)数据进行训练,同时改进AI架构,使用更高能耗的计算设施。
3. 模型缺少解释性:对此一方面可以采用例如SHAP[1]的统计分析,对特征的重要性进行评估,或使用像GNNExplainer[2]这样的工具,对基于GNN的模型训练完成后给出解释。
4. 有限的训练数据集:可行的解决方案包括标注更多的数据,以及使用模型进行数据增强,基于现有数据集生成新的类似的训练数据。
5. 过拟合:应对方式包括正则化、丢弃神经元dropout、提早停止训练、使用容量更小的模型以及采用更多的训练数据。
综述着重强调了模型的可解释性以及训练过程中的效率,其中既包括了训练成本高,也包括了训练好的模型只适用于特定领域。并针对性地给出了更详细的可行建议。
参考文献:
1. Lundberg, S. M. & Lee, S.-I. A unified approach to interpreting model predictions. In Proceedings of the 31st International Conference on Neural Information Processing Systems, 4768–4777 (2017).
2. Ying, R., Bourgeois, D., You, J., Zitnik, M. & Leskovec, J. GNNExplainer: Generating explanations for graph neural networks. Adv. Neural Inf. Process. Syst. 32, 9240 (2019)
论文 Abstract
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢