【论文标题】Structure-aware Protein Self-supervised Learning
【作者团队】Can Chen, Jingbo Zhou, Fan Wang, Xue Liu, Dejing Dou
【发表时间】2022/04/06
【机 构】百度、麦吉尔大学
【论文链接】https://arxiv.org/pdf/2204.04213v1.pdf
蛋白质表征学习方法已经显示出巨大的潜力,可以为许多下游任务产生有用的表征,特别是在蛋白质分类方面。此外,最近的一些研究显示,用自监督学习的方法解决蛋白质标签不足的问题有很大希望。然而,现有的蛋白质语言模型通常是在蛋白质序列上进行预训练,而没有考虑重要的蛋白质结构信息。为此,本文提出了一种新的基于结构的蛋白质自监督学习方法STEPS,以有效地捕捉蛋白质的结构信息。具体而言,本文预训练了一个图神经网络模型,以保留蛋白质的结构信息,自监督的任务分别从成对残基距离的角度和二面角的角度进行学习。此外本文利用现有的预训练蛋白质语言模型对蛋白质序列进行预训练,以加强自监督学习,作者通过一个新颖的伪双重优化方案,确定了蛋白质语言模型中的序列信息和GNN模型中的结构信息之间的关系,并在几个监督式的下游任务上验证了提出的方法的有效性。
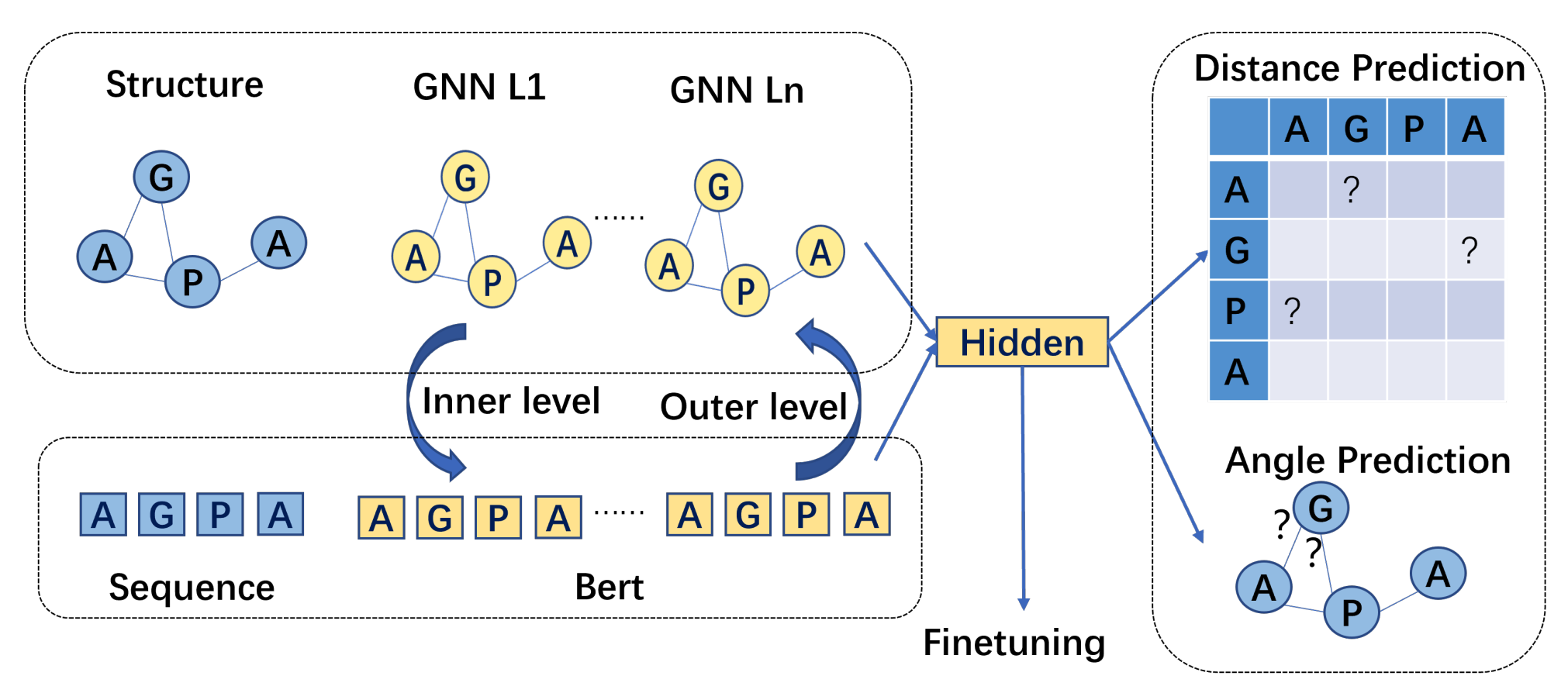
上图展示了GNN模型通过两个自我监督的任务捕获蛋白质结构信息:成对距离预测任务和二面角预测任务。此外,一个伪双重优化方案确定了蛋白质LM和GNN模型之间的关系,这增强了自监督学习的效果。
- 成对距离预测任务
本文利用距离预测网络NNdis,它将残基i和残基j的节点隐藏表征之间的向量差作为输入,旨在预测i和j之间的成对残基距离。这样做的目的是,残基的相互作用在决定蛋白质的不同功能方面起着重要作用。这里构建了一个多分类模型,将距离划分为T个bin进行预测。
- 角度预测任务
本文利用角度预测网络NNang(-),它将mask掉角度的残基表示作为输入以预测mask的角度。在这个过程中随机屏蔽15%的残基的归一化径向基角度特征,并将屏蔽的蛋白质送入GNN模型,从而得出被屏蔽的残基的隐藏表示。
- 伪双重优化
本文通过最小化自监督损失函数的方式来确定蛋白质LM和GNN模型之间的关系。这种关系是由蛋白质序列和蛋白质结构之间的约束所定义的,其中蛋白质序列决定了蛋白质结构。双重优化包括两部分,外层和内层,其中外层为调整GNN以影响LM,内层为调整LM以重建给定的GNN模型的蛋白质结构。这意味着LM和GNN之间的关系是由共同的蛋白质结构决定的
- 预训练
GNN模型2层1024维度在来自Deeploc的alphafold预测和酶数据库的40000个蛋白结构上进行预训练,LM模型使用ProtBert-BFD
- 微调
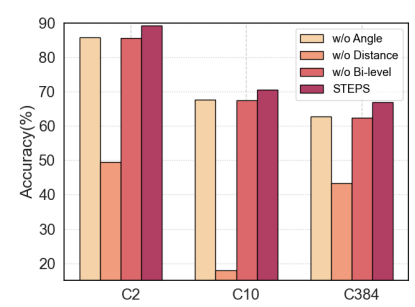
下游任务分为:C2(Membrane-bound vs Water-soluble二分类),C10(亚细胞定位十分类)和C384(酶催化反应EC编号384分类),消融实验表明双重优化和2个自监督任务都很重要。
创新点
- 第一个明确地将蛋白质结构信息纳入自监督学习的人,本文提出了两个新的自监督任务,分别捕捉成对残基距离信息和二面角信息。
- 采用了一种伪双重优化方案来利用蛋白质LM中的序列信息。
- 进行了各种监督的下游任务来验证STEPS的有效性。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢