【论文标题】Convolutions are competitive with transformers for protein sequence pretraining
【作者团队】Kevin K Yang, Alex X Lu, Nicolo K Fusi
【发表时间】2022/05/20
【机 构】微软
【论文链接】https://doi.org/10.1101/2022.05.19.492714
【代码链接】https://github.com/microsoft/protein-sequence-models
预训练的蛋白质序列语言模型在很大程度上依赖于Transformer的结构。然而,Transformer的运行时间和内存要求随着序列长度的增加而呈四次方变化。本文研究了基于卷积的架构在蛋白质序列遮蔽语言模型预训练和后续微调中的潜力。在预训练任务中,CNN与Transformer竞争力,同时资源需求与序列长度呈线性关系。更重要的是,在下游评估中CNN具有竞争力,有时甚至优于Transformer,包括结构预测、零样本突变效应预测和域外泛化。由于CARP的计算量与输入序列呈线性依赖,并且不依赖于输入的位置嵌入,因此可以直接应用于比长序列更长的序列。这些经验性的结果表明,需要通过分解结构和预训练任务的影响来加深本文对蛋白质序列预训练的理解。
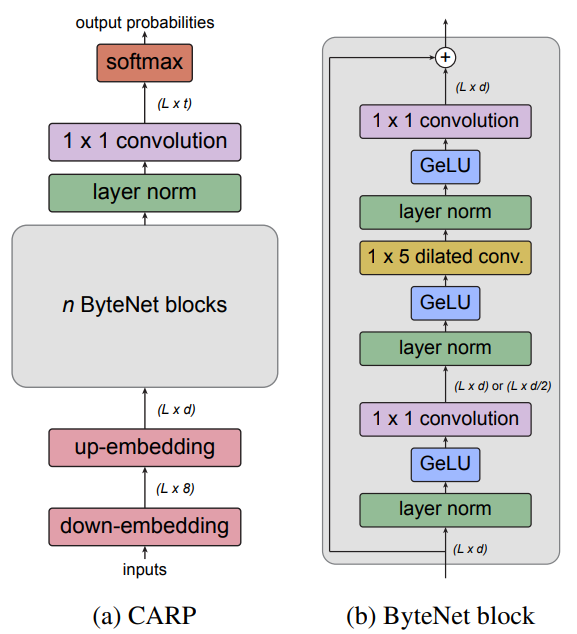
上图展示了CARP的架构,CARP结合了ByteNet编码器扩展的CNN架构和简单的输入嵌入和输出解码层。本文在UniRef50上训练蛋白质序列卷积遮蔽语言模型,称之为CARP(Convolutional Autoencoding Representations of Proteins)。在预训练任务中,这些模型在参数大小上与Transformer有几个数量级的竞争。最大的CARP,具有大约6.4亿个可学习参数(CARP640M),在各种下游预测任务上与目前最先进的Transformer蛋白质序列遮蔽语言模型ESM具有竞争力。
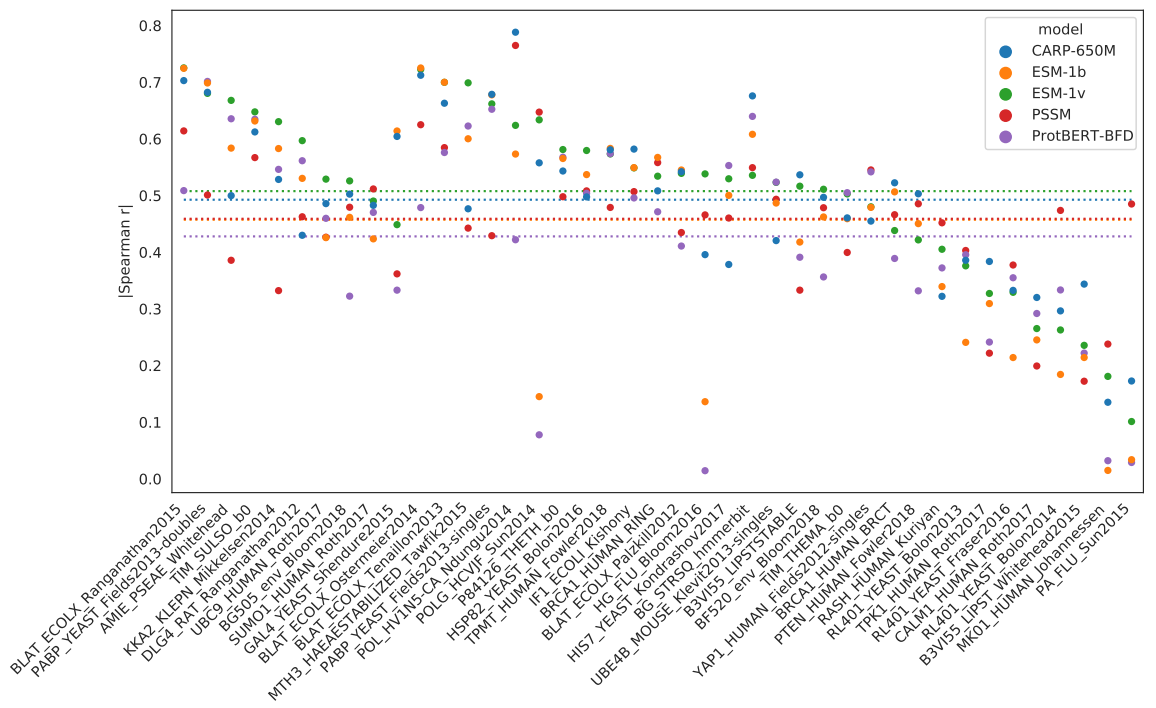
上图展示了零样本的蛋白质fitness预测。来自DeepSequence的41个深度突变扫描数据集的比较。点是每个数据集的Spearman相关性。水平线显示整个数据集的平均Spearman相关性。ESM-1b、ESM-1v、PSSM和ProtBERT-BFD的值取自ESM-1v实验 。从41个数据集的平均值来看,CARP-640M的Spearman相关性为0.49,而ESM-1b为0.46,ESM-1v为0.51,PSSM为0.46,ProtBERT-BFD为0.43。CARP-640M在41个数据集中有22个优于ESM-1b,在41个数据集中有18个优于ESM-1v,在41个数据集中有26个优于PSSM,在41个数据集中有25个优于ProtBERT-BFD。
ESM-1v文章发现,使用完整的UniProt序列而不是只使用变异结构域的序列,可以获得更好的零样本预测。然而,这在ESM-1x中并不总是可能的,因为这些蛋白质的一些UniProt序列长于1022个残基。作为进一步的概念证明POC,本文对来自化脓性链球菌的Cas9的突变效果进行了零样本预测,其长度为1368个残基,获得的Spearman相关性为0.26。这些结果表明,预训练的卷积可以对蛋白质突变对适配性的影响进行零样本预测,包括对长于ESM-1x所允许的序列。
创新点
本文证明,在MLM预训练任务和各种下游的蛋白质序列建模任务中,卷积都可以和Transformer相媲美,甚至优于Transformer,而且卷积和Transformer一样,都可以从预训练中获益。
在没有预训练的情况下,卷积和转化器在下游任务中的表现是不同的,这显示了将预训练和结构分开的重要性。与变换器不同的是,卷积与输入序列的长度呈线性关系,这在为长的蛋白质序列建模时变得很重要。自然语言处理方面的工作也表明,卷积可以比变换器需要更少的计算FLOPs,即使是短序列。
虽然本文使用的是标准的dilated 卷积,但也有为序列建模设计的更有效的卷积变体[Wu等人,2019],可能会进一步提高模型速度。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢