【论文标题】Design of Peptide-Based Protein Degraders via Contrastive Deep Learning
【作者团队】Kalyan Palepu, Manvitha Ponnapati, Suhaas Bhat, Emma Tysinger, Teodora Stan, Garyk Brixi, Sabrina R.T. Koseki, Pranam Chatterjee
【发表时间】2022/05/24
【机 构】MIT、哈佛、杜克大学
【论文链接】https://doi.org/10.1101/2022.05.23.493169
【数据链接】https://tinyurl.com/cutnclip
针对致病蛋白的治疗方式是多种疾病适应症的治疗金标准。不幸的是,这些蛋白质中的很大一部分被认为是 "不可药用 "的,主要是由于其无序的性质和不稳定性。因此,为不可药用的目标设计功能性肽,无论是作为独立的结合蛋白还是与有效domain的融合,都为治疗干预提供了独特的机会。本文调整了最近的对比性语言-图像预训练(CLIP)的模型,设计了一个统一的、基于序列的框架来设计靶点特异性多肽。此外,通过利用已知的实验结合蛋白作为支架,本文创建了一个简化的推理流程,称为Cut&CLIP,有效地选择多肽进行下游筛选。最后,本文通过实验将候选肽与E3泛素连接酶域相融合,并证明了人类细胞中致病蛋白目标的稳健的细胞内降解,促使技术进一步发展,用于未来的临床转化。
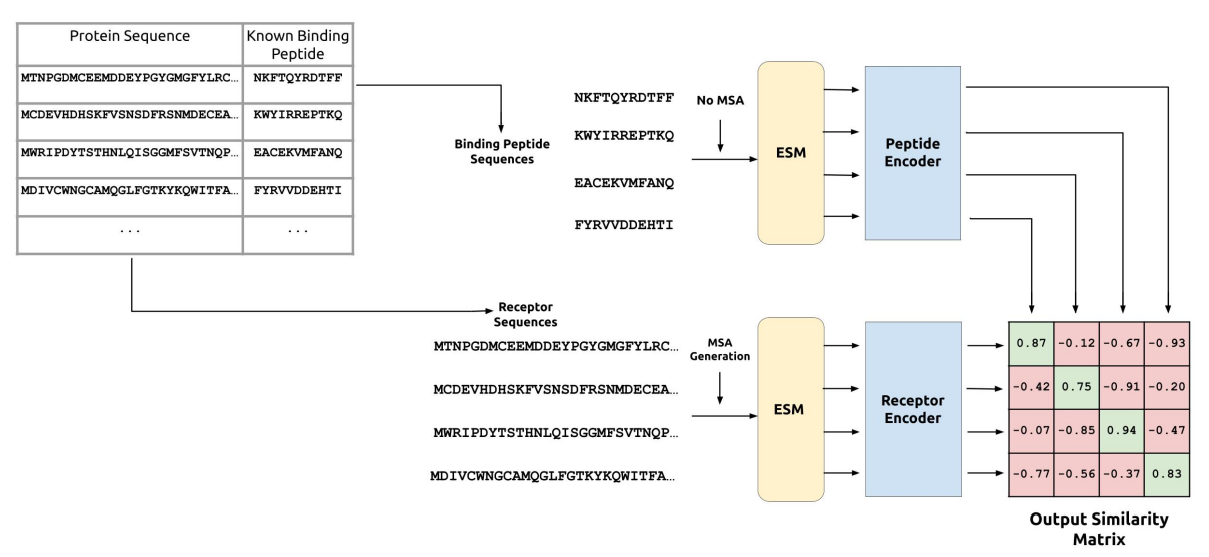
上图展示了 多肽-蛋白质对的CLIP训练过程。肽和受体编码器在ESM嵌入上进行联合训练,以预测已知的肽-受体嵌入对之间的高余弦相似度和所有其他对的低余弦相似度。具体来说,受体MSA和多肽序列首先被输入ESM预训练的Transformer蛋白语言模型。这些预训练的模型是在数百万个不同的氨基酸序列上训练的,使编码器能够提取特征丰富的嵌入,这些嵌入对序列的多样性具有鲁棒性,同时在一个相对较小的数据集上训练。本文对受体MSA采用ESM-MSA-1b模型,对肽序列采用ESM-1b模型,它不需要MSA输入。然后将这些ESM嵌入作为输入来训练受体和多肽的编码器。
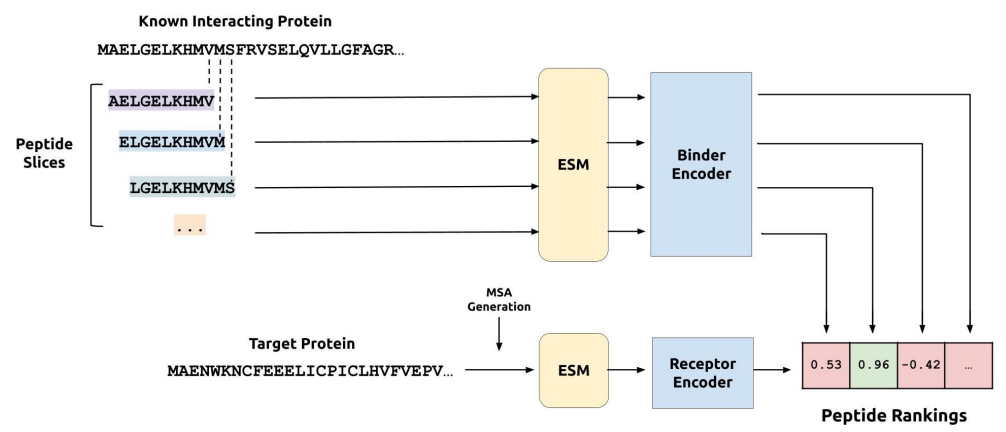
上图展示了Cut&CLIP推理流程。一个与目标蛋白相互作用的已知相互作用蛋白被切割成多肽片段,通过训练好的CLIP模型实现其下游ranking。
受之前利用目标蛋白和已知结合物的共晶体来设计有效的肽基降解剂的结果的启发,本文决定采用CLIP模型来预测结合肽,使用经过实验验证的相互作用的蛋白质来查询target。与本文以前的工作相比,目前的推理流程只需要从已建立的PPI数据库或实验筛选结果中获得潜在结合物的序列,使本文能够更灵活地识别起始scaffolds。然后,本文计算相互作用的蛋白质的所有k-mers的CLIP肽嵌入,并通过它们与目标蛋白的CLIP受体嵌入的余弦相似度进行排序。
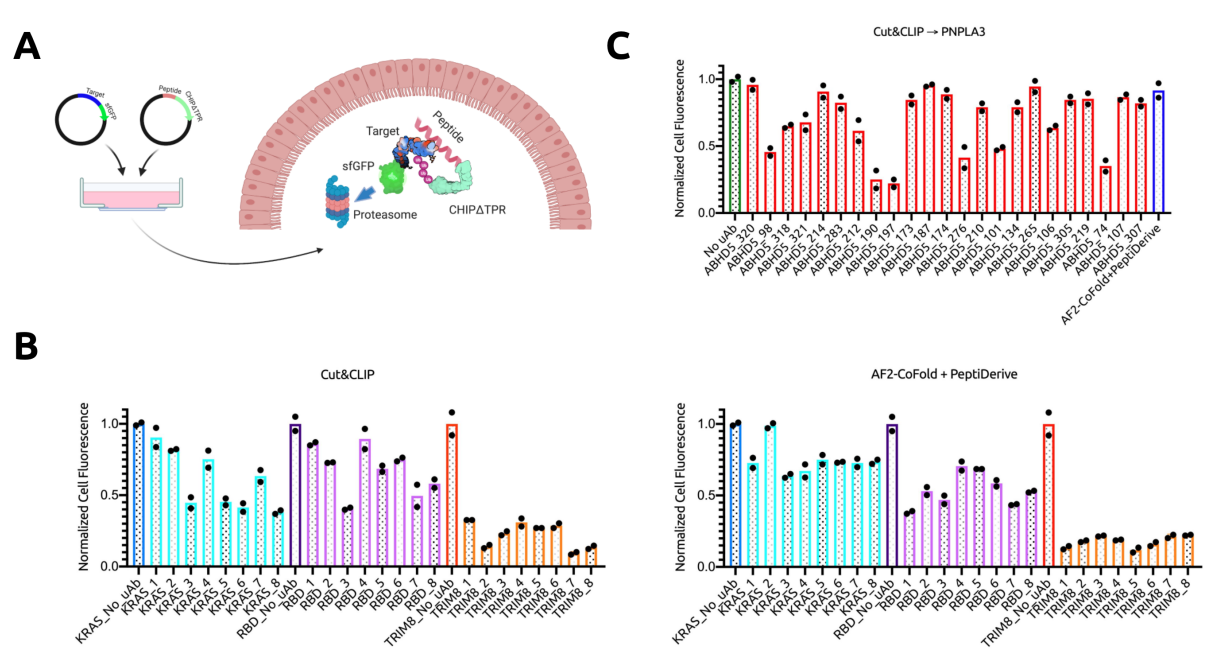
上图展示了Cut&CLIP的实验验证。为了评估Cut&CLIP与使用AlphaFold的较慢的基于结构的方法相比的效用,本文选择了三个目标蛋白进行实验表征:SARS-CoV-2的穗状受体结合域(RBD),TRIM8 E3泛素连接酶,以及KRAS肿瘤蛋白。将他们在PPI数据库的配对输入到Cut&CLIP管道,以及适应AlphaFold-Multimer复合物预测算法的colabfold流程后接PeptiDerive(AF2-CoFold+PeptiDerive)
A) uAb降解系统的结构和机制。CHIP∆TPR与靶向肽的C端融合,因此可以标记靶向sfGFP复合物,以便在质粒转染后,在蛋白酶体中进行泛素介导的降解。
B)通过流式细胞仪分析KRAS-sfGFP、RBD-sfGFP和TRIM8-sfGFP的降解。所有样品都在独立的转染重复中进行(n=2),并以GFP+荧光为门槛。归一化的细胞荧光是通过将样品的GFP+%除以其各自的 "无uAb "对照来计算。
C) 通过流式细胞仪分析PNPLA3-sfGFP的降解。所有样品都在独立的转染重复中进行(n=2),并以GFP+的荧光为门槛。归一化的细胞荧光是通过将样品中GFP+的百分比除以 "无uAb "对照的百分比来计算的。最终多肽来自PNPLA3-ABDH5的AF2-CoFold+PeptiDerive策略。
创新点
正如OpenAI的CLIP联合图像-文本嵌入空间使得像DALL-E 2这样的生成模型能够以标题嵌入为条件生成图像一样,本文的联合受体-肽嵌入空间自然表明了从头生成肽的机会。一旦有了目标蛋白的嵌入,最佳的肽嵌入是已知的,所有需要做的就是将肽嵌入解码成一个序列。这对于生成对靶点降解剂来说可能特别关键,尤其是对潜在的相互作用伙伴的实验数据最少的靶点。考虑到这种多功能性,与更多的标准模型相比,CLIP架构因此成为目标指定的肽设计的理想框架。
总的来说,这项工作代表了基于序列的语言模型在治疗相关的蛋白质设计中的初步应用。Cut&CLIP的未来迭代将纳入高亲和力肽设计的Kd值,并预测生成序列的脱靶倾向。通过将Cut&CLIP和uAb技术与有效的递送载体,如AAVs或LNPs,相结合,该肽引导的蛋白质降解平台最终可能成为一种有效的治疗策略,以解决一系列被认为无法用标准的小分子手段治疗的疾病。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢