【论文标题】ODBO: Bayesian Optimization with Search Space Prescreening for Directed Protein Evolution
【作者团队】Lixue Cheng, Ziyi Yang, Benben Liao, Changyu Hsieh, Shengyu Zhang
【发表时间】2022/05/20
【机 构】腾讯
【论文链接】https://arxiv.org/pdf/2205.09548v2.pdf
【代码链接】https://github.com/sherrylixuecheng/ODBO/
定向进化是蛋白质工程中的一种通用技术,它模仿自然选择的过程,在诱变和筛选之间反复交替进行,以寻找能优化某一感兴趣的属性的序列,如催化活性和与特定目标的结合亲和力。然而,可能蛋白质空间太大,无法在实验室中进行详尽的搜索,而且在庞大的序列空间中,功能性的蛋白质非常稀少。机器学习方法可以加速定向进化,通过学习将蛋白质序列映射到功能,而不需要建立基础物理学、化学和生物途径的详细模型。尽管这些机器学习方法拥有巨大的潜力,但它们在为目标功能识别最合适的序列时遇到了严重的挑战。这些失败可以归因于对蛋白质序列采用高维特征表示的普遍做法和低效的搜索方法。为了解决这些问题,本文提出了一个高效的、以实验设计为导向的蛋白质定向进化闭环优化框架,称为ODBO,它采用了新的低维蛋白质编码策略和贝叶斯优化的组合,通过离群点检测对搜索空间进行预筛选。作者进一步设计了一个初始样本选择策略,以尽量减少用于训练机器学习模型的实验样本数量。本文进行并报告了四个蛋白质定向进化实验,证实了所提出的框架在寻找具有感兴趣属性的变体方面的能力。作者希望ODBO框架能大大降低定向进化的实验成本和时间成本,并能在更广泛的范围内进一步推广,成为fitness实验设计的有力工具。
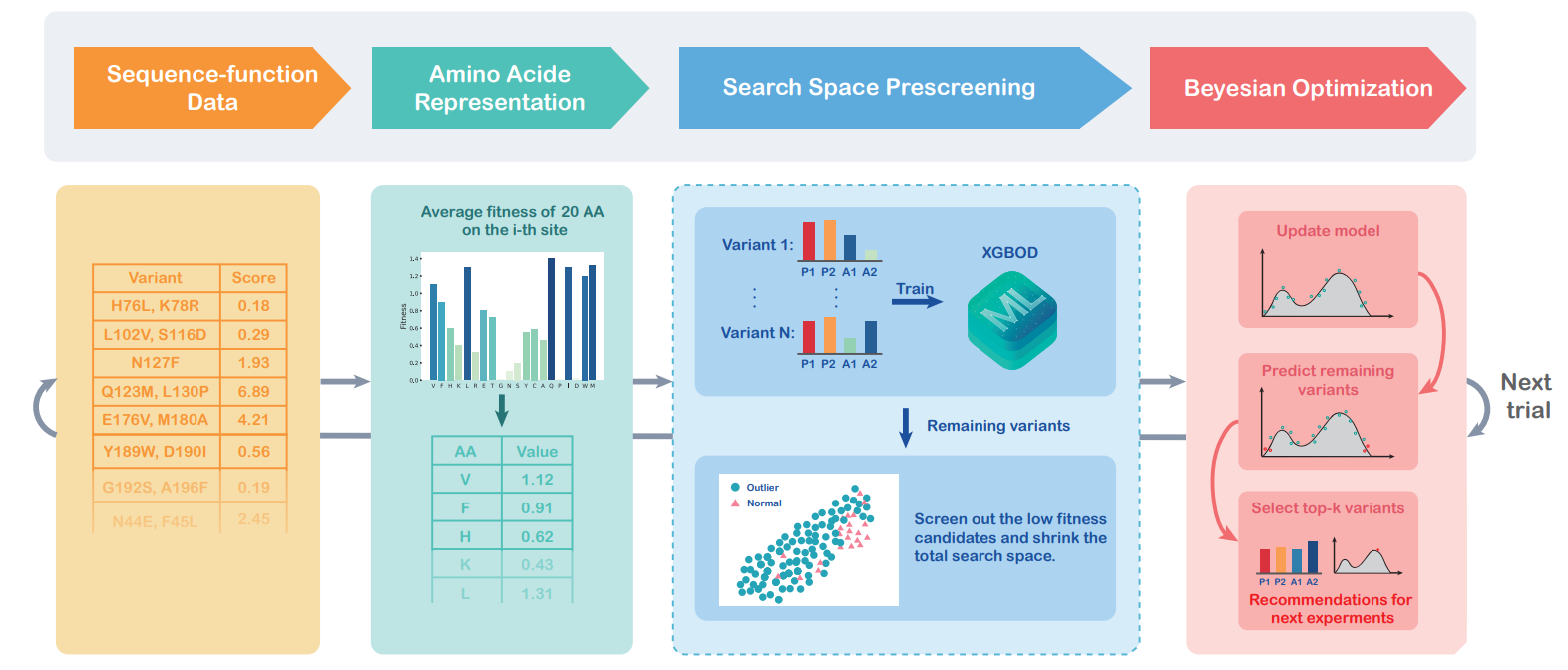
上图展示了ODBO,一个用于蛋白质定向进化闭环优化的新框架。首先准备好蛋白质的序列和各自的功能测量。随后,蛋白质的氨基酸由提议的基于功能值的编码策略进行编码。蛋白质的矢量表示被输入到XGBOD,用于搜索空间的预筛选,它可以积极地缩小搜索空间的大小,以过滤掉潜在的低fitness样本。在优化过程中,随着观察次数的增加,可以选择更新缩小的搜索空间和XGBOD模型。最后,一个适当的贝叶斯优化算法被用来在缩小的搜索空间内推荐下一轮的实验样本。
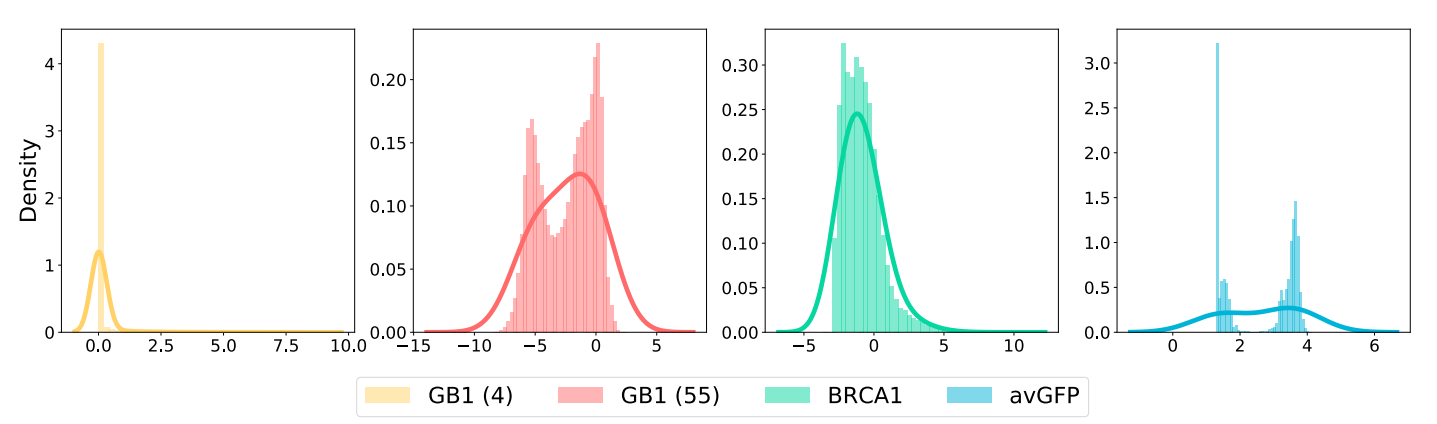
为了验证所提方法的有效性,本文对四个蛋白质定向进化数据集进行了实验,其中包括三个代表性的蛋白质,即蛋白质G B1结构域、BRCA1 RING结构域和绿色荧光蛋白,包括两种实验方案,即k位点的饱和诱变和非饱和诱变。上图显示了不同数据集的fitness属性值分布。
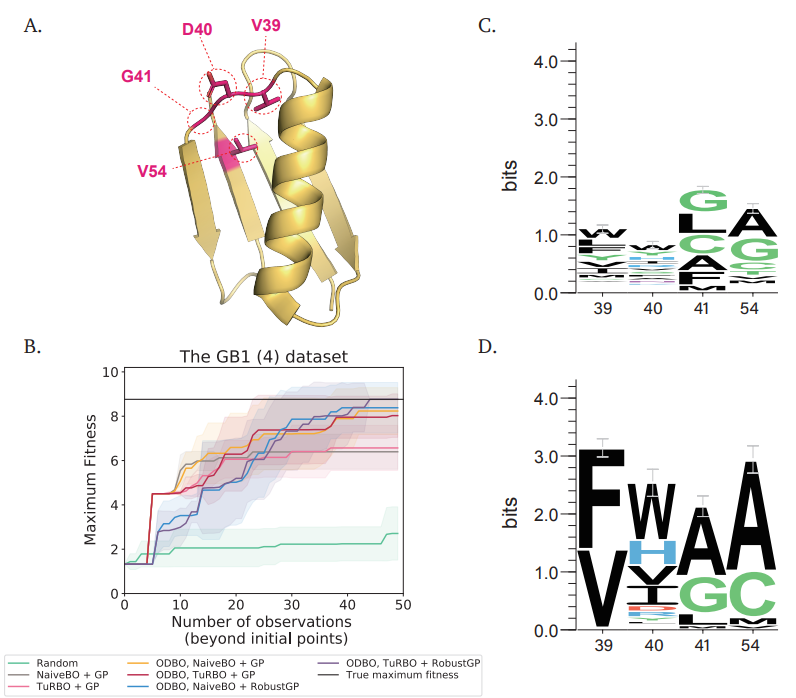
上图展示了GB1(4位点饱和突变)数据集的贝叶斯优化在40次初始实验和50次迭代的结果,以及搜索结果的生物学解释。
(A)GB1蛋白(PDB ID:2QMT)三维结构上的四个突变点。突变位点用红色圆圈突出显示
(B) GB1 蛋白质数据集上不同BO搜索方法的比较。线条代表了10次独立搜索运行的平均最大fitness,相应的阴影区域代表了相关的标准偏差。
筛选的初始变体集可以是随机选择的,也可以是为了最大化考虑的变异信息。随机选择变体是最简单的方法。然而,由于实验成本高,筛选产量低,必须最大限度地利用从昂贵的实验中获得的信息,以进一步提高对未见过的序列的模型准确性。因此,明智地选择待测的初始变体对于确保贝叶斯优化的有效搜索至关重要。在本文中,本文提出了一种初始样本选择策略,从候选序列的空间中反复选择信息量最大的变体,确保所有20个氨基酸在所选序列中至少出现一次。如果没有初始实验数据,实验者可以应用这一策略,输入每个氨基酸在每个位置j或所有位置的出现次数,得到一个初始样本集。
本文对这个数据集测试了七个建议的优化方法,包括 :
- a. 为下一轮随机选择的实验样本("随机");
- b. 带有GP代理模型的Naive BO("NaiveBO + GP");
- c. 带有GP代理模型的TuRBO("TuRBO + GP");
- d. 带有GP代理模型的Naive BO和通过XGBOD预先筛选的搜索空间("ODBO, NaiveBO + GP");
- e. 带有GP的TuRBO和通过XGBOD预先筛选的搜索空间("ODBO, TuRBO + GP");
- f. 带有RobustGP的Naive BO和通过XGBOD预筛选的搜索空间("ODBO, NaiveBO + RobustGP");
- g. 带有RobustGP的TuRBO和通过XGBOD预筛选的搜索空间("ODBO, TuRBO + GP")。
本文对所有的优化方法都使用了预期改进(EI)获取函数和1的批次大小。
(C) 整个GB1数据集的前1%实验的序列标识(fitness大于2.15)。
(D) 通过ODBO、TuRBO+RobustGP搜索出的fitness大于2.15的实验的序列标识。可以看到只有两个氨基酸在39号位点被BO选中,这两个氨基酸是整个数据集中第三和第五个富集的选择。对于40、41和54位点,图C和D中最常出现的氨基酸也是类似的。在ODBO的结果中,只有少数特定的氨基酸出现在39、41和54位点,而在40位点的氨基酸分布比较均匀。另外可以注意到,与每个位点上最频繁的氨基酸结合的序列是实际的最佳变体(FWAA,fitness=8.76)。
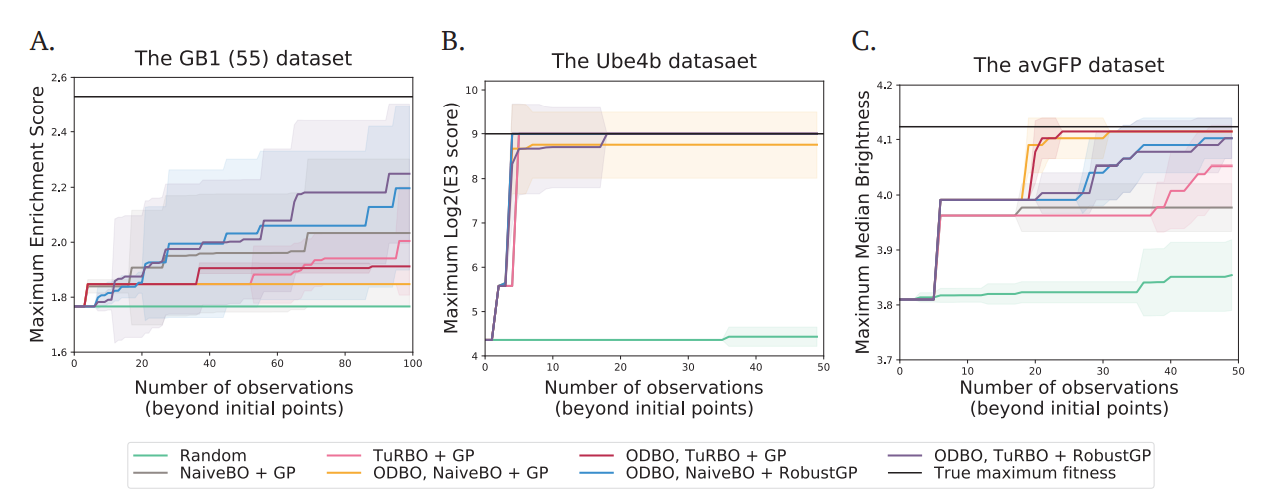
上图显示了其他非饱和突变数据集上优化的结果。对于GB1(55)数据集,每个竞争方法运行100次,批次大小为q=1;对于Ube4b和avGFP数据集,运行50次,批次大小为q=1。曲线绘制了10次独立优化运行的平均结果,阴影部分显示了10次运行的相应标准偏差。
在所有的测试案例中 在所有的测试案例中,从BO获得的平均最大fitness都优于从随机搜索获得的fitness。GB1 (55) 是最具挑战性的案例,没有任何一种搜索策略能成功地找到数据集中实际的最佳富集度。ODBO, BO + RobustGP和ODBO, TuRBO + RobustGP方法为 GB1 (55). 所有测试的算法对Ube4b数据集都显示出极高的效率,而且大多数的BO 算法在10次搜索迭代内成功找到了真正的最大值(9.00)。此外,所有的ODBO算法都达到了接近真实最大值的平均亮度(亮度=4.12)。这些观察表明 本文的基于函数值的特征化和BO算法在这些非饱和突变的数据集上也可以表现良好。
创新点
- 本文引入了一个高效的ODBO框架,通过整合离群点检测和贝叶斯优化的搜索空间预筛选,来解决具有大搜索空间的高维和异质科学问题的实验设计。为了实现成功的全局优化搜索,本文针对蛋白质定向进化的两个实验场景,提出了一种新颖的、基于低维函数值的蛋白质编码策略。
- 本文还设计了一个初始样本选择策略来协助实验者,它可以从候选序列空间中迭代选择信息量最大的变体作为初始样本。
- 本文的实证结果表明,所提出的框架可以在四个蛋白质定向进化数据集上提供卓越的性能,同时以最小的实验测量提供生物的可解释性。ODBO框架适用于k位的饱和诱变,并且对不同的批次大小和获取函数的选择都很稳定。搜索空间预筛选和基于RobustGP的代用模型的引入导致了更有效的样本采集,促进了更快地找到最高适配度的样本。此外,与天真的BO算法相比,应用具有更好的开发探索平衡的TuRBO优化算法可以进一步提高最优变体的搜索效率。
- ODBO框架可以很容易地被推广到更广泛的适应性实验设计中。未来工作的一个可能方向是将ODBO应用于不同的控制实验问题,特别是那些具有大搜索空间的问题,并向实验者推荐信息丰富的后续实验,以节省湿法实验室的巨大实验资源。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢