【论文标题】Contrastive Representation Learning for 3D Protein Structures
【作者团队】Pedro Hermosilla, Timo Ropinski
【发表时间】2022/05/31
【机 构】乌尔姆大学
【论文链接】https://arxiv.org/pdf/2205.15675v1.pdf
从三维蛋白质结构中学习已经在蛋白质建模和结构生物信息学中获得了广泛的关注。不幸的是,可用的结构数量比计算机视觉和机器学习中常用的训练数据量要低几个数量级。此外,当只考虑有注释的蛋白质结构时,这一数量甚至进一步减少,使得现有模型的训练变得困难,容易出现过度拟合。为了应对这一挑战,本文为三维蛋白质结构引入了一个新的表征学习框架,使用无监督的对比学习来学习蛋白质结构的有意义的表征,利用蛋白质数据库中的蛋白质。本文展示了这些表征如何被用来解决大量的任务,如蛋白质功能预测、蛋白质折叠分类、结构相似性预测和蛋白质-配体结合亲和力预测。此外,本文还展示了用预训练及微调网络如何使得任务性能的显著提高,在许多下游任务中取得新的最先进的结果。
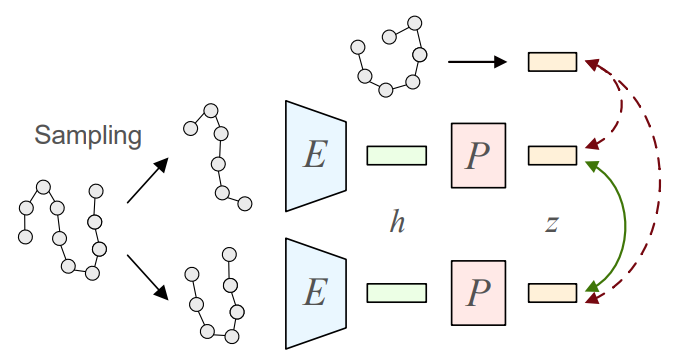
受最近计算机视觉领域工作的启发,本文的框架是通过最大化同一蛋白质子结构的表征之间的相似性,以及最小化不同蛋白质子结构之间的相似性来训练的。对于每个蛋白质,本文从中抽取两个子结构Gi和Gj。然后,本文使用蛋白图编码器hi = E(Gi)来计算这些子结构的潜在表示,hi和hj。本文进一步将这些潜在表征投射到更小的潜在表征中,即zi和zj,使用具有单个隐藏层的多层感知器(MLP),zi=P(hi)。最后,使用余弦距离(s(zi , zj))来计算这些表征之间的相似性。然后,本文最小化同一蛋白质的表征z之间的距离,最大化不同蛋白质的表征之间的距离。
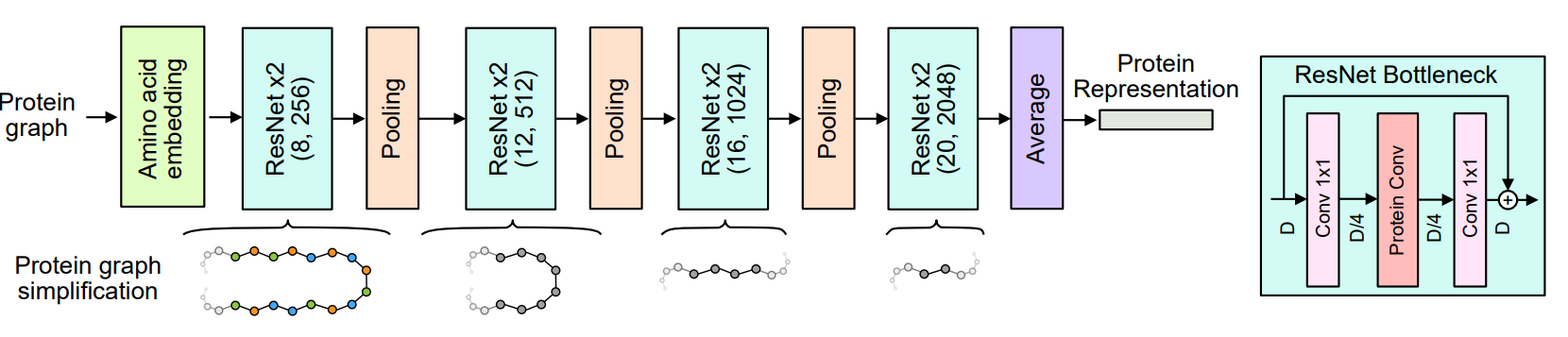
本文的蛋白图构建方式为,节点特征为alpha碳坐标,边特征为朝向,序列距离、空间距离。本文使用一个氨基酸嵌入作为本文的输入特征,然后通过连续的ResNet Bottleneck块和池化操作来处理。为了获得最终的蛋白质表征,本文使用来自其余图节点的特征的平均值。
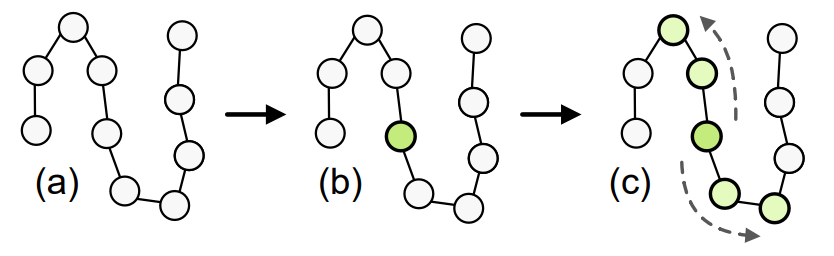
蛋白质链由一个或几个稳定的亚结构组成,称为蛋白质结构域,这些结构在不同的蛋白质中重复出现。这些子结构可以表明不同蛋白质之间的进化历史,以及蛋白质所执行的功能。
本文的采样策略使用蛋白质子结构的概念,对每个蛋白质沿多肽链的两个不同的连续子结构进行采样。本文通过首先在蛋白质链中随机选择一个氨基酸xi∈N来实现这一目的。然后,本文使用邻接矩阵A沿着蛋白质序列的两个方向行进,同时在这个过程中选择每个氨基酸xi+t和xi-t。这个过程一直持续到本文覆盖了蛋白质链的一定百分比p,据此本文的实验表明,p值在40%和60%之间可以提供最好的结果。如果在这个采样过程中,本文在其中一个方向上达到了序列的终点,本文就继续在另一个方向上采样,直到本文覆盖了所需的百分比p。所选的氨基酸组成了子结构,然后作为输入给图编码器E。请注意,由于本文的框架是从无标记的数据中学习的,所以本文不从蛋白质链中专门抽取蛋白质域,这就需要注释。本文取而代之的是随机的子结构,这些子结构可能由一个完整的或部分的结构域组成,或者,在大的蛋白质中,甚至跨越几个结构域。然后,训练目标是对同一蛋白质链的随机子结构进行类似的表示,其中完整结构的属性必须被推断出来。本文将在实验中表明,这些表示法能够编码描述结构模式和蛋白质功能的特征。
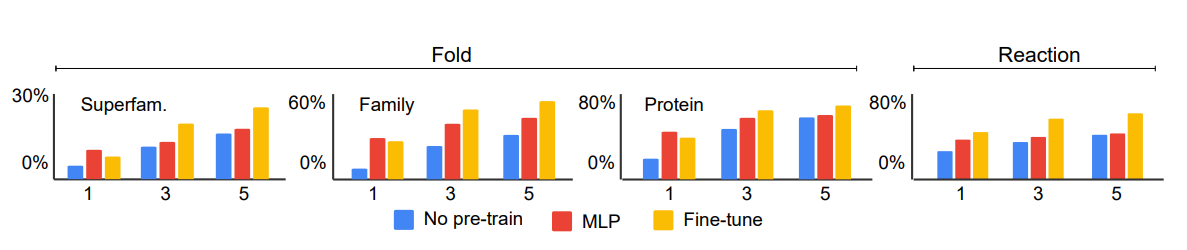
fold和酶分类任务的准确度与每类注释的蛋白质数量有关。本文评估了当每类注释的蛋白质数量有限时,框架的三个版本的稳健性。上图显示了在训练期间每类只有1、3或5个蛋白质时的结果。当每类只有1个蛋白质时,使用预训练的表征(MLP)比从头开始训练模型(无预训练)取得了更高的准确性,比微调(微调)的准确性更高。然而,如果蛋白质的数量增加到3个或5个,微调就比两者都好。这些实验说明,我们的预训练框架在处理小数据集时提高了泛化能力,减少了过度拟合。
创新点
在本文中,本文介绍了蛋白质结构的对比性学习。虽然对蛋白质结构的学习已经显示出显著的效果,但它受制于注释数据集的可用性相当低,这增加了对无监督学习技术的需求。在本文中,本文证明了通过将蛋白质感知的数据转换与最先进的学习技术相结合,本文能够在不需要这种注释数据的情况下获得一个学习表征。这是非常有益的,因为与序列数据相比,有注释的三维结构的可用性是有限的。
此外,本文已经表明,使用本文预训练的模型,本文可以在一大批相关的蛋白质任务上实现新的最先进的性能。本文相信,本文的工作是将无监督学习方法迁移到大规模蛋白质结构数据库的第一个重要步骤。
在未来,本文可以预见,学习到的表征不仅可以用来解决本文所展示的任务,而且还可以帮助解决其他蛋白质结构问题。例如,蛋白质-蛋白质相互作用的预测,可以用所学的表征之间的余弦距离来解决。
此外,本文计划发布所有PDB蛋白质的表征,并使本文的技术可用,从而使这些表征可以被新发现的蛋白质所使用。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢