近日,电子信息与电气工程学院自动化系沈红斌教授、袁野副教授的课题组(模式识别与生物信息学课题组)在《自然-计算科学》(Nature Computational Science)上发表题为《利用图神经网络对空间转录组数据进行细胞聚类》(Cell clustering for spatial transcriptomics data with graph neural networks)的研究论文。

论文链接:
论文提出了一种基于图卷积神经网络的空间转录组细胞聚类方法(Cell Clustering for Spatial Transcriptomics,CCST)。此前针对该类数据的处理模型大都基于“同种细胞在空间上相互临近”的这一假设。相比之下,CCST并不依赖于局部特征,而是可以从细胞的全局空间分布中学习节点嵌入。具体方案为:首先将空间转录组建模为图结构的数据。图中每一个节点代表一个细胞,其原始表征为高维的基因表达信息。图的邻接矩阵则依据细胞之间的空间距离建立。随后分别提取图上的局部特征和全局特征,以最大化局部和全局特征间的互信息为目标,训练图神经网络模型,从而得到带有全局结构信息的节点嵌入,再对各细胞所对应的表示向量进行聚类来识别细胞类型,最后从生物功能角度对聚类结果进行了深入分析。
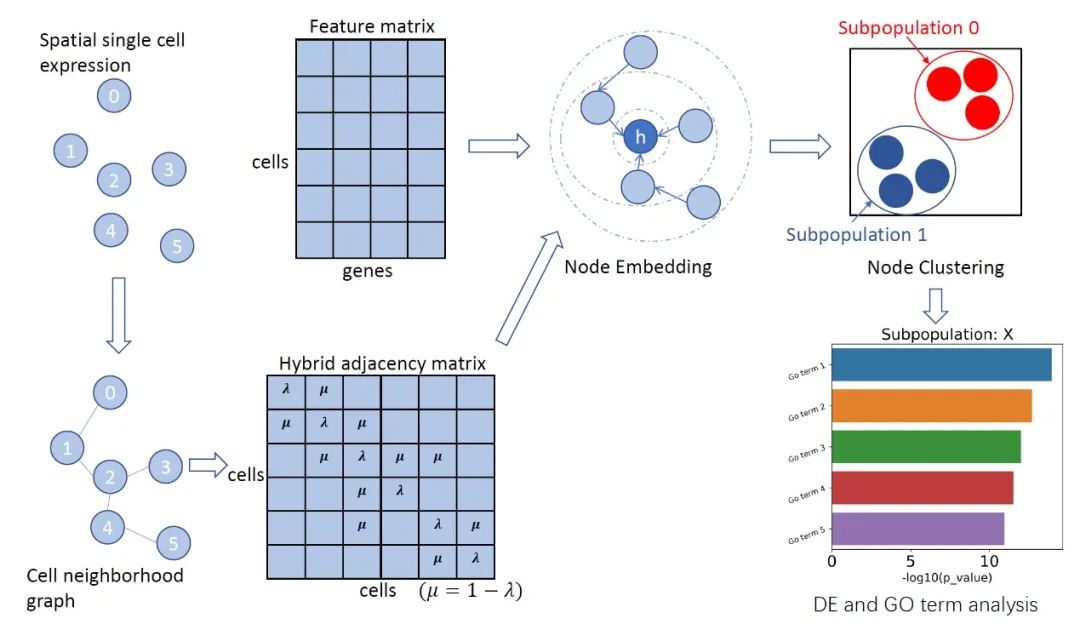
CCST方法示意图
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢