本文介绍一篇来由美国NEC实验室Erik Kruus等人于2022年8月在线发表在KDD上的文章。在这篇文章中,作者团队提出通过TCR-肽对的物理建模来扩展训练集,以解决当前数据集中出现的数据稀缺问题。实验证明,用物理建模和数据增强的伪标记来训练深度神经网络,比现有两个数据集中的基线有所改进。

论文链接:
https://dl.acm.org/doi/10.1145/3534678.3539075
源码:
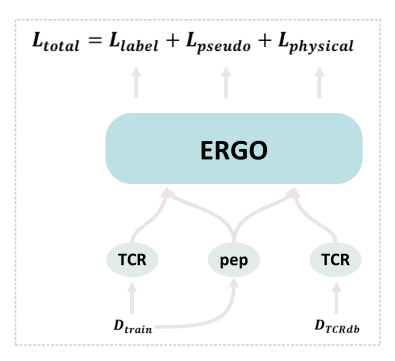
方法概述
作者团队提出的方法训练了一个深度学习模型,用于从3个损失中预测TCR肽相互作用(如上图所示):1)来自给定已知TCR-肽对的监督交叉熵损失;2)基于未知TCR-肽对的对接能量的监督交叉熵损失;3)来自伪标记(通过教师模型)未知TCR-肽对KL-divergence损失。
作者团队使用ERGO-I作为所有实验的基础模型,ERGO-II通过进一步考虑辅助信息,即CDR3的链、V和J基因、MHC类型和T细胞类型。作者选择ERGO-I而不是ERGO-II的原因如下:作者们的目标是验证预测两个分子的相互作用的机器学习模型可以进一步物理建模来改进。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢