【论文标题】Predicting enzyme substrate chemical structure with protein language models
【作者团队】Adrian Jinich, Sakila Z. Nazia, Andrea V. Tellez, Dmitrij Rappoport, Mohammed AlQuraishi, Kyu Rhee
【发表时间】2022/10/02
【机 构】康奈尔、加州大学尔湾分校、哥大
【论文链接】https://doi.org/10.1101/2022.09.28.509940
未注释的或孤儿酶的数量大大超过了那些已知底物化学结构的酶的数量。虽然存在一些酶的功能预测算法,但这些算法通常预测的是酶EC编号或酶家族,这限制了它们在实验上可检验的能力。本文利用蛋白质语言模型、化学信息学和机器学习分类技术,通过预测底物的化学结构类别来加速孤儿酶的注释。本文以结核分枝杆菌的孤儿酶为案例,关注其蛋白质组中高度丰富的两个蛋白质家族:短链脱氢酶/还原酶(SDRs)和依赖S-腺苷蛋氨酸的甲基转移酶(SAM-MTase)。从预训练的、自监督的蛋白质语言模型ESM transformer中获得的蛋白质序列嵌入作为输入训练的机器学习分类模型,可以对各种各样的预测任务有很好的准确性。其中包括SDRs的氧化还原辅助因子偏好,SAM依赖性甲基转移酶的小分子与聚合物(即蛋白质、DNA或RNA)底物偏好,以及对两个酶家族的首选底物进行更详细的化学结构预测。然后,本文用这些训练好的分类器对结核杆菌和其他分枝杆菌的蛋白质组中未加注释的全部SDR和SAM-MTase进行预测,产生了一套可进行生化测试的预测。本文的方法可以扩展和普及到其他酶家族和蛋白,而且它将有助于加速大量孤儿酶的注释。
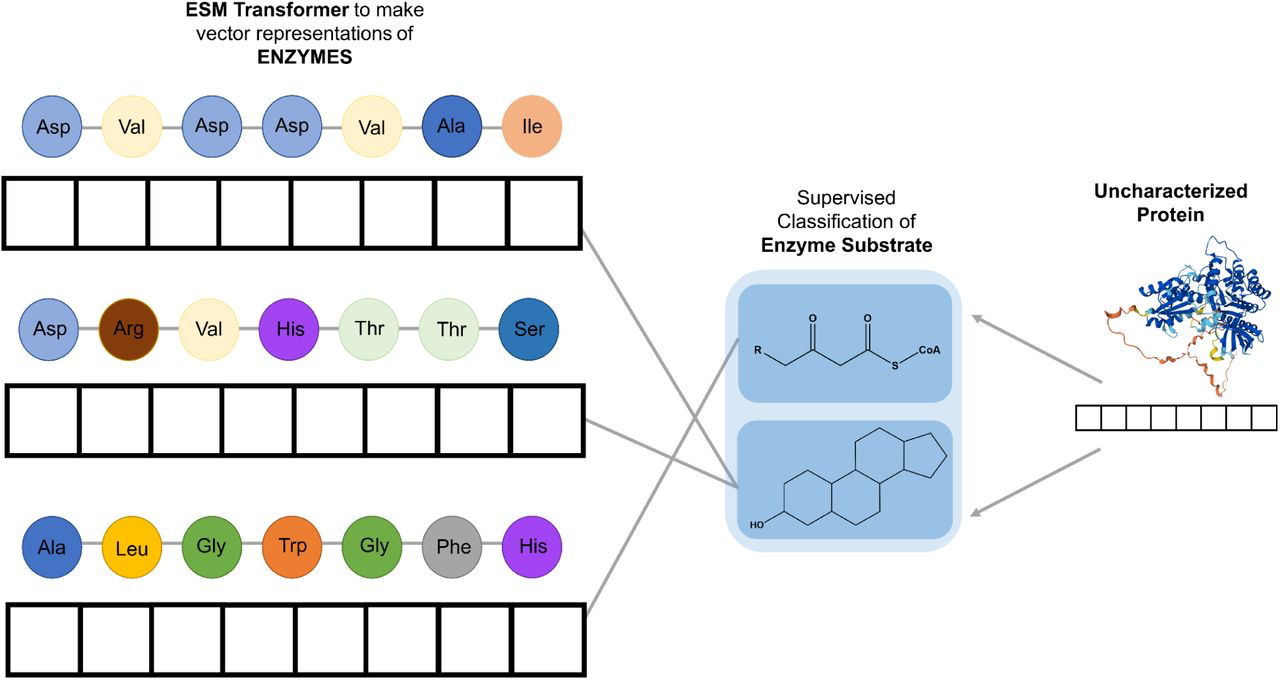
上图展示了基于语言模型的底物分类预测总体框架
- 数据
首先利用蛋白质家族的InterPro数据库获得SDR和SAM-MTase家族成员的条目,使用这些InterPro条目作为查询,以获得UniProtKB中的SDR和SAM-MTase蛋白集,并获得了2,665,904个SAM-MTases和1,344,540个SDR。然后使用Rhea生化反应数据库和ChEBI数据库来获得与每个酶相关的注释的生化反应(底物、产物和辅助因子)。最后训练时将UniProtKB中注释分数为4-5分及以上以及Rhea数据库中具有单一首选氧化还原辅助因子的酶作为训练集的一部分,由此产生的标签被进一步人工处理。
- 聚类
为了按分子结构类别对酶的底物和产物进行分组,本文将UniProtKB与Rhea数据库和ChEBI的标识符进行交叉引用,以获得与被注释的SDR或SAM-MTase相关的每个底物和产物的SMILES字符串表示。虽糊rdkit从每个化合物的SMILES表示中,计算出相应的摩根指纹。最后使用scikit-learn中实现的K-means聚类法来生成N个分子结构聚类,
- 模型训练
使用ESM-1b的33层全局平均的1280维向量作为表征,同时训练scikit-learn中实现的逻辑回归和xgboost库中实现的梯度提升树,用于所有监督分类任务。具体使用5折交叉验证,与SMOTE来纠正类别的不平衡,使用auc评价。
训练过程中,一些聚类导致的分类任务平衡性很差,即它们只有少量的注释酶的底物映射到这些聚类中,这些聚类被排除在训练外。
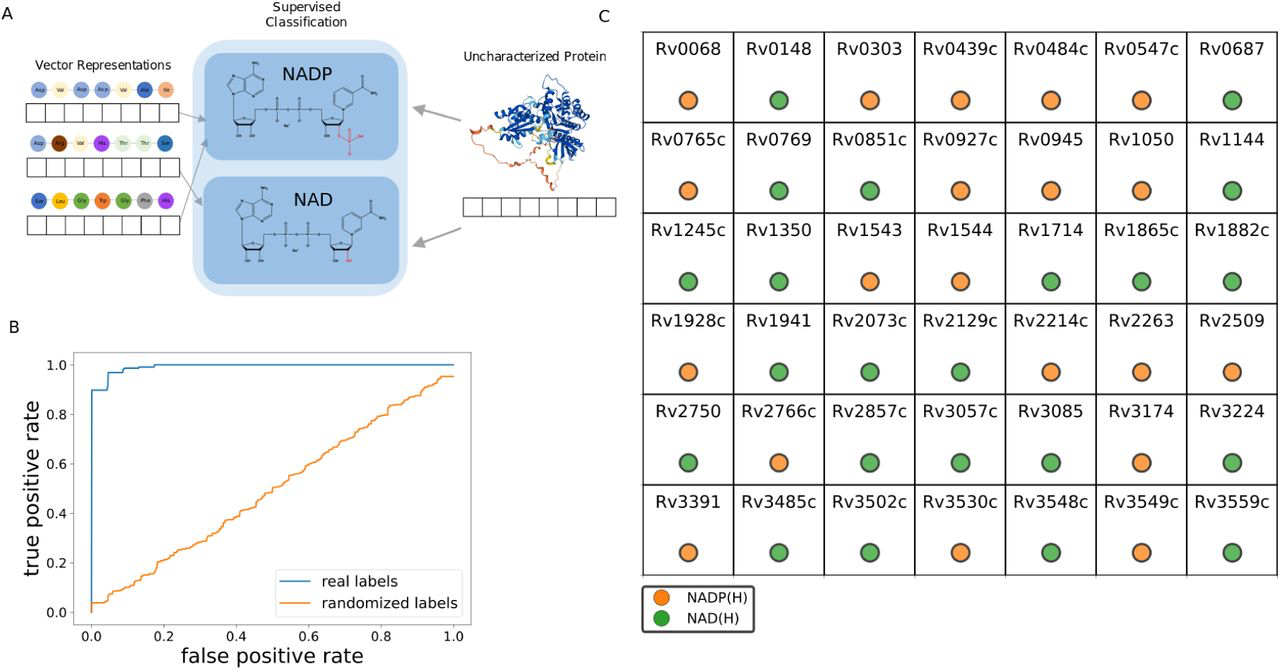
本文的第一个酶的功能预测任务集中在预测属于SDR酶家族的蛋白质的氧化还原辅助因子的偏好上。
作者从UniProtKB获得了338个SDR酶的序列和它们相应的氧化还原辅助因子偏好。通过上文描述的过程训练逻辑回归机器学习模型的氧化还原辅助因子分类器达到了很好的预测精度(AUC-ROC = 0.98)。
然后作者用训练好的逻辑回归分类器来预测结核杆菌和烟草杆菌中所有孤儿SDR的氧化还原辅助因子偏好。本文发现结核杆菌中的孤儿SDR在辅助因子偏好方面的分布大致相同:在42个孤儿SDR中,23个被预测为对NAD(H)有偏好,19个对NADP(H)有偏好。在烟草杆菌的情况下,本文发现预测对NADP(H)有偏好的孤儿SDR的比例略高:在169个孤儿SDR中,79个预测对NAD(H)有偏好,90个对NADP(H)有偏好。
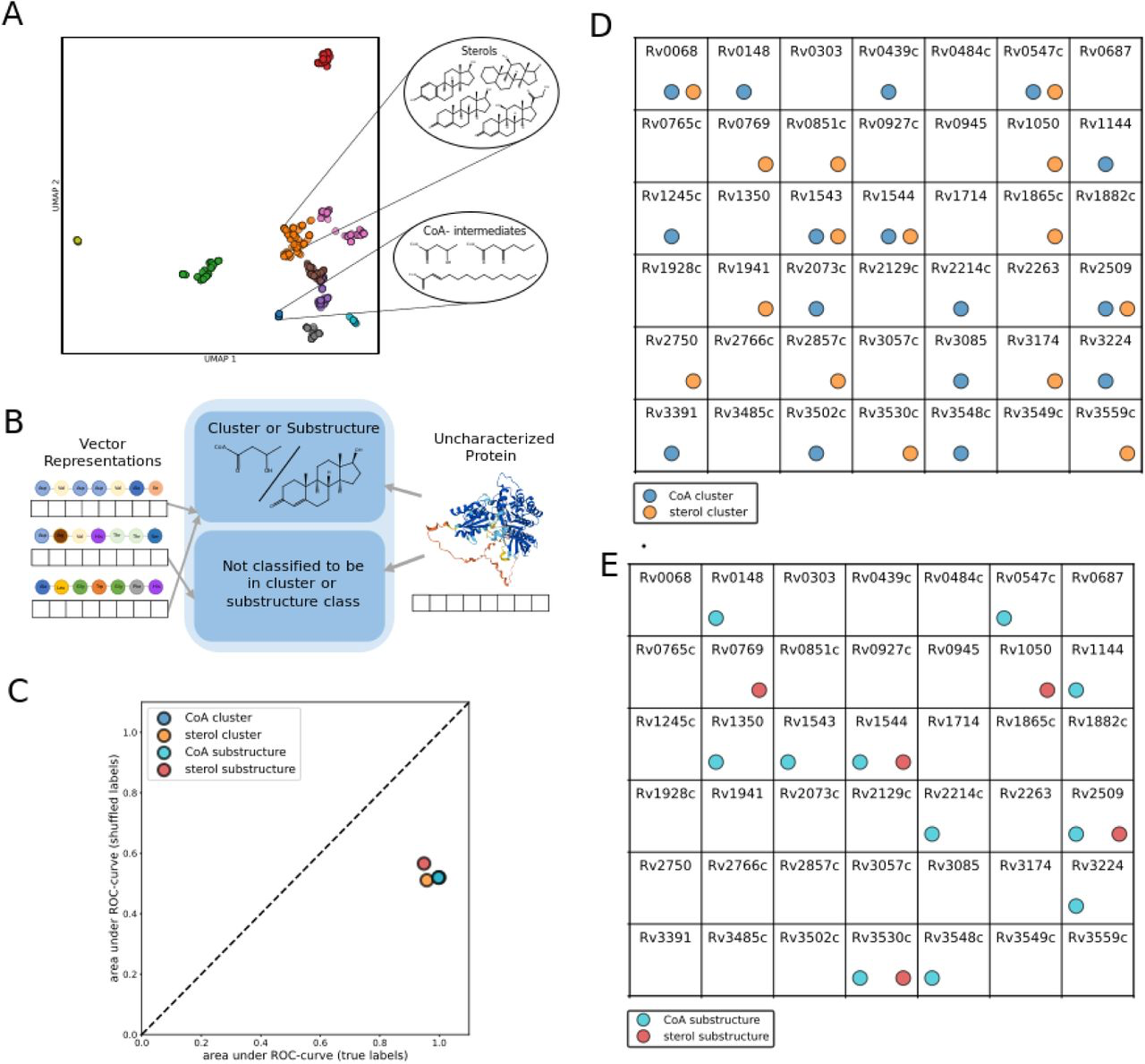
上图显示了第二个任务,通过分子聚类或子结构搜索预测SDR底物和产物的化学结构类别的结果。虽然以这种方式构建序列到底物的映射问题并不能揭示底物的确切分子结构,但它可以通过建议将模板化合物作为候选底物来帮助指导体外实验。
图A展示了使用摩根指纹对所有Rhea数据库中由被注释的SDR酶催化的反应的底物和产物进行UMAP投影和分子结构聚类的结果。每种颜色代表10类中的一个独特的k-means聚类。
本文发现,有几个聚类经检查可以解释为富含特定类型的化合物。例如,一个聚类由与酰基载体蛋白(ACP)结合的不同长度的β-酮酰基、β-羟基酰基和β-烯酰基底物组成,而另一个聚类富集了与辅酶A(如β-酮酰基-CoA)结合的同类型底物。有两个聚类富含含甾醇的化合物,另一个("聚类-6")是由维甲酸衍生物组成的。然而,有几个群组是由不同的化合物类别的更多样化的组合组成的。例如,一个包含前列腺素、鞘脂、叶绿素衍生物以及脂肪酸和三酰甘油衍生物的组合。
B图为训练和分类任务的示意图,本文为每个分子结构聚类训练了独立的二元分类器,将SDR酶的ESM-1b嵌入作为输入,并预测SDR是否能对属于该结构聚类(或包含特定化学子结构)的化合物产生催化作用。
D图和E图展示了使用基于结构聚类(D)或化学子结构(E)的标签策略,预测结核分枝杆菌中所有孤儿SDR是酶的底物的化学结构类。本文用训练好的分类器来预测,对于结核杆菌和烟曲霉蛋白组中的所有孤儿SDR酶,它们的底物属于哪个结构聚类或化学子结构标签。作者在使用两种方法(聚类与子结构)之间的共识作为预测的优先次序的衡量标准后,发现有五种酶(Rv0769、Rv1050、Rv1544、Rv2509和Rv3530c)被预测为作用于类固醇底物。此外,两种方法都预测有9种酶会对与辅酶A结合的活化底物产生催化作用。
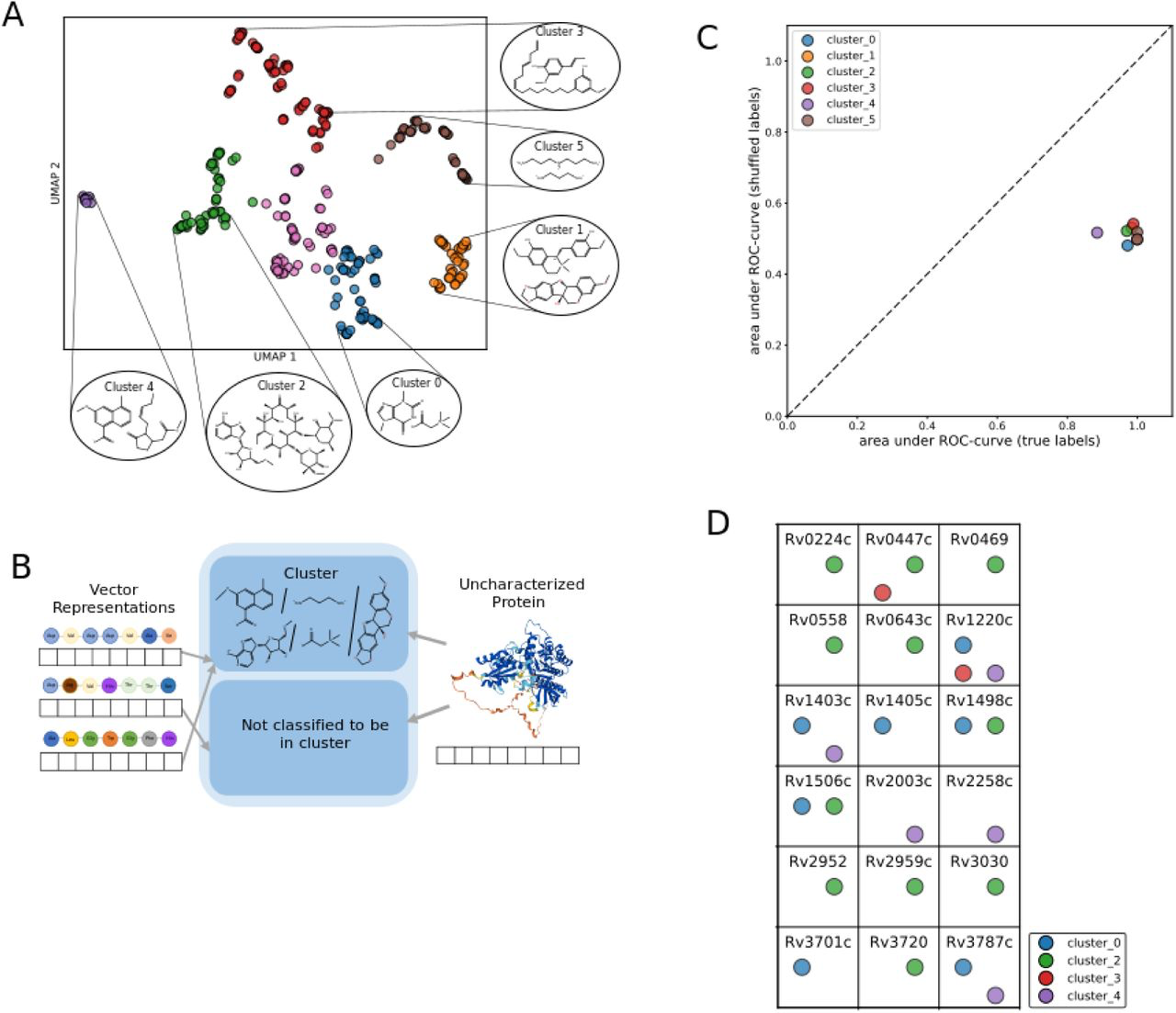
上图展示了预测孤儿SAM-MTase是否作用于小分子或聚合物(RNA、DNA或蛋白质)。
本文训练的分类器将ESM-1b嵌入作为输入,并预测一个SAM-MTase是否有属于两个不同类别的底物:小分子代谢物(包括脂类)或生物聚合物(包括蛋白质、DNA或RNA)。交叉验证的逻辑回归分类器达到了0.99的预测AUC。
随后本文用分类器来预测,对于结核病中所有被预测为作用于小分子的SAM-MT酶孤儿酶,其底物最可能的结构类别/聚类。从42个孤儿SAM-MTase中,本文的分类器能够预测18个酶的底物聚类,其中12个本文的分类器将底物缩小到一个聚类,另外6种酶,本文的分类器预测底物属于两个或多个底物聚类。本文的分类器能够预测一个酶的底物属于一个定义明确的聚类,这突出了它相对于目前的计算方法能够更详细地描述孤儿酶的功能。
创新点
- 从自监督的蛋白质语言模型中获得的预训练向量表征或嵌入,可用于一系列酶底物和辅助因子预测任务。
- 本文预测底物结构类别的方法是为每个化合物结构群训练一组独立的二元分类器,把这个任务看作是一个多标签,而不是一个多类别的分类问题。虽然也可以把这个问题看作是多类分类,但是酶通常是杂交的,有多种底物,多标签分类是一个更自然的选择。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢