【论文标题】Protein Language Model Predicts Mutation Pathogenicity and Clinical Prognosis
【作者团队】Xiangling Liu, Xinyu Yang, Linkun Ouyang, Guibing Guo, Jin Su, Ruibin Xi, Ke Yuan, Fajie Yuan
【发表时间】2022/10/03
【机 构】东北大学、格拉斯哥大学、北大、西湖大学
【论文链接】https://doi.org/10.1101/2022.09.30.510294
准确预测癌症突变的影响,有可能改善现有的治疗方法并确定新的治疗目标。本文在两个与临床相关的任务中对最先进的预训练的蛋白质语言模型进行了基准测试,即识别致病突变和预测病人生存。本文的结果显示,即使没有多重序列比对的模型的表现也可以优于基线。本文还证明,进化指数,一个基于蛋白质语言模型训练目标的分数,可以在多种癌症类型中实现统计学意义上的生存预测,这是迈向蛋白质语言模型临床效用的关键一步。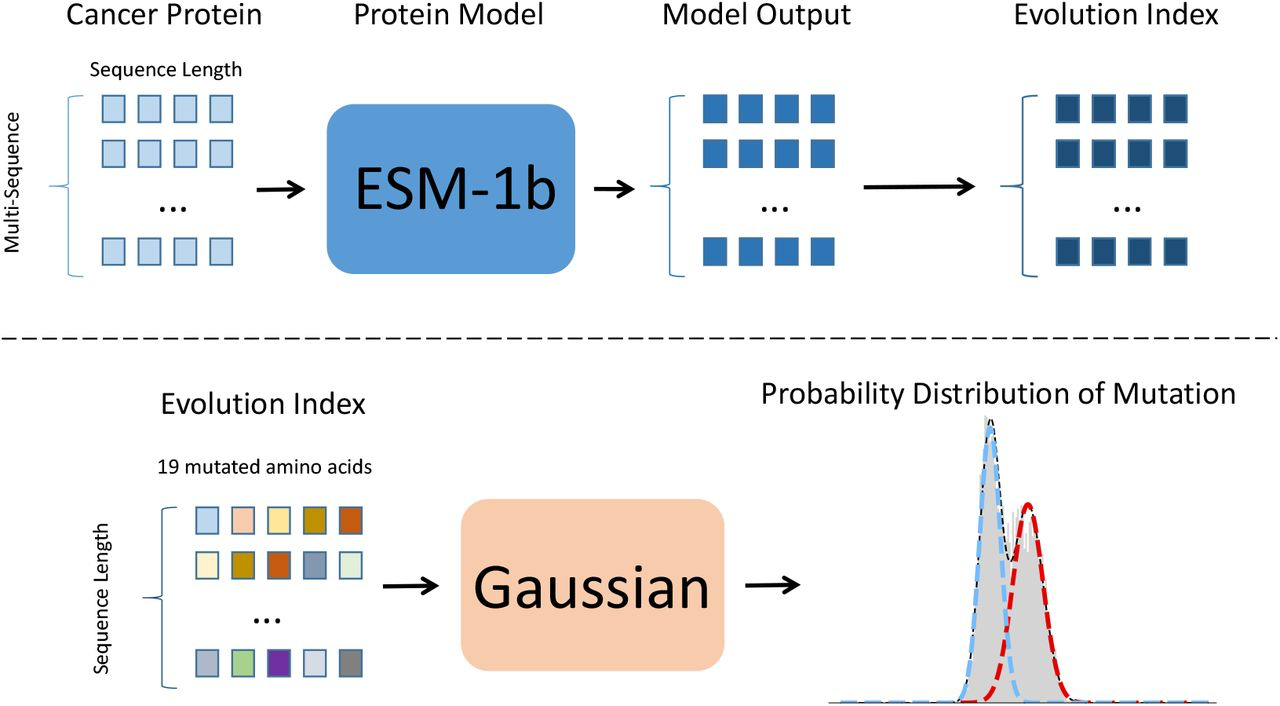
上图展示了零样本致病性预测模型的框架。
对于筛选后的ClinVar中20个常见的癌症蛋白的每一个突变,模型将把整个蛋白质序列作为输入,利用没有同源信息的预训练蛋白语言模型,输出是序列为致病性或良性的概率。这些概率是通过拟合双主成分高斯混合模型得到的分配概率,被称为进化指数的度量。进化指数(EI)是观察到突变体序列的概率与观察到野生型序列的概率之间的负对数比率。EI已被证明是反映蛋白质语言模型捕捉氨基酸序列编码信息的能力的一个直观分数,EI的值越高,表明突变体序列与野生型序列的偏差越大。因此,具有较高平均值的成分代表了较高的致病性。
ClinVar测试集上显示,ESM-1b获得的AUC为0.874,平均ACC为0.826,而ESM-1v取得了更好的结果,AUC为0.909,超过了目前报道的最好性能。两个高斯主成分之间的分离程度直接评估了ESM-1b所捕获的预测信号的强度。通过将分数与相应突变的基因组位置对齐,研究者可以进一步检查预测的生物学含义。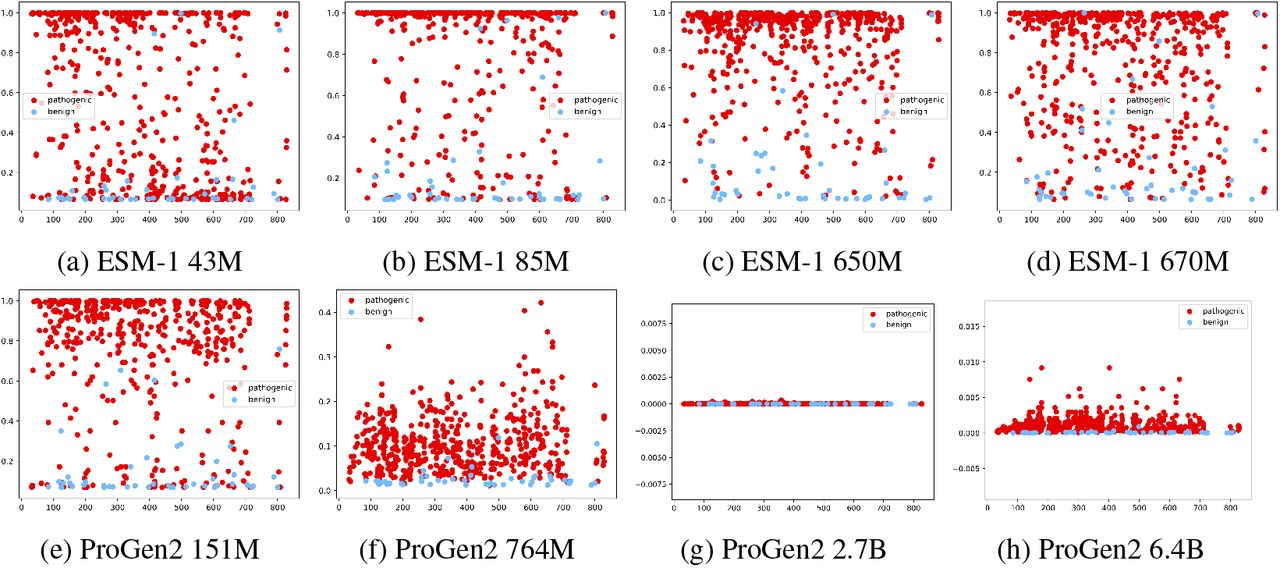
上图展示了六个大规模预训练的蛋白质模型在突变预测上的效果。
在实验结果中,本文发现在输入蛋白质序列上掩码氨基酸位点对模型的预测能力有较差的表现(没有掩码的ESM-1b: AUC=0.874;被掩码的ESM-1b。AUC=0.706)。同时,本文发现没有MSA训练的模型,如ESM-1b和ESM-1v,不比有MSA训练的模型,如EVE差,甚至更好(ESM-1v:AUC=0.909;EVE:AUC=0.887)。这一结果表明,从大规模蛋白质数据库中学习到的生物信息比从专门的同源序列中学习到的信息更丰富,其优点是计算时间少得多。同时,本文发现,训练数据集越大,模型的预测能力越强。
本文还评估了不同规模的模型之间的性能。本文发现,预训练的蛋白质模型的规模与它学习蛋白质突变致病性的能力不成正比,随着模型规模的增加,ESM-1的AUC分别为0.725、0.769、0.874和0.856。ESM-1和ProGen2的零点致病性预测性能并没有随着模型规模的增加而提高;它在764M参数时达到了一个峰值,然后逐渐下降。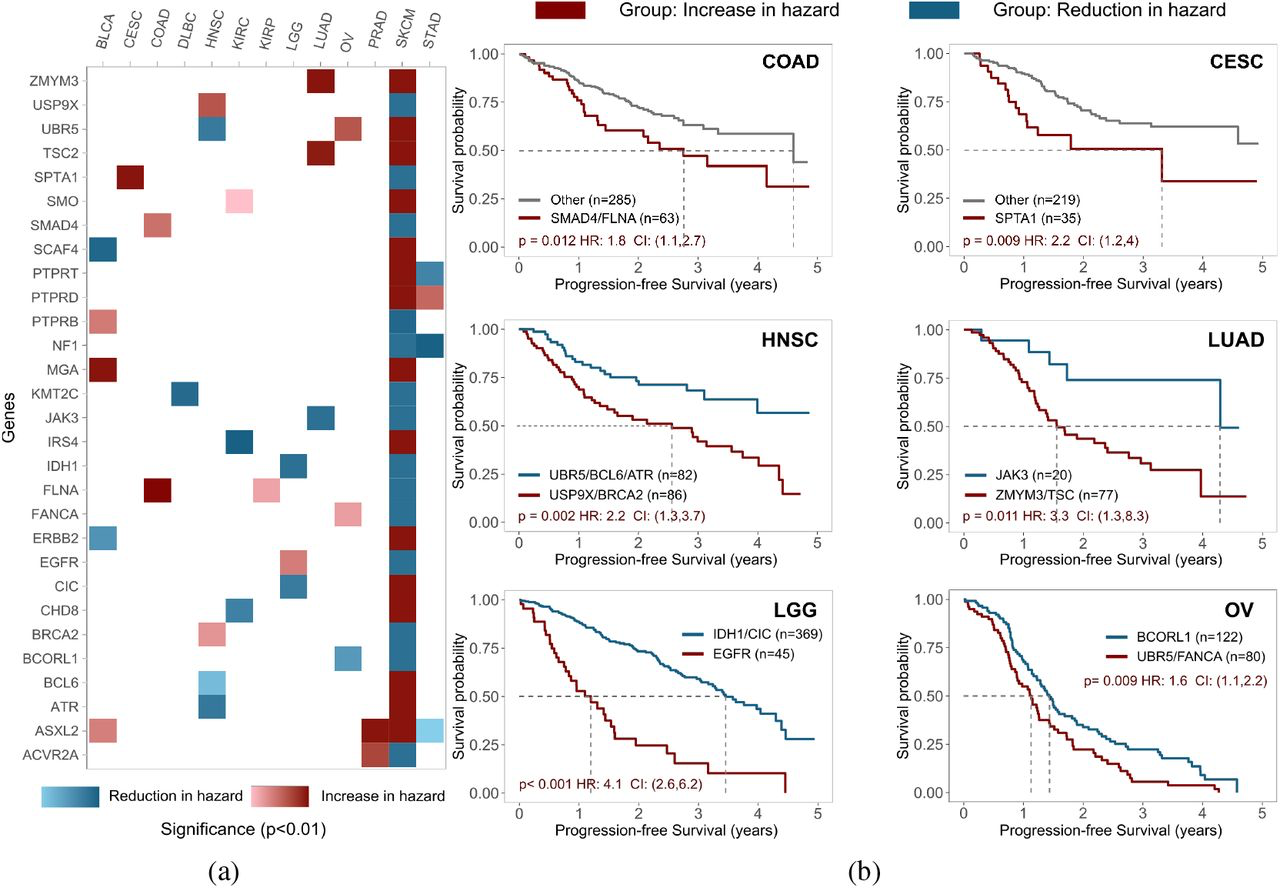
上图展示了使用ESM-1b计算的进化指数对真实世界癌症患者数据的预后预测结果。
A图展示了对不同TCGA数据库的10,248名患者的蛋白质EI进行了多变量Cox比例风险回归,按性别和年龄分层,以无进展间隔期(PFI)作为不良结果。对于本文的框架有效的13种癌症类型,本文发现特定蛋白质的高EI值明显有助于更好/更坏的生存。例如,结直肠癌(COAD)中SMAD4和FLNA蛋白的高EI值显示了患者危险风险增加的显著证据。相反,在肺癌(LUAD)中,JAK3蛋白的高EI值有助于降低危险风险。
此外,本文根据每个癌症类型的Cox回归所建议的特定蛋白的EI是否>0,将患者分层为危险增加组和危险减少组。本文绘制了Kaplan-Meier(KM)曲线,用估计的危险比(HR)和log-rank检验的p值检验两组之间的生存差异。预测的准确性用一致性指数(c-index)来评估。c-index比较了PFI预测的受试者的等级,PFI使用基于实际疾病结果的分层Cox模型进行预测。本文观察到在6种癌症类型中,危险增加组和危险减少/其他组之间存在显著差异。
这些结果表明,进化指数可以在多种癌症类型中实现具有统计学意义的生存预测。
创新点
- 本文验证了大规模预训练的蛋白质语言模型可以有效和准确地预测癌症驱动基因突变的影响。从同源序列中学习的模型和从单一序列中学习的模型都获得了可比较的结果。
- 本文提出了一个基于零样本的致病性突变预测任务的系统基准。实验结果表明,类似BERT的模型,如ESM-1b,比那些依赖生成模型的模型更适合该任务。本文还发现,预训练的蛋白质模型的大小与它预测致病性突变的性能不成正比。
- 在TCGA数据中证明了蛋白质语言模型的预后价值。预训练的蛋白质模型所捕获的致病性信息可以在六种癌症类型中区分出高风险和低风险的病人,而传统的方法还没有被证明是成功的。
参考文献
Jordan Hoffmann, Sebastian Borgeaud, Arthur Mensch, Elena Buchatskaya, Trevor Cai, Eliza Rutherford, Diego de Las Casas, Lisa Anne Hendricks, Johannes Welbl, Aidan Clark, et al. Training compute-optimal large language models. arXiv preprint arxiv:2203.15556, 2022
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢