【论文标题】Incorporating Pre-training Paradigm for Antibody Sequence-Structure Co-design
【作者团队】Kaiyuan Gao, Lijun Wu, Jinhua Zhu, Tianbo Peng, Yingce Xia, Liang He, Shufang Xie, Tao Qin, Haiguang Liu, Kun He, Tie-Yan Liu
【发表时间】2022/11/17
【机 构】华中科大、微软、中科院、北大
【论文链接】https://doi.org/10.1101/2022.11.14.516404
抗体是一种多功能的蛋白质,可以与病原体结合并为人体提供有效的保护。最近,基于深度学习的计算抗体设计引起了人们的关注,它可以从数据中自动挖掘出抗体信息并与人类经验互补。然而,这些计算方法严重依赖高质量的抗体结构数据,而这些数据是相当有限的。此外,互补决定区(CDR)是抗体中决定特异性和结合亲和力的关键部分,它是高度可变的很难预测。因此,数据限制问题进一步提高了抗体生成的难度。对此,大量的抗体序列数据可以帮助建立预训练模型并减轻对结构数据的依赖。本文中将预训练范式纳入抗原特异性抗体设计模型,基于序列数据预训练的抗体语言模型,以此进行基于表位的抗体序列和结构一次性生成,以避免来自自回归方式的影响。本文的方法在不同的任务上都取得了比基线更好的性能。
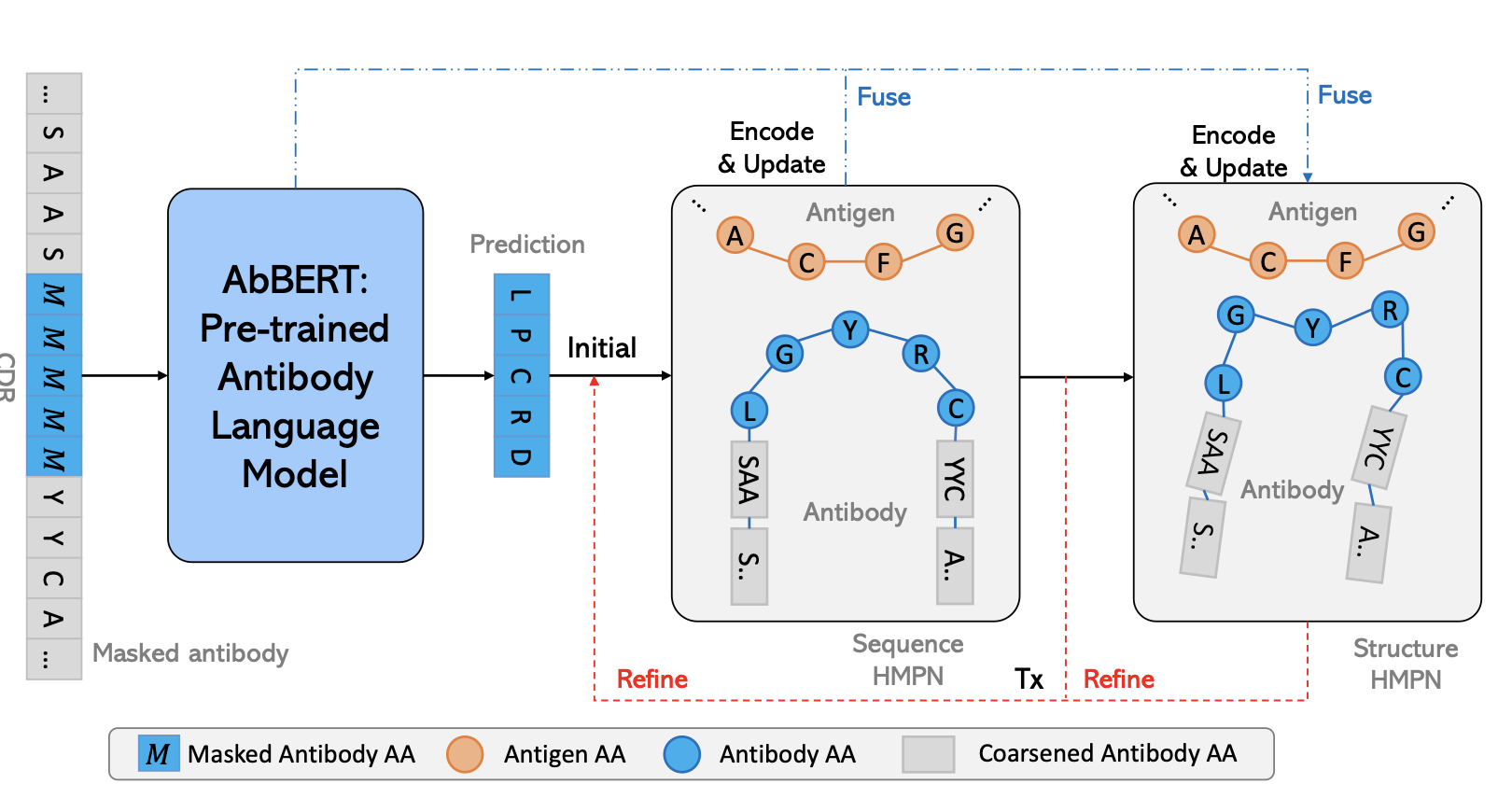
上图展示了本文方法的整体框架。AbBERT是预训练抗体模型。它的 "软 "预测将被送入序列HMPN H_seq,在编码和生成更新的序列后,结构HMPN H_str编码更新的图,然后预测结构,序列和结构
预测迭代优化了T次。
本文将抗体设计设定为先生成序列,再进行结构预测的过程。通过分层图神经网络对残基和原子表示进行分层编码后,可以生成CDR的氨基酸并预测结构。这里的序列解码采用的是one-shot的方式,而不是自回归方式。自回归生成有两个问题。一个是解码效率问题。分步解码的速度很慢,需要n次(长度)解码,而one-shot解码只需要一次。另一个不好的地方是错误积累问题。在生成步骤中,上一个解码步骤的错误会传播到当前的解码步骤中,这将导致错误的扩大,从而导致错误的预测,特别是在后面的步骤中。另外本文的one-shot方式避免了错误积累的问题,但它不能保证同时生成的残基的准确性。因此本文为one-shot预测的残基和结构增加了T个优化步骤来提高性能,由于T比n小得多故效率得到了保证。
one-shot方式的解码方式为,采用多次迭代的编码隐表征通过Hseq进行氨基酸多分类预测,本文在第一个和第t-1个细化步骤之间使用 "软 "预测进行H_str编码,只有最后一个步骤T将输出一个“硬”预测,从确定特定氨基酸的概率中采样。
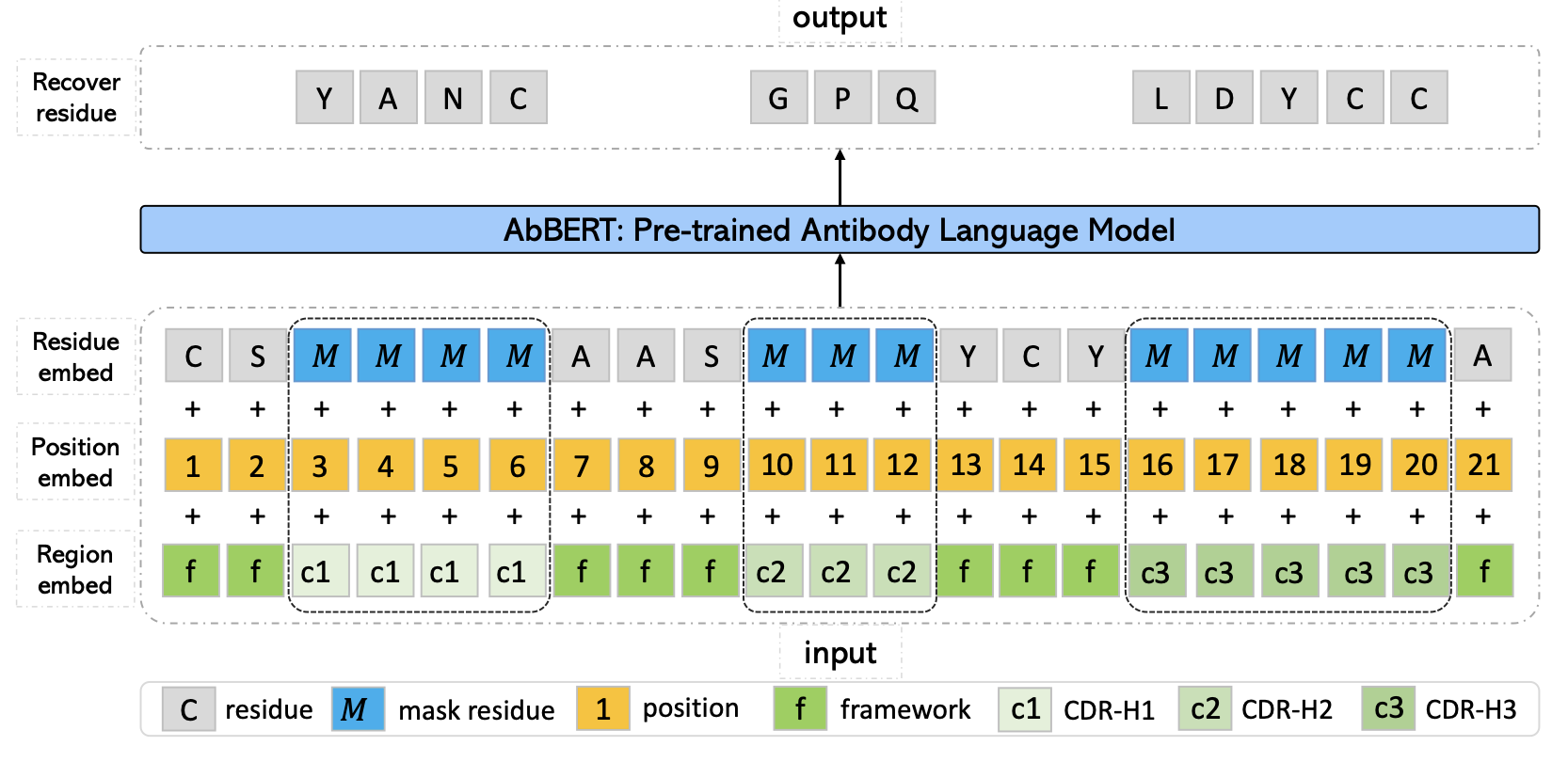
上图展示了AbBERT的输入形式,掩码对CDR进行操作,另外专门对框架区和可变区相应地设置单独的区域嵌入作为输入。
本文通过两种方法来结合预训练的AbBERT进行抗体设计。首先,预训练的AbBERT为H_seq和H_str提供初始化,即从AbBERT中给出一个“软”概率。其次,对于Hseq和Hstr,本文将来自AbBERT的隐表征与来自编码器的MPN的残基表征在最后的预测器前进行融合。
另外本文也比较了不同的微调方法,包括固定不微调(作为特征提取器),对预训练模型的所有参数进行微调,以及前缀微调,即在固定原始模型的同时只对添加的前缀进行微调。结果显示本文采用的采用的前缀微调比其他两种微调表现得更好。这符合期望,因为结构抗体数据的规模有限,而且前缀调整法在参数调整和知识转移之间取得了最好的平衡,全微调容易过拟合,固定不微调难以迁移到共设计中。
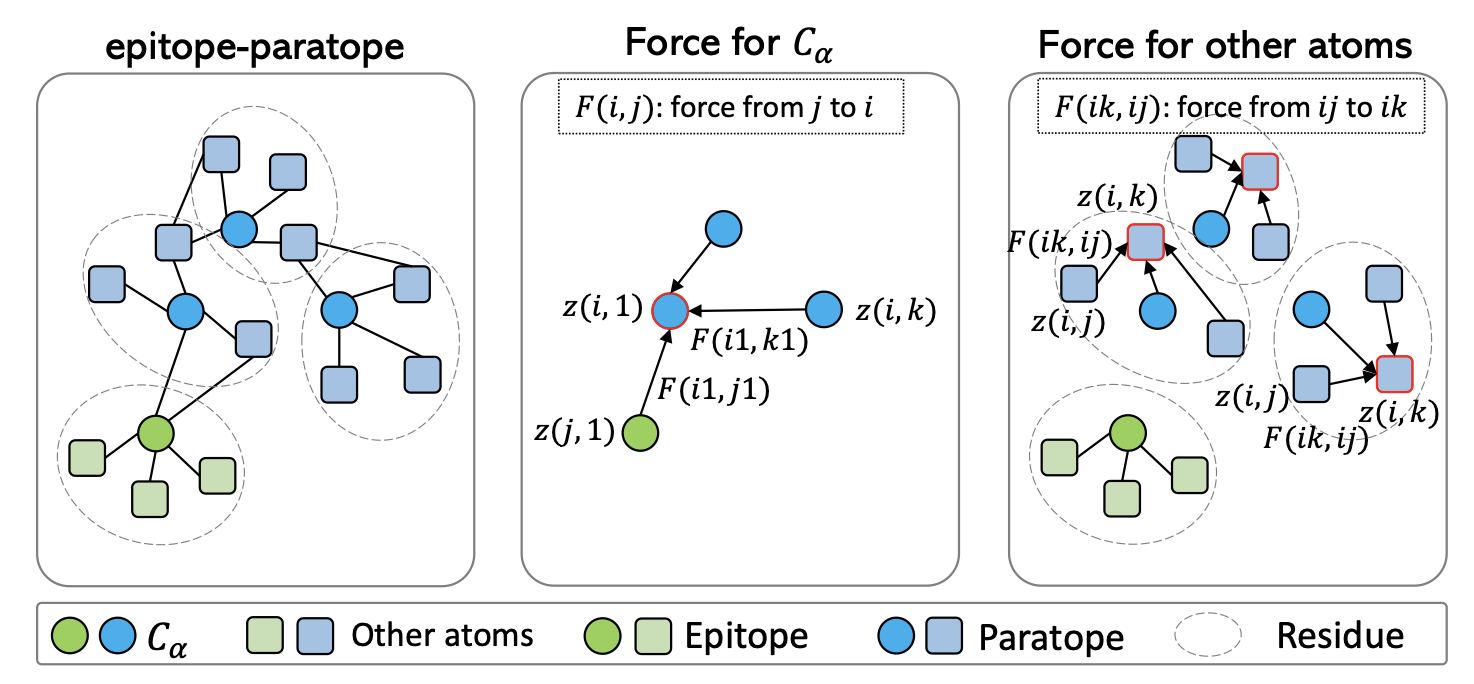
本文在抗体结构解码上的关键任务为上图展示的力场计算。本文使用力场预测而非坐标预测的原因是结构更新必须保持与表位的旋转和平移的等变性。力通过残基和原子的隐表征进行计算,Cα原子和其他原子之间的力被分别单独计算,最近的Cα原子之间的作用力被计算出来以更新近邻的Cα坐标。对于其他原子,它们的坐标将根据同一残基中的原子之间计算的力进行更新。
创新点
(1) 背景信息。对于抗原本文只考虑表位而不是整个抗原,对于抗体本文除了CDRs之外还增加了框架区。
(2) 图的初始化。本文使用本文的AbBERT输出作为残基初始化,而不是随机猜测。
(3) 解码策略,与低效的自回归生成方式不同,本文的one-shot解码大大加快了推理过程,并具有更高的精度。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢