本文节选自DrugAI
AlphaFold2的巨大成功有望变革结构生物学,然而对于蛋白质的研究,结构仅仅只是开始,对于蛋白质功能的解读,例如,未知蛋白质功能注释,突变影响,蛋白质工程,折叠的稳定性等研究更加具备实际意义。近日,西湖大学人工智能团队最新论文对AlphaFold2的功能研究给出了一些有意思的发现,相关论文发表在人工智能顶级会议NeurIPS2022。
论文题目
Exploring evolution-aware & -free protein language models as protein function predictors
代码数据集
https://github.com/elttaes/Revisiting-PLMs
论文地址
https://arxiv.org/pdf/2206.06583v2.pdf
随着深度学习领域的发展,大规模蛋白质语言模型(PLM)在蛋白质预测任务中的取得了很大的成绩,比如蛋白质3D结构预测和各种功能预测。特别的,AlphaFold2,一个 具有革命性的人工智能蛋白质模型,在CASP14蛋白质结构预测任务上取得了原子级别的预测准确度,该成果可能会重塑结构生物学。
然而,除了结构预测之外,还没有相关研究对于AlphaFold的核心组件Evoformer(一种新颖的PLM)进行结构以外任务的评估检验。在本文中研究者们聚焦Evoformer的表征能力研究,尤其是探究其蛋白质功能预测水平。
作者的主要动机是:既然蛋白质结构决定功能,而AlphaFold2如此精准的结构预测能力是否也具有较强的功能预测能力。针对此,研究者探究了三种流行的PLM的表征能力:ESM-1b(单序列PLM)、MSA-Transformer(多序列比对PLM)和 Evoformer(基于结构的PLM),尤其关注Evoformer的能力。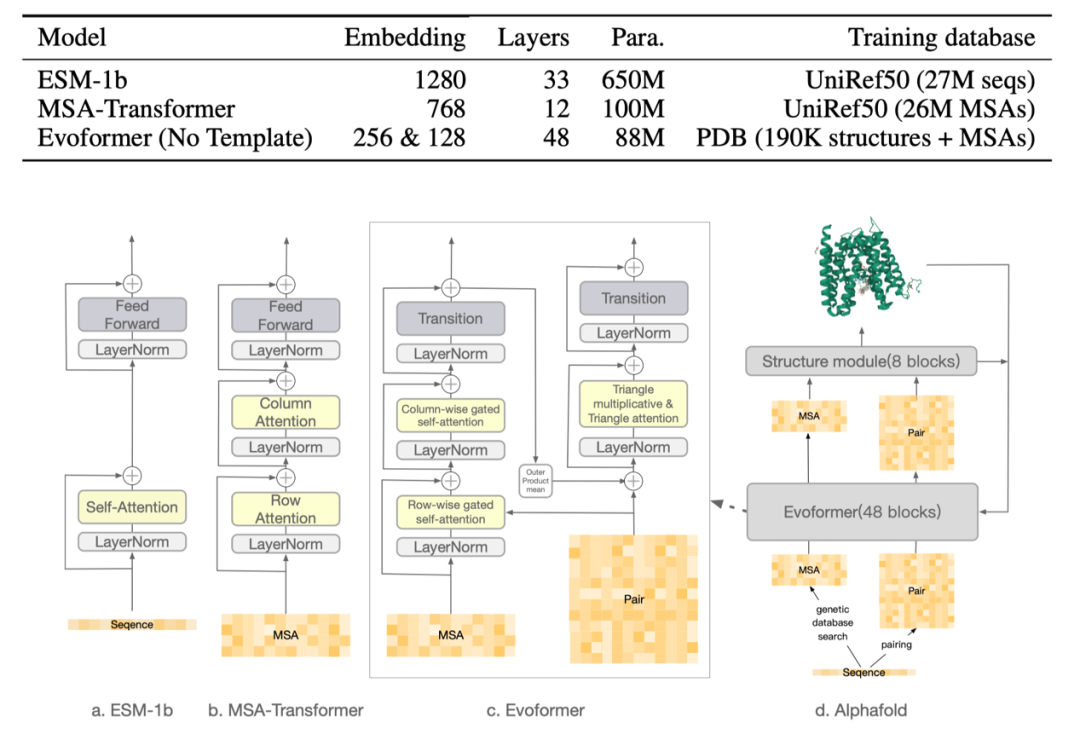
图1:三种主流蛋白质语言模型神经网络对比
蛋白质是生命活动的主要执行者,因此,破译其结构和功能特性背后的机制至关重要。其中蛋白质序列-结构-功能关系使得基于序列的机器学习方法在结构和功能预测上取得很大成功,这些方法可以从氨基酸 (AA) 序列推断蛋白质结构和功能。亿级别参数的大规模的蛋白质语言模型通过自监督学习方法已经成为AI预测蛋白质功能的最主流做法。
通过对各种蛋白质相关任务的实验,我们得到以下结论:
(1) AlphaFold2训练的Evoformer能够得到对结构和功能预测都有用的表征。
(2) Evoformer产生的表征对于蛋白质或残基级别预测任务都很有用。
(3) Evoformer在结构预测和蛋白稳定性预测方面优于ESM-1b和MSA-Transformer,但在其他功能预测任务上不如ESM-1b。
(4)与ESM-1b和MSA-Transformer相比, Evoformer在零样本突变任务上表现很差。
(5) 基于进化信息的蛋白质语言模型仅在结构预测任务中优于无进化信息的ESM-1b模型,在大多数功能预测任务中通常比ESM-1b差。
(6) 与结构预测类似,基于进化信息的蛋白质语言模型在预测蛋白质功能时也对MSA的数量比较敏感。
本文主要观点是通过大量的实验证实:AlphaFold2虽然在蛋白质结构预测任务上取得了革命性的成果,但其核心表征学习组件Evoformer并不能直接带来蛋白质功能预测的显著提升,更不可能替代已有的SOTA, 也就是Facebook研发的 ESM1b与MSA-Transformer。
更多阅读
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢