不同于往届将蛋白质结构预测作为主要赛道,CASP 15更侧重预测蛋白质复合体结构(原来CAPRI比赛的内容)和RNA结构(原来RNA-Puzzles比赛的内容)。在这届的RNA结构预测中,过去蛋白质结构预测的很多老牌强队,如密苏里大学陈世杰组、RNApolis,David Baker组、密歇根大学张阳组,山东大学杨建益组,也逐渐参与到这些新兴赛道中。
近日,第15届蛋白质结构预测比赛(CASP 15)正式落下帷幕。从CASP网站上已公布的六个赛道排名上看,华人团队表现亮眼,在蛋白质单体/多体结构预测、蛋白质-蛋白质复合体结构预测、RNA结构预测、蛋白质-小分子配体复合物结构预测(得分有异议,最终结果尚未公示)等4个赛道上名列前二。其中成立于2021年4月的智峪生科,更是在后两大赛道中分别取得冠亚军两项荣誉。
值得注意的是,不同于往届将蛋白质结构预测作为主要赛道,CASP 15更侧重预测蛋白质复合体结构(原来CAPRI比赛的内容)和RNA结构(原来RNA-Puzzles比赛的内容)。
与此同时,由于前一届AlphaFold2和RossetaFold在蛋白质三级结构预测中取得重大突破,与之相关的细化(Refinement)、接触预测(Contact and Distance prediction,二级结构预测相关)、模型准确性估计(Domain-level estimates of model accuracy)均被删除。
据悉,今年5月CASP 15赛程伊始,CASP比赛发起人John Moult教授曾对外表示,“蛋白质预测上的成就,以及开源工具的不断丰富,都为解决RNA折叠问题提供了不少帮助。此外,蛋白质通过与其他蛋白质和分子相互作用而变得独特,因此我认为这(预测蛋白质复合物结构)比预测单个蛋白质结构更重要。这是在各种行业应用,尤其在药物设计中需要解决的基础性问题。”
今年的参赛队伍多于往届,并且有较多的国内组织参与。据CASP 15会议官方统计,全球共计163个计算生物学家小组参加了比赛。
其中,来自中国的参赛队伍数量基本与美国持平,包含华深智药的Omegafold、清华产业研究院AIRfold、百度飞桨的HelixFold、深势科技的Uni-Fold、上海天壤的TRFold、分子之心的许锦波教授团队、以及清华、北大、人大、山大、浙工大、江苏理工、上海科大、西湖大学、中科院计算所等诸多业界和高校团队。
因此,随着CASP 15在2022年寒冬打响赛道变革第一枪,生命科学的春天已经悄然来临。
CASP 15转变之年
本届赛程从2022年5月初持续到8月中旬,包含蛋白质单体/多体结构预测、蛋白质复合体结构预测、RNA结构预测、蛋白质-小分子配体复合物结构预测、蛋白质构象集合、准确性估计等六个赛道。
对此,雷峰网&《医健AI掘金志》主要对蛋白质单体结构预测、RNA结构预测两大赛道展开论述。
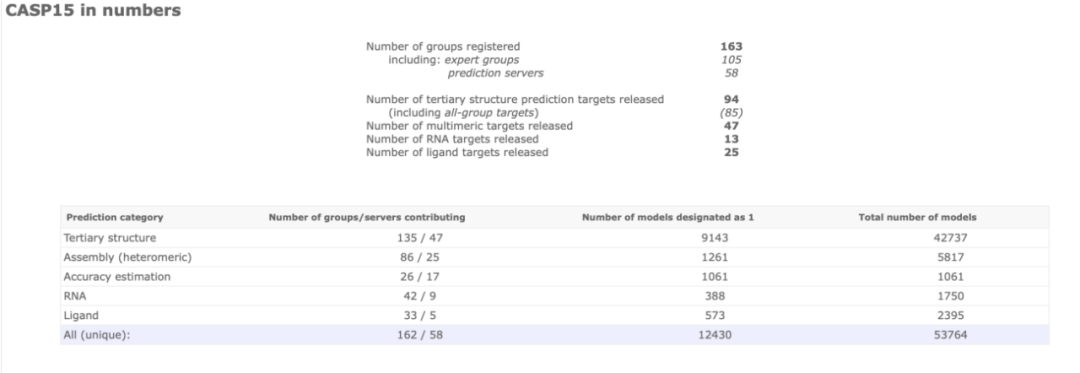
CASP15中的靶标体系和不同赛道参赛情况
注意:数据有重合,会出现一个小组参加多条赛道的情况
在蛋白质单体结构预测方面,多方法组合优化仍是“必杀技”。
在本届竞赛中,蛋白质单体结构预测赛道共有135个小组参加,共预测了94个蛋白单体靶标体系。以CASP网站的排名来看,山东大学杨建益为首的Yang-Server服务器、郑伟博士开发的UM-TBM服务器(基于D-I-TASSER算法)、日本Infinite Curation公司小田贤幸的人工组PEZYFoldings分列前三,并与后续算法梯队拉开距离。
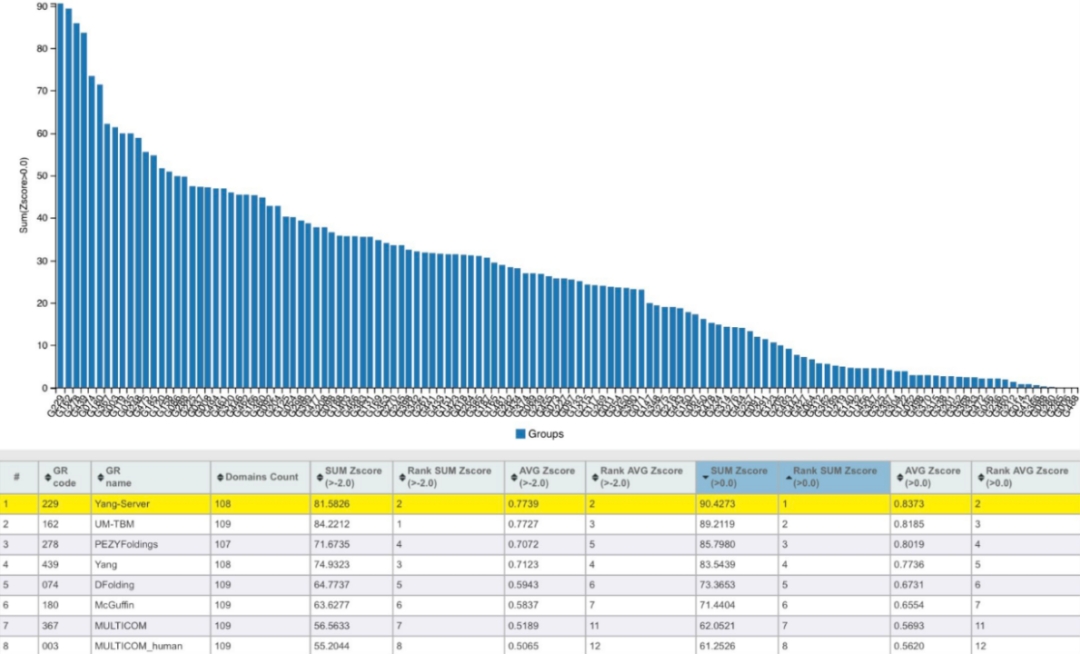
据悉,夺得榜首的杨建益教授,曾就职于南开大学等高校,2021年7月加入山东大学数学与交叉科学研究中心,研究方向包括蛋白质结构与功能预测、RNA结构预测等,此前曾与张阳、David Baker等学者合作,成功开发了I-TASSER和trRosetta等著名结构预测算法。
此次杨建益团队带来的Yang-Server方法,是在以前trRosettaX的基础上,改进了氨基酸距离和角度的预测,并用trRosetta 的能量函数进行折叠。而对于trRosetta预测不好的结构,则直接使用了AlphaFold2的预测结果。

杨建益
实际上,通过组合使用AlphaFold2或其他方法而取胜并非个例。排名前三的UM-TBM服务器,以及人工组PEZYFoldings,在方法设计或工艺优化上均看到了AlphaFold 2的影子。
其中,UM-TBM是对密歇根大学张阳课题组以前所建立的多个工具的使用和改进,再通过AlphaFold 2以及其他深度学习方法产生的结构约束,和AlphaFold 2产生的多个结构一起进行重新折叠和优化。
PEZYFoldings则是AlphaFold2的变体。首先它通过利用不同搜索工具以及不同蛋白质序列数据库来产生不同的同源序列组合,其次将这些不同的同源序列组合分别输入AlphaFold2进行预测,再根据AlphaFold2内部的PLDDT将预测出来的多个结构进行排名以及结构比对,从而得出最后的人为挑选。
遗憾的是,今年没能够看到“Alphafold3”的参赛,不过Deepmind一向已挑战科学难题为己任,难道它们认为蛋白质单体结构预测问题已经解决?
对此,深圳湾实验室周耀旗教授表示,“虽然AlphaFold 2没有直接参加比赛,但从上面三个最佳预测方法的分析可以看出, CASP15还是由AlphaFold2隐形取胜。”

周耀旗教授
值得注意的是,在近日CASP 15总结会议上,领导AlphaFold团队的John Jumper博士也线上现身,并透露出一个重要信号:DeepMind 即将发布利用更大数据库训练的AlphaFold3。”
周耀旗教授表示,“从技术层面考虑,或许AlphaFold3会再一次抛开其他方法,但也不会抛得太远,因为方法本质不会变化太多。”
密苏里大学许东教授补充道,“但至少证明,谷歌后续将加快商业化进程。因为现在很多人关心的不光是结构预测,还有结构预测到底能干什么。近日注意到,Isomorphic(谷歌母公司Alphabet下的AI制药公司)聘请了多位有制药背景以及机器学习的高管和员工,还将公司总部从伦敦扩展到瑞士洛桑,此前这里有多家大型制药公司,比如罗氏、诺华和拜耳。”

许东教授
实际上,自2021年7月AlphaFold蛋白质结构数据库开源以来,其中的蛋白质组数据已达48种,包含人类,老鼠,果蝇,玉米、亚洲稻种、大豆及酵母等重要作物,大肠杆菌与白色念珠菌等病原体,以及多种罕见热带疾病的生物蛋白质组等。2022年1月28日,DeepMind团队宣布“希望此举可以加快疾病研究进程”。弦外之音直指Isomorphic Laboratories。
此外,在12月13日CASP 15落幕当天,《Nature》发文“After AlphaFold: protein-folding contest seeks next big breakthrough”,提到了Deepmind缺席原因或要“憋大招”---目前Deepmind正在开发用”语言模型”预测蛋白质结构的方法(其中包含一个由社交网络巨头Meta开发的方法),可能有助于预测突变如何改变蛋白质的结构。
如今看来,站在计算生物学和药物设计的风口,谷歌或将再次“大展拳脚”。
RNA结构预测首次加入赛道,然而AI的光辉还没有照亮RNA结构预测。
RNA结构预测引起了研究小组的广泛兴趣。其中,共有42个小组参加了RNA三维结构预测,共预测了12个靶标体系。
实际上,长期以来,由于新的、即将解析的RNA结构太少、导致RNA结构研究一直间歇性开展。周耀旗教授表示,“以前RNA-Puzzles是有一个RNA就让大家预测一个,这次CASP 15一下子放出来12个RNA序列(从结构上可以分成三类:天然的RNA、人工设计的RNA、蛋白质和RNA相互作用的复合结构),充分体现了CASP组织者的能力和AlphaFold2给CASP带来的品牌效应。”
不过,周教授也提到,“这一打的RNA序列和有100多个蛋白结构域的预测比赛相比,还是差得很远。”
从目前CASP网站公布排名来看,前两名都为华人团队,分别为智峪生科的Alchemy RNA2、密苏里大学陈世杰组的Chen(综合他们组过去所开发的Vfold3D,IsRNA,RNAJP三个方法)。
值得注意的是,此次排名靠前的团队,采用的都是基于能量函数的传统方法,而使用AI深度学习进行端到端的预测,或者先进行AI结构约束的预测再进行能量优化的团队,反而因为RNA已知结构数据过少导致训练过度,在RNA结构预测上并没有体现出AI的优越性。
对此,周耀旗教授做了一个形象的描述:“AI的光辉还没有照亮RNA结构预测。”
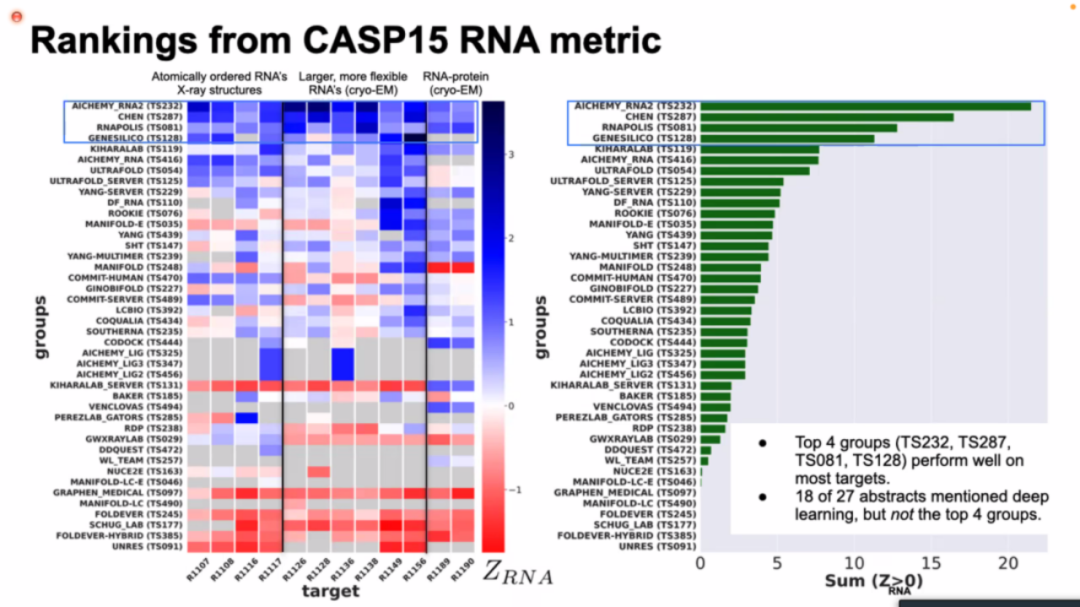
RNA结构预测方法的排名
为RNA三维结构预测的新手,智峪生科为何能够一举夺冠?
据智峪生科介绍,Alchemy_RNA2是由CTO熊鹏博士所带领打造,并由智峪生科CEO王晟博士担任“幕后参谋”。
王晟是CASP的老选手,从2008年的CASP 8作为观察员参赛以来,至今已参加8届,并在CASP 12/14中两获蛋白接触图预测第一名。
熊鹏曾在澳大利亚格里菲斯大学从事博士后和研究员工作,师从周耀旗教授,2020年4月回国,后以访问学者身份进入深圳湾实验室周耀旗课题组,继续从事蛋白质/RNA的序列、结构与功能关系方面的基础研究。
作为智峪生科的联合创始人之一,此次夺冠的AIchemy_RNA2方法的核心,正是基于熊鹏在周耀旗课题组工作时所主导开发的RNA-BRiQ统计能量函数。

熊鹏、王晟
王晟博士对雷峰网&《医健AI掘金志》说到,“RNA-BRiQ相互作用不再由简单的距离和角度来表示,而是考虑了RNA的相关原子在三维结构上的电子云分布,通过6个纬度的统计来刻画,在每个维度上将空间进行离散化进行精确的统计和量化计算。该能量函数在RNA结构预测上对极性相互作用有着很好的表征能力,因为稳定RNA结构的主体能量是极性相互作用、有着强烈的空间方向分布。RNA-BriQ这种统计能量函数方法,对于人工合成的RNA,或从未出现在PDB数据库中的天然RNA结构的建模,有着非常优异的效果。”
但仅仅是沿用传统方法还不够,AI能否用于RNA结构预测?王晟博士进一步说到,“本届比赛智峪生科共有两支队伍参加RNA结构预测,一支是熊鹏博士带队的Alchemy_RNA2,关注统计能量函数;一支是沈涛所带队的Alchemy RNA,联合港中文李煜教授和复旦大学孙思琪教授团队,共同研发了AI预测方法。该方法是全球第一款端到端RNA三维结构预测的深度学习模型。”
相比于其他团队(例如David Baker组,张阳组,和杨建益组)的AI结构预测模型,智峪生科团队搭建的Alchemy RNA有如下一些特点:
首先,和蛋白质预测不同,RNA领域里的结构数据非常稀少。去除冗余之后,我们只有1000个左右的RNA结构数据。Alchemy RNA利用了一个预训练的语言模型,RNA foundation model (RNA-FM),它在2300万个非冗余RNA序列上用自监督的方式进行训练,学习到了丰富的RNA序列信息。Alchemy RNA利用RNA-FM得到的RNA序列表征来送入模型,其中在大量数据库中学习到的RNA序列表征能够帮助模型快速收敛。
其次,Alchemy RNA通过引入多任务训练来运用更多的先验知识来帮助模型学习到更多的生物学语义。它将RNA中特有的二级结构碱基互补配对信息以损失函数的方式加入进来使模型在优化时学到碱基配对的约束信息。
最后,Alchemy RNA通过自蒸馏的训练方式提前在真实结构数据上训练好一个教师模型用来生成自蒸馏数据的伪标签,Alchemy RNA额外从RNAStralign和bp-RNA-1m数据库中构建了一个自蒸馏数据。
这些创新使得Alchemy RNA在有限的RNA结构数据上训练出了一个高效准确的深度学习模型,它能够端到端的预测RNA 3D结构。在CASP15比赛中,Alchemy RNA作为全自动的端到端预测方法,在所有的AI预测方法中名列第一位。
更重要的是,Alchemy RNA还可以全自动地判断输出结构的预测置信度。也就是说它可以自动判断它输出的结构是否是合理的,是不是还需要人工检查。这个功能非常实用,可以帮助我们高效地融合专家知识。
目前智峪生科在CASP中所展示出来的AI方法,由于主要采取的是多序列匹配MSA作为输入,因此对于这12个测试结构中的天然RNA,是可以搜索到同源序列并产生多序列匹配的,进而可以使用我们的AI方法进行预测。
而对于那些人工合成的RNA,或从未出现在PDB数据库中的天然RNA结构,虽然目前无法产生多序列联配,但是Alchemy RNA就会基于预测置信度来提示--对于这些RNA则需要进一步处理。这样研究人员便可以借助统计能量函数方法或其它人工介入的手段进行操作。
王晟博士表示,“上述的这种基于预测置信度的做法是一种简单的融合AI与统计能量函数的手段。值得一提的是,目前由于RNA的结构数量过少以及MSA信息的依赖,纯AI方法还没有能够实现针对任意序列的RNA结构的精确预测,还处于一个前AlphaFold2时代。但在未来,随着我们积累越来越多的RNA结构数据、训练更强大的RNA序列基础模型、以及更深入的进行AI方法与统计能量函数的融合,我相信一定会让整个RNA结构预测领域,进入真正的AlphaFold2时代。”
生命科学领域或合成生物学领域,将迎来哪些变化?
CASP比赛已近30年,一直以与时俱进著称,因此本届比赛被诸多圈内人视为“后AlphaFold2时代”首届比赛。
换言之,蛋白结构预测取得突破性进展后,单体结构预测将进入一个新的渐进发展阶段,但计算结构生物学领域中RNA结构的预测问题,以及复合体建模的问题(包括了蛋白质和其他生物分子,如蛋白质,核酸以及小分子,多糖等等),即将进入到一个广受关注的时代。
王晟博士表示,“过去蛋白质结构预测的很多老牌强队,也会逐渐参与到这些新兴赛道中,比如这一届的RNA结构预测中,就看到了David Baker组、密歇根大学张阳组,山东大学杨建益组等熟悉面孔的加入。”
新老团队的流向再次说明:RNA结构预测水平的进步,因为其在药物设计和合成生物学方向的落地应用而率先脱颖而出。
那么,RNA结构预测水平的进步,生命科学领域或合成生物学领域而言有何意义?
具体来看,本次比赛一共有12个RNA靶标体系,在功能上可以分为四类:功能RNA分子(ribozyme,riboswitch)、病毒基因组RNA片段、人工设计的RNA分子,以及蛋白RNA复合物。
功能RNA分子在基因表达调控方面起重要作用;病毒基因组RNA对于我们理解病毒复制传播机理、开发抗病毒药物重要帮助;人工设计RNA分子有助于合成生物学应用;蛋白RNA复合物的应用方向是药物开发。
那么无论是此次在RNA结构预测领域夺冠的智峪生科,还是其他团队的加入,都证明实现高精度RNA结构预测,必将为非编码RNA功能的发现及解释,RNA药物的设计与开发带来深远的影响。
针对这个问题,王晟博士做了详细介绍,认为目前在RNA结构预测领域的研究,有利于解析更多的RNA结构、设计出结构更稳定的RNA序列、成为关键药物靶点、以及赋能合成生物学领域。
首先,它将有助于研究人员解析更多的RNA结构。尽管Alchemy RNA在大多数天然RNA家族和RNA类型上的预测精度令人印象深刻、以及Alchemy RNA2在人工设计的RNA结构上的惊人表现,但考虑到RNA的多样性及其灵活的结构,人们还是应该用实验的方法去解析更多的RNA结构,从而极大的扩充PDB种的RNA结构数量。因此,智峪生科开发RNA预测方法,有可能成为指导实验设计和数据处理的初始模型。
其次,针对药物设计领域,例如在mRNA疫苗的设计上,需要关注设计的RNA序列的结构稳定性,这样就可以提高疫苗设计的成功率。此外,基于RNA的小分子药物设计方向,RNA分子也逐渐可能成为将来可以成药的靶点,也依赖于高精度的RNA 3D结构预测,和高精度的RNA-小分子复合体力场。同时,RNA和蛋白质复合体也可能成为关键的药物靶点,针对复合体界面的药物设计可以极大拓展药物设计的空间。
除了药物设计领域,高精度RNA预测也可以赋能合成生物学领域。例如,如何构建高精度的密码子优化算法,非常依赖于高精度的RNA结构预测,尤其是从RNA binding site到蛋白质前面一些氨基酸对应的mRNA的结构,会影响到到蛋白质的高效可溶表达。另外,RNA分子也可能成为非常有潜力的生物传感器,对小分子的检测可能做到非常高的灵敏度,这个方面的应用也是非常依赖于高精度的RNA结构预测。
据王晟博士介绍,智峪生科已自主打造了完整蛋白质结构预测、设计、生产体系,以此开展药物辅助研发及合成生物学业务。那么基于蛋白质/RNA结构预测,已经有了落地打算,“接下来将主要应用在合成生物学的关键元件的发现和设计中,包括一些关键的生物传感器的设计,用于检测酶催化的产物或者底物等,也可以用于密码子的优化以帮助蛋白质高效可溶表达。”
CASP比赛将走向何处?
实际上,此次比赛中也有不少圈内人格外关注复合物结构预测问题。也就是各种生物大分子之间,例如蛋白质,核酸,多糖,脂类等等,生物大分子-小分子之间的相互作用模式问题。
诸多圈内人对雷峰网&《医健AI掘金志》表示,“这一定会成为下一届,或者以后CASP比赛的主流方向。”
江苏理工学院生物信息与医药工程研究所所长,普美瑞生物首席科学顾问常珊博士,作为受邀CASP 15总结会议的团队负责人表示:
从技术落地的角度来说,今年新开赛道都非常有必要取得突破。RNA结构预测与最近的RNA疗法等密切相关、蛋白-ligand相互作用预测是药物设计和筛选的基础、复合物组装对抗体开发和蛋白质降解(PROTAC)等均有重要作用。尤其是蛋白质或核酸分子在折叠形成三维结构之后,通常需要装配成各种复合物形式在生命活动中发挥其功能,因此复合物结构的预测是生命科学领域的又一重大挑战。
王晟博士补充道,生物学方向可能更关注复合体问题。了解这些相互作用,可以进一步明确分子的功能,从而为人工干预和药物设计提供思路和基础。此外,在合成生物学领域,精确的复合体建模,也为关键的元件发现和设计提供了基础。比如,在蛋白质-小分子相互作用的预测上,可以辅助我们在酶发现上进一步提高精度和效率,实现在海量序列中发现有功能的酶。了解蛋白质和多糖的相互作用,可以帮助我们设计参与多糖合成的酶,这也是非常受工业界和科学家关注的方向。
在他看来,对蛋白质进行系统深入的研究,能让我们从更深层次诠释生命体的构成和运作变化规律,进而全面揭示生命运行、发展的机制,激发生物科学、药物研发、合成生物学方面的发展。因此蛋白质研究、预测蛋白质结构等,是学术与产业界深度参与的领域。
“在AI时代,得益于算力和算法模型的极大提升,我们终将迎来一个生命科学大爆发时代。”王晟博士表示。不同于往届将蛋白质结构预测作为主要赛道,CASP 15更侧重预测蛋白质复合体结构(原来CAPRI比赛的内容)和RNA结构(原来RNA-Puzzles比赛的内容)。在这届的RNA结构预测中,过去蛋白质结构预测的很多老牌强队,如密苏里大学陈世杰组、RNApolis,David Baker组、密歇根大学张阳组,山东大学杨建益组,也逐渐参与到这些新兴赛道中。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢