用机器学习去研究蛋白质结构预测,吸引了众多科技大厂、科研机构的目光纷纷投入其中,这期间,他们也产出了重要成果。
如在 2021 年 《Science》的十大年度突破中,DeepMind 携预测蛋白质结构的 AI 模型 AlphaFold 上榜,在这项工作的基础上,研究人员现在已经使用人工智能来设计可用于疫苗、建筑材料或纳米机器的全新蛋白质。
在《Science》今年 9 月发表的一篇论文中,华盛顿大学医学院生物化学教授 David Baker 等研究者提出,AI 可以通过两种思路从头设计蛋白质。
当然,对这一领域的热情,自然也少不了 Meta 的加入,就在刚刚过去的 11 月,Meta 拿下微生物领域蛋白质结构预测,开放 6 亿 + 宏基因组蛋白质结构图谱,这是首个蛋白质宇宙「暗物质」的综合视图。
时间刚刚过去一个月,在 FAIR 公布的最新两篇论文中,该研究发现 ESM2 语言模型通过学习深层语法,就能生成天然蛋白质以外的新蛋白质,并可以编程生成复杂和模块化的蛋白质结构。其中 ESM2 模型参数为 150 亿,是迄今为止最大的蛋白质语言模型。
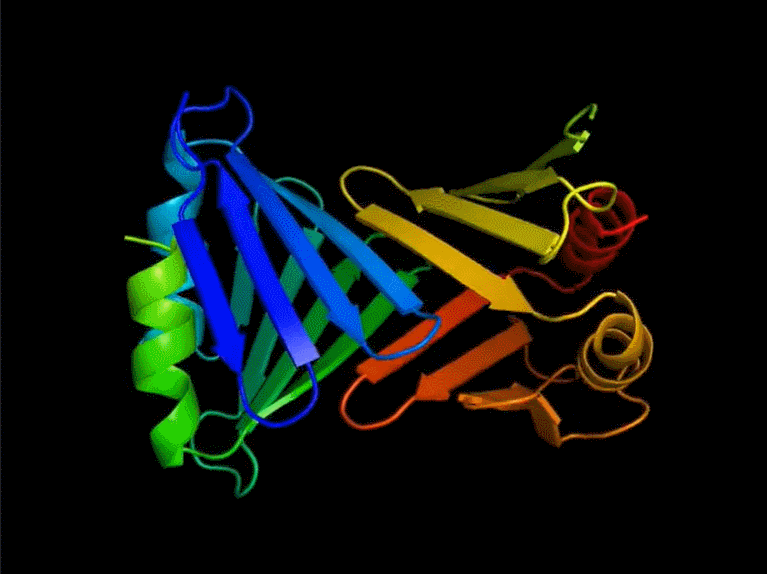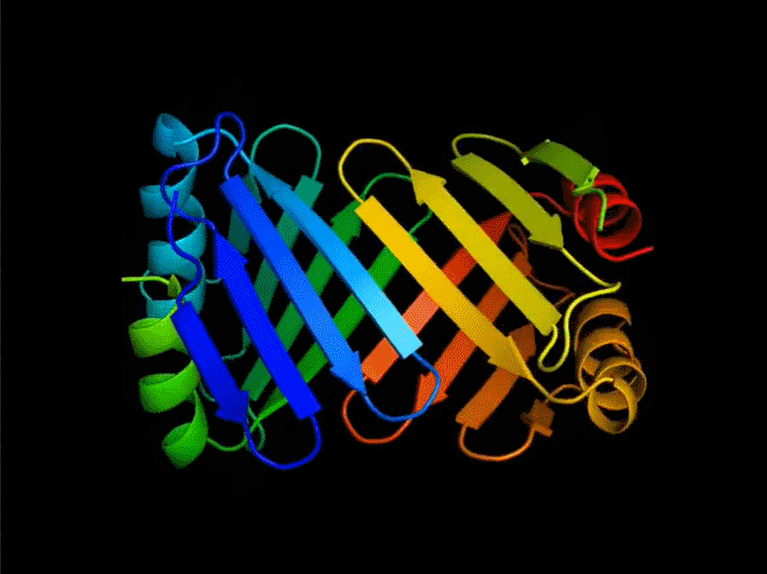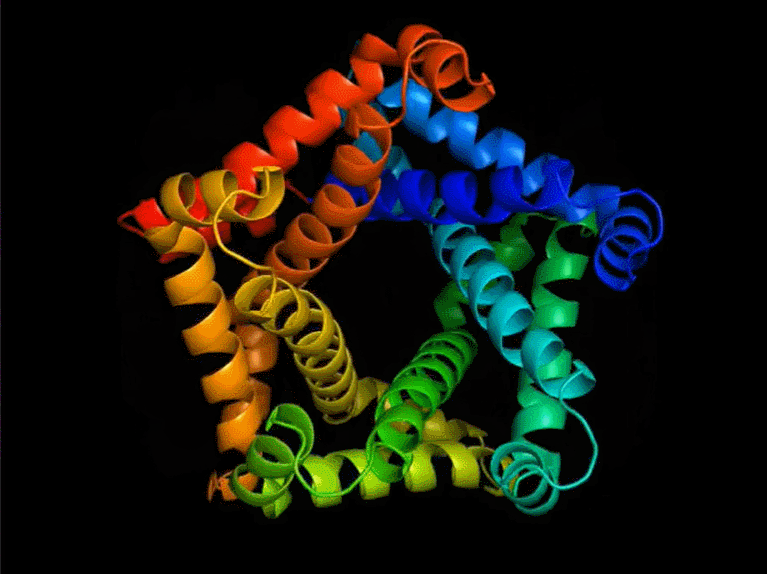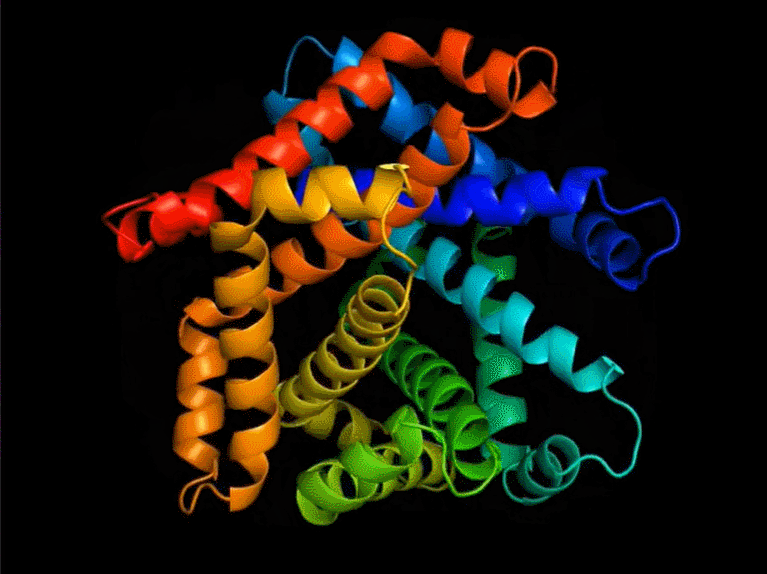
Yann LeCun 在推特上表示:FAIR 开发的新蛋白质设计系统,可以产生与自然界中观察到的蛋白质完全不同的蛋白质;在这个蛋白质设计系统之上,蛋白质可以通过一种编程语言来指定。

在论文《Language models generalize beyond natural proteins》中,ESM2 通过学习深层语法,成功设计了 152 种蛋白质。

论文地址:https://www.biorxiv.org/content/10.1101/2022.12.21.521521v1.full.pdf
在论文《A high-level programming language for generative protein design》中,该研究实现了一种用于生成蛋白设计的高级编程语言。这使得编程生成具有复杂和模块化结构的大蛋白质和复合物成为可能。

论文地址:https://www.biorxiv.org/content/10.1101/2022.12.21.521526v1.full.pdf
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢