【标题】ProT-VAE: Protein Transformer Variational AutoEncoder for Functional Protein Design
【作者团队】Emre Sevgen, Joshua Moller, Adrian Lange, John Parker, Sean Quigley, Jeff Mayer, Poonam Srivastava, Sitaram Gayatri, David Hosfield, Maria Korshunova, Micha Livne, Michelle Gill, Rama Ranganathan, Anthony B. Costa, Andrew L. Ferguson
【发表时间】2023/01/24
【机 构】Evozyne、英伟达
【论文链接】https://doi.org/10.1101/2023.01.23.525232
由于缺乏良好的序列-功能映射的理论模型以及蛋白质序列空间的巨大规模,数据驱动的具有所需功能的蛋白质序列的设计受到了挑战。深度生成模型在通过自然训练数据学习序列与功能的关系以及从该分布中取样来设计具有工程功能的合成序列方面已经显示出成功。本文介绍了一个深度生成模型,称为ProT-VAE,它融合了变异自动编码器的优点,以学习可解释的、低维的隐嵌入和生成解码的条件序列设计。该模型在通用预训练的Transformer编码器和解码器堆栈之间添加了一个轻量级的、针对特定任务的变分自编码器,以无监督或半监督的方式进行无对齐训练以促进对功能序列的理解、优化和生成设计。本文使用英伟达公司的BioNeMo框架,验证了其在功能预测和设计新型蛋白质序列方面的性能,并进行了实验合成和测试。结果显示ProT-VAE隐空间展示了和进化与功能的关联,可以有条件地产生具有高功能和大量序列多样性的新序列。本文预计,该模型可以提供一个可扩展的通用平台,用于机器学习指导的定向进化,以数据驱动的方式设计具有功能的新型合成蛋白质。
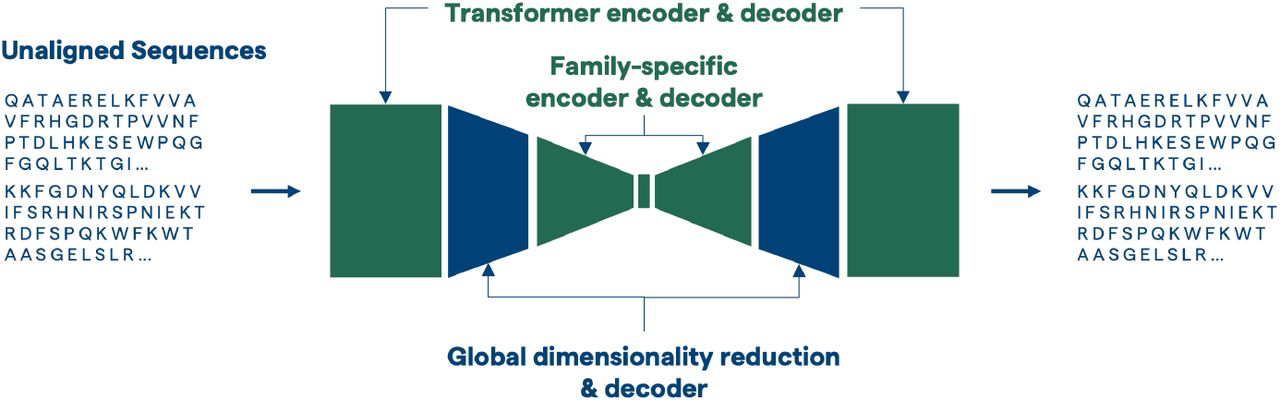
上图展示了ProT-VAE模型架构。一个大型语言模型编码器和解码器作为VAE的最外层模块,未对齐的蛋白质序列首先被送入一个pLM编码器,随后进行卷积降维压缩,压缩后的表征最终被送入一系列全连接层,最后形成一个低维表征。在解码过程中,降低的维度以同样的方式重建,以重建未对齐的蛋白质序列。
- 第一部分的大型预训练模型来自ProtT5,其在英伟达BioNeMo框架中是现成的。该模型有12层,12个注意头,隐藏维度为768,参数为198M。本文ProtT5nv从ProtT5模型开始初始化,随后用UniRef50中的蛋白质序列进一步训练该模型,模型在224个V100 GPU上使用数据并行方式进行训练。
- 第二部分是一个通用的降维区块,其作用是将大约300,000维的transformer隐变量有效地压缩到一个更简单的30000维的中间层表征。这些中间层也同样在大型蛋白质数据库上的重建任务进行了预训练,不需要对每个蛋白质家族进行调整。这个区块由几个堆叠的降维层组成,其中每个层包括1×1卷积,LayerNorm,GeLU激活,过滤器的大小在每一步都是递增的。
- 第三部分是一个三层全连接的最大平均差分自编码器(MMD-VAE),将降维块的扁平化输出压缩到蛋白质家族特定的低维隐空间。该网络是针对特定设计任务中每个目标蛋白家族从头开始初始化和训练的。
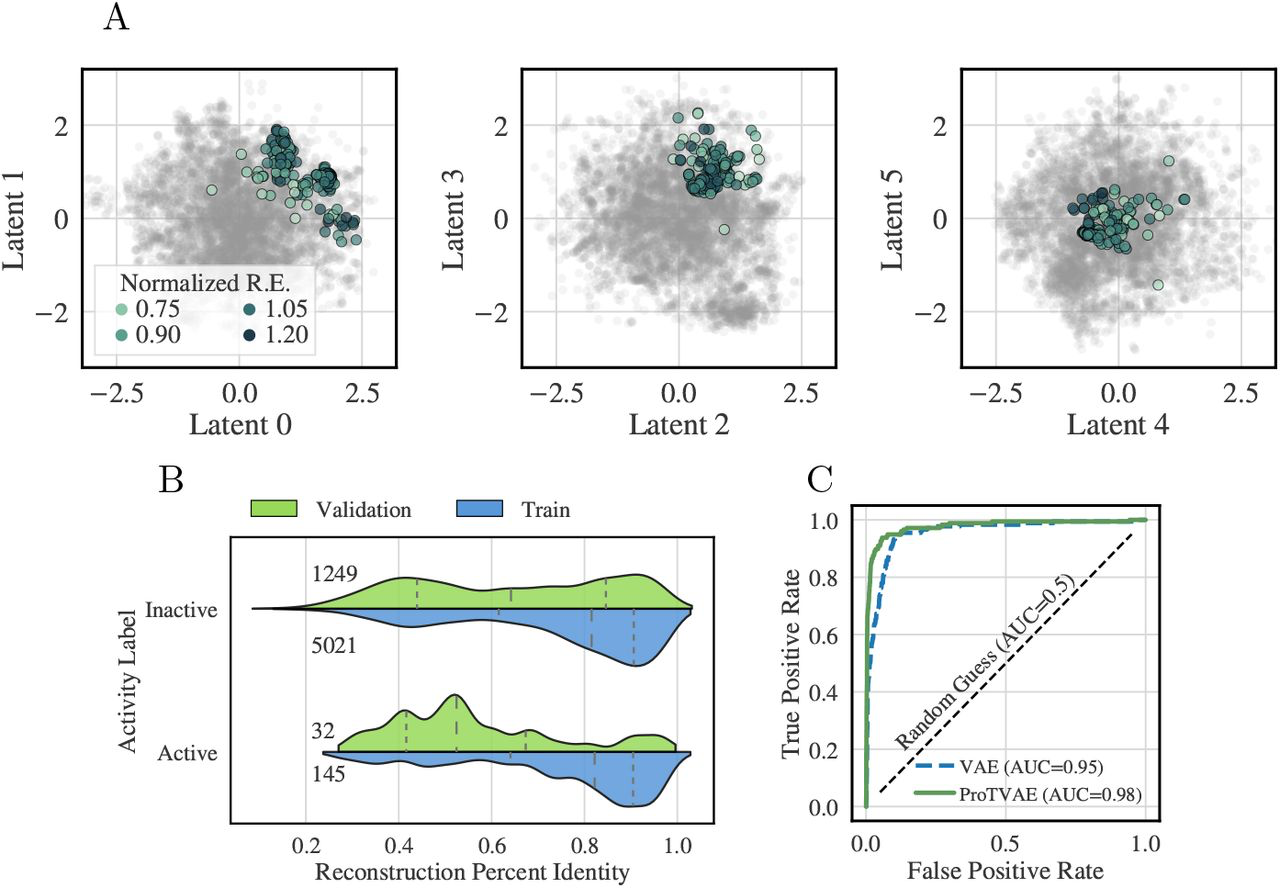
上图展示了ProT-VAE在没有MSA的情况下,SH3家族的功能活性分析结果。
A图展示了隐空间的二维投影按归一化的相对富集度着色,其中深色的点对应于更活跃的序列。
B图展示了以活性为特征的SH3序列的重建强度。每个分类的分布由小提琴图表示,分为训练集(下半部蓝色)和验证集(上半部绿色)中的分布。其中虚线对应于从左到右移动的25%、中位数和75%的四分之一范围。
C图展示了通过隐空间训练的分类模型预测功能。本文通过5折交叉验证来训练逻辑回归分类器,该表明隐空间的使蛋白质的功能得到了定位。绿色实线对应的是ProT-VAE模型,蓝色虚线对应的是以前基于MSA的VAE模型,黑色虚线是无效假设。
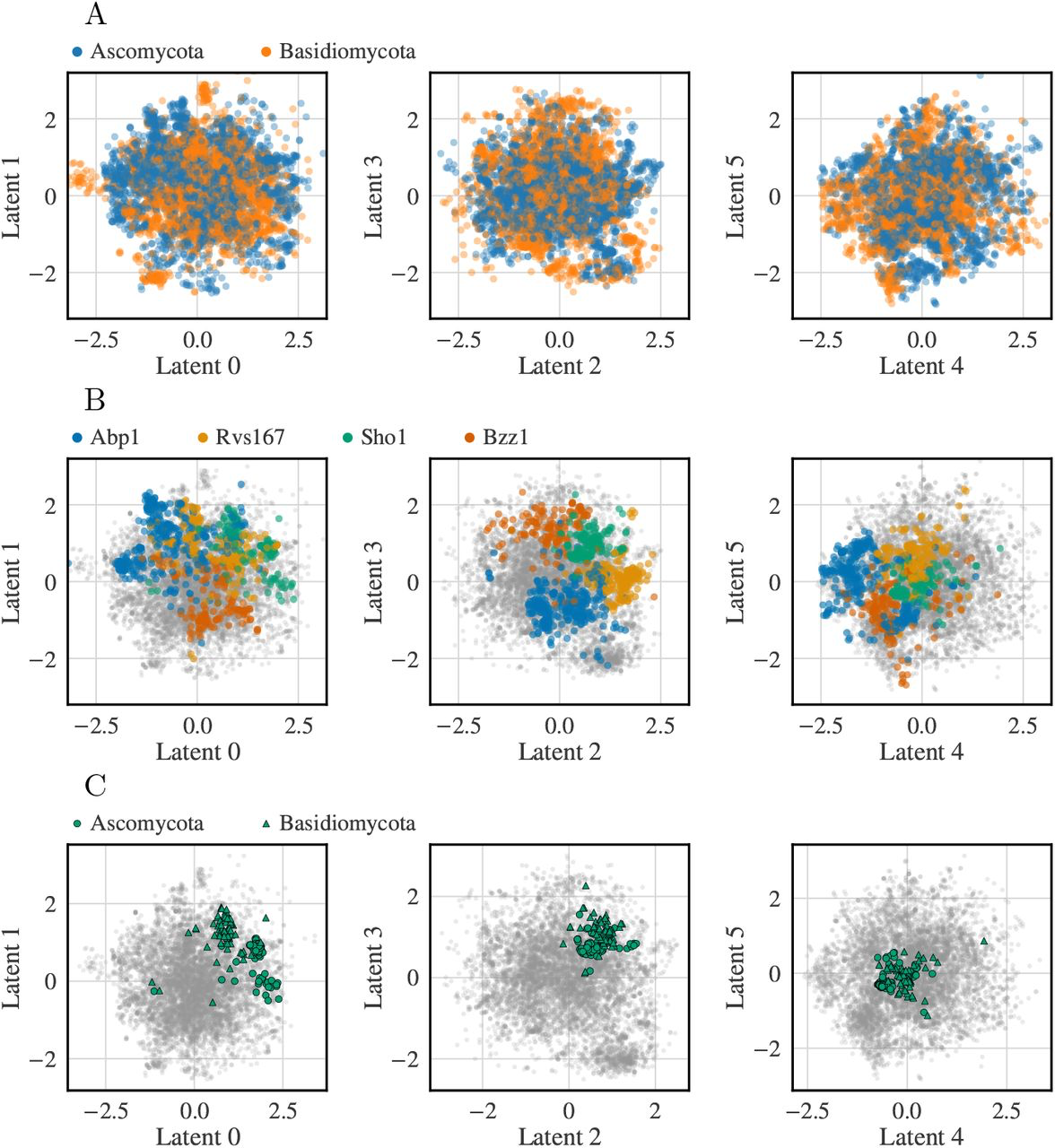
上图展示了ProT-VAE在SH3家族的系统发育分析结果。
A图展示了隐空间的二维投影按两个系统发育组(Ascomycota和Basidomycota)着色,无明显组织。
B图展示了隐空间的二维投影由旁系群着色(蓝色为Abp1,橙色为Rvs167,绿色为Sho1,黄色为Bzz1),显示出强烈的集群。
C图只考虑Sho1类,本文再按系统发育着色,观察到在这个类属组中,子囊菌区和基底菌区有良好的分离。
创新点
-
本文提出了ProT-VAE,一个准确、生成、快速和可迁移的序列-功能关系模型,可用于数据驱动的蛋白质工程。通过融合转化器和VAE的理想特征,该模型允许在无监督或半监督的情况下进行无对齐训练,并提供可解释的低维隐空间,促进对序列功能的理解和生成设计。
-
本文的分析和实验结果证明位于网络核心的瓶颈层中学习到的隐空间根据祖先的历史和生物功能自然地理解了蛋白质序列,暴露了注释序列中的功能梯度,支持高准确度的回归模型来预测新序列的功能,并通过内插解码和条件生成沿着自然序列之间的隐空间路径来实现合成序列的设计。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢