单细胞多组学 (scMulti-omics) 技术允许同时量化多种模态,以捕捉复杂分子机制和细胞异质性的复杂性。现有工具无法有效地推断出不同细胞类型中 active 生物网络以及这些网络对外部刺激的反应。
在此,来自山东大学参与的多机构研究团队,开发了基于深度学习的单细胞数据多组学分析平台:DeepMAPS,用于从 scMulti-omics 进行生物网络推理。DeepMAPS 在异构图中对 scMulti-omics 进行建模,并使用多头图(multi-head graph)Transformer 以稳健的方式学习局部和全局上下文中的细胞和基因之间的关系。
通过构建包含细胞和基因的异构图,DeepMAPS 可以同时识别它们的联合嵌入,并能够在完整框架中推断特定于细胞类型的生物网络以及细胞类型。此外,异构图 Transformer 的应用以可解释的统一多关系对细胞-基因关系进行建模。通过这种方式,可以大大缩短图中的训练和学习过程,以考虑更远距离的细胞影响。
该研究以「Single-cell biological network inference using a heterogeneous graph transformer」为题,于 2023 年 2 月 21 日发布在《Nature Communications》 上。
论文链接:https://www.nature.com/articles/s41467-023-36559-0#Sec9
在这项工作中,研究人员开发了 DeepMAPS,这是一个用于从 scMulti-omics 数据推断细胞类型特定生物网络的异构图 Transformer 框架。该框架采用了一种先进的 GNN 模型,即异构图 Transformer (Heterogeneous Graph Transformer,HGT),具有以下优点:
(1)它制定了一个以细胞和基因为节点,以它们之间的关系为边的一体化异构图。
(2)该模型捕获细胞和基因之间的相邻和全局拓扑特征,同时构建细胞-细胞关系和基因-基因关系。
(3)HGT 模型中的注意机制能够估计基因对特定细胞的重要性,可用于区分基因贡献,增强生物学解释性。
(4)该模型无假设,不依赖于基因共表达的约束,因此可能推断出其他工具通常无法发现的基因调控关系。
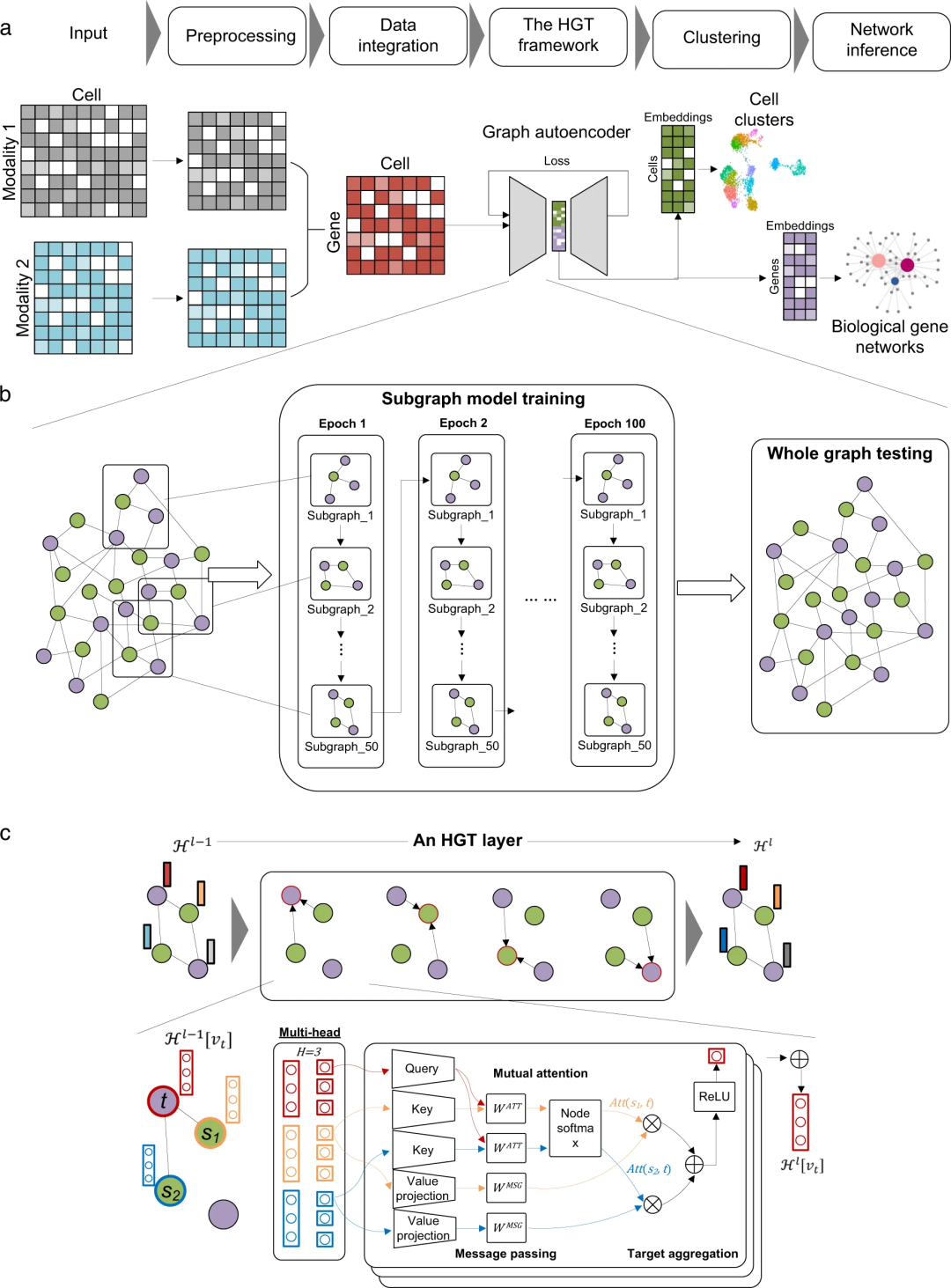
图示:DeepMAPS 和 HGT 插图的工作流程。(来源:论文)
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢