
1 介绍:
Transformer-M是一种基于Transformer的多功能分子模型,在2D和3D分子表示学习中都表现良好。对于一个分子,它的2D和3D形式描述了相同的原子集合,但使用了不同的结构表征。因此,关键的挑战是设计一个表达和兼容的模型,以捕获不同公式的结构知识,并训练参数从这两种信息中学习。Transformer比其他架构更有利,因为它可以显式地将结构信号插入模型中作为偏置项(例如,位置编码)。可以方便地将二维和三维结构信息通过分离的通道设置为不同的偏置项,并将其与注意层中的原子特征结合起来。
Transformer- m的骨干网络由标准Transformer模块组成。作者开发了两个独立的通道来编码二维和三维结构信息。2D通道采用度编码、最短路径距离编码和从2D图结构中提取的边缘编码。最短路径距离编码和边缘编码反映了一对原子的空间关系和键合特征,并作为softmax最大注意中的偏置项。度编码被添加到输入层的原子特征中。对于3D通道,作者使用3D距离编码对3D几何结构中原子之间的空间距离进行编码。每个原子对的欧几里得距离通过高斯基核函数进行编码,并将用作softmax注意中的偏差项。对于每个原子,将其与所有其他原子之间的三维距离编码相加,并将其添加到输入层的原子特征中。图1给出了一个说明。
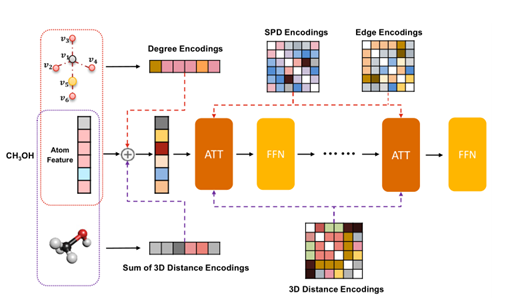
除了两个结构通道中的参数外,Transformer-M中的所有其他参数(例如自关注和前馈网络)对于不同的数据模式都是共享的。作者为Transformer-M设计了一种联合训练方法来学习其参数。在训练过程中,当批处理中的实例仅与2D图结构相关联时,2D通道将被激活,3D通道将被禁用。类似地,当批处理中的实例使用3D几何结构时,3D通道将被激活,2D通道将被禁用。当同时给出二维和三维信息时,两个通道都会被激活。这样就可以从单独的数据库中收集2D和3D数据,并根据不同的训练目标训练Transformer-M,使训练过程更加灵活。
在OGB大规模挑战(OGBLSC)中使用PCQM4Mv2数据集来训练Transformer-M,它由340万个2D和3D形式的分子组成。通过针对3D数据的预文本3D去噪任务,训练模型预测不同格式的每个数据实例的预先计算的HOMO-LUMO间隙。通过预训练模型,可以直接使用或微调不同数据格式的各种分子任务的参数。首先,作者证明了在PCQM4Mv2任务的验证集上,仅包含二维分子图,Transformer-M大大超过了以前的所有模型。这种改进归功于联合训练,它有效地缓解了过拟合问题。其次,在PDBBind上 (2D&3D),与强大的基线相比,经过微调的Transformer-M实现了最先进的性能。最后,在QM9 (3D)基准上,与最近的方法相比,微调后的Transformer-M模型实现了具有竞争力的性能。所有结果表明,Transformer-M具有在化学领域广泛应用的通用模型的潜力。
2 TRANSFORMER-M
定义2D和3D的两种分子式使用了相同的原子特征空间,但结构的表征不同(图形结构E vs几何结构R)。因此,关键的挑战是设计一个兼容的体系结构,该体系结构可以利用E或R(或两者)中的结构信息,并以一种有原则的方式将它们与原子特性结合起来。
Transformer是实现这一目标的合适支柱,因为可以将结构信息编码为偏置项,并将它们适当地插入不同的模块中。此外,使用Transformer,可以通过将结构信息分解为成对编码和原子编码,以统一的方式处理E和R。
在这项工作中使用的主干架构是Transformer模型。Transformer由堆叠的Transformer块组成。Transformer块由两层组成:自关注层,然后是前馈层,两层都具有规范化和跳过连接。表示工作方式如下:
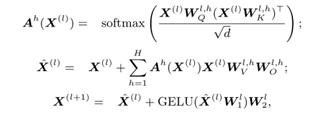
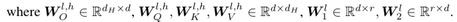
使用两个术语来编码图中任意原子对之间的结构关系。首先,对两个原子之间的最短路径距离(SPD)进行编码,以反映它们的空间关系。其次,沿着i和j之间的最短路径编码边缘特征(例如化学键类型),以反映键信息。
对欧几里得距离进行编码,以反映三维空间中任意一对原子之间的空间关系。对于每个原子对(i, j),用高斯基核函数处理它们的欧几里得距离并进行编码。

通过这种简单的方法,分子的二维和三维结构信息被整合到一个Transformer模型中。很容易检查Transformer-M是否保留了两种数据格式的等变属性。
下一步是学习Transformer-M中的参数,以便从每种数据格式中捕获有意义的表示。为了实现这一目标,作者开发了一种简单灵活的联合训练方法来学习Transformer-M。具体来说,在训练期间,如果数据实例来自2D格式的数据集,则2D通道被激活,3D通道被禁用。模型参数将被优化以最小化2D目标。当数据实例来自3D格式的数据集时,只有3D通道被激活,模型将学习最小化3D目标。如果模型以2D和3D两种格式的分子作为输入,这两个通道都会被激活。由于联合训练,Transformer-M可以更好地泛化。之前的一些研究发现,二维图形结构和三维几何结构包含互补的化学知识。通过参数共享的二维和三维数据联合训练,该模型可以学习更多的化学知识,而不是对数据噪声过拟合,并且在二维和三维任务上都有更好的表现。
作为最初的尝试, Transformer-M开辟了一种开发通用分子模型的方法,以处理不同数据格式的各种化学任务。这是一个起点,未来有更多的可能性可以探索。例如,在这项工作中,使用了一种简单的方法,将二维和三维结构的结构信息线性组合,作者认为应该有其他有效的方法来融合这种编码。该模型还可以与以前的多视图对比学习方法相结合。如何使用这些方法预训练该模型是值得研究的。
3 实验
3.1大规模预训练
Transformer-M是在OGB大规模挑战赛的PCQM4Mv2训练集上进行预训练的。训练样本总数为337万。每个分子都与其二维图形结构和三维几何结构相关联。每个分子的HOMO-LUMO能隙作为标签。采用12层Transformer-M模型。隐藏层和前馈层的维数设置为768。注意头的数量设置为32。高斯基核数设置为128。为了训练Transformer-M,为每个数据实例提供三种模式:(1)激活2D通道并禁用3D通道(2D模式);(2)激活3D通道,禁用2D通道(3D模式);(3)激活两个通道(2D+3D模式)。训练过程中每个数据实例的模式是根据预定义的分布随机绘制的,实现类似于Dropout。在这项工作中,使用了两个训练目标。第一个是监督学习目标,旨在预测每个分子的HOMO-LUMO能隙。此外,还使用了一个名为3D位置去噪的自监督学习目标,这是特别有效的。在训练过程中,如果数据实例处于3D模式,在每个原子的位置上添加高斯噪声,并要求模型从噪声输入中预测噪声。该模型被优化为最小化上述两个目标的线性组合。
3.2 PCQM4MV2性能(2D)
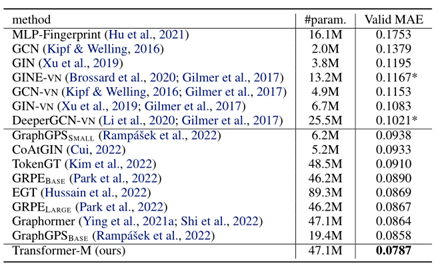
PCQM4Mv2的验证集仅由2D格式的分子组成。该任务的目标是预测HOMU-LUMO能量差,评估指标是平均绝对误差(MAE)。结果如表1所示:Transformer-M大大超过了所有基线。Transformer-M的总体架构与graphhormer模型相同,但它们之间的唯一区别是graphhormer仅使用2D数据进行训练,而Transformer-M使用2D和3D结构信息进行训练。因此,可以得出结论,Transformer-M在二维分子数据上表现良好,共享参数的2D-3D联合训练确实有助于模型学习更多的化学知识。
3.3 PDBBIND性能(2D & 3D)
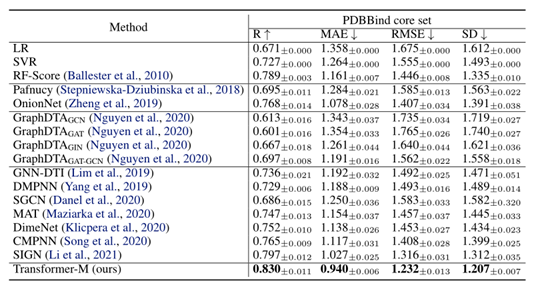
将Transformer-M模型与包括经典方法、基于CNN的方法和GNN的竞争性基线进行了比较。结果如表2所示。Transformer-M在所有评估指标上的表现都明显优于所有基线。值得注意的是,PDBBind数据集的数据实例是蛋白质配体复合物,而Transformer-M模型是在简单分子上进行预训练的,这证明了Transformer-M的可转移性。
3.4 QM9性能(3D)
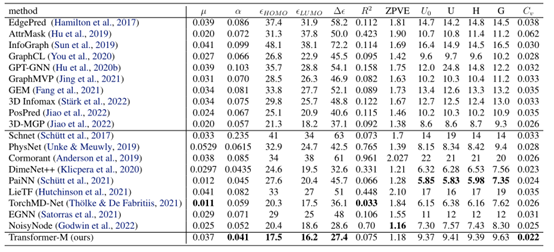
使用QM9数据集以3D数据格式评估Transformer-M在分子任务上的表现。结果如表3所示。Transformer-M取得了具有竞争力的性能,这表明该模型与3D分子数据兼容。特别是,Transformer-M在HUMO、LUMO和HUMO-LUMO预测上表现最好。这表明在预训练任务中学到的知识可以更好地转移到类似的任务中。
3.5消融实验
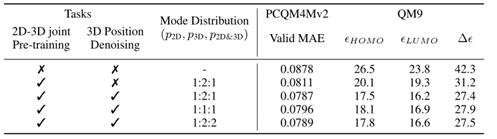
Transformer-M模型通过两个任务在PCQM4Mv2训练集上进行预训练:(1)预测分子在2D和3D格式下的HOMO-LUMO间隙。(2)三维位置去噪。对PCQM4Mv2和QM9数据集进行消融研究,以检查这两个目标是否有利于下游任务。结果如表4所示。可以看出,联合预训练在PCQM4Mv2和QM9数据集上都显著提高了性能。此外,3D位置去噪任务也是有益的,特别是在3D格式的QM9数据集上。
通过实验研究了不同分布对模型性能的影响。选择三个分布(p2D, p3D, p2D&3D)分别为:1:1:1,1:2:2和1:2:1。结果如表4所示。在PCQM4Mv2和QM9数据集上得到了一致的结论:1)对于所有三种配置,Transformer-M模型都取得了较强的性能,这表明联合训练对超参数选择具有鲁棒性;2)在3D模式上使用稍大的概率可以获得最佳效果。
4 结论
这项工作向通用分子模型迈出了第一步。提出的Transformer-M提供了一种很有前途的方法来处理2D和3D格式的分子任务。使用两个独立的通道对二维和三维结构信息进行编码,并将其集成到主干Transformer中。当输入的数据是特定格式时,相应的通道将被激活,另一个通道将被禁用。通过对2D和3D分子数据的简单训练任务,模型自动学习利用不同数据格式的化学知识并正确捕获表征。进行了大量的实验,所有的实证结果都表明,Transformer-M可以同时在2D和3D任务上取得较强的性能。Transformer-M的潜力可以在化学领域的广泛应用中得到进一步的探索。
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢