编译 | 曾全晨
审稿 | 王建民
今天为大家介绍的是来自Connor W. Coley团队的一篇解决图神经网络中分子手性问题的论文。如果分子具有立体化学-一种空间结构特征,即使它们的图连接性相同,也可能表现出不同的物理和生物特性。然而,现代用于从分子结构中学习结构-性质关系的神经网络架构将分子视为图结构数据,无法处理分子的这种立体性。在这里,作者开发了两种自定义聚合函数,用于消息传递神经网络来学习具有四面体手性的分子的性质,这是一种常见的立体化学形式。

近年来,化学领域的机器学习技术取得了重大进展,使得可以直接从分子结构中学习,而无需将它们预处理为固定长度的向量。这些进展在各种情况下都取得了改进,包括性质预测,预测合成和分子优化。然而,立体化学在深度学习的视野下仍然大多未被探索,尽管它是分子表示中的一个重要方面。立体异构体是具有相同图连接性但空间排列不同的分子结构。每一种类型都会影响小分子的可访问构象,进而影响分子的性质。立体异构体可能具有不同的物理性质(例如熔点和沸点),药物动力学性质(例如吸收、分布、代谢和排泄)以及生物活性(例如蛋白质亲和性)。研究专注于四面体手性及其对性质预测的影响。原则上,具有四面体手性的分子既不是2D图形也不是3D结构,而介于两者之间。也就是说,手性中心的立体化学指定限制了可访问构象的范围,但并不限定一个单一的3D结构。因此,作者认为使用3D表示来处理四面体手性对于构象灵活的分子来说过于受限。
消息传递神经网络(MPNNs)通过将原子视为节点、化学键视为边,将分子表示为图形来进行操作。MPNNs通过迭代地聚合邻居节点的表示来进行操作;传统的图形结构数据的聚合函数,如求和、均值和最大值,都是对称操作符。由于立体异构体具有相同的图连接性,对称聚合器在两个不同的手性中心上操作将会将它们的邻居节点折叠为相同的表示,无论手性如何;也就是说,聚合函数是有效实现MPNN架构中手性的主要障碍。
模型方法
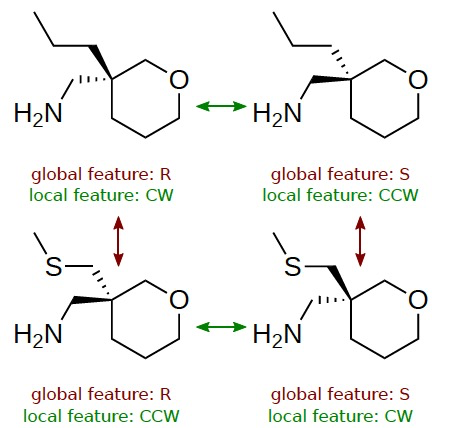
图 1
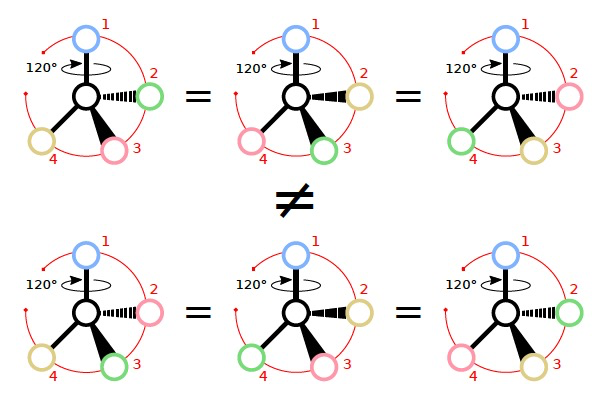
图 2
表面上,MPNNs可以通过原子或键级描述符来捕捉立体化学,例如R/S原子特征来区分对映异构体(即非可重叠的立体异构体,它们是彼此的镜像)。这些特征代表手性的全局度量,通过CIP侧链排序规则来确定。图1说明了为什么作为原子特征的全局手性描述符对于MPNNs来说是不足以有意义地区分这些结构的。通过全局描述符,两个“R”结构将接收相同的原子级特征,而两个“S”结构将接收不同的特征。然而,虽然共享相同的全局描述符,但四面体中心的局部配置对于每个“R”结构是不同的。
另一种方法是通过局部手性描述符来捕获相关信息,图1中用CW/CCW标签表示,这在SMILES表示法中使用。手性中心根据相对于其空间方向的邻近原子的顺序(例如在提供的SMILES字符串中)而接收奇偶标签(CW或CCW),将它们分配给12个等价排列方式中的一个局部手性组。作者的模型在保持物理上有意义的不变性的同时,将这个局部奇偶位与有序的邻居集合结合起来,而不是直接将该奇偶位作为原子特征使用,以避免对任意的记录和SMILES规范化约定敏感。
对于四面体的手性问题,模型只要确保相同手性组上P(C)的聚合函数的模型结构能保证输出结果相同即可:
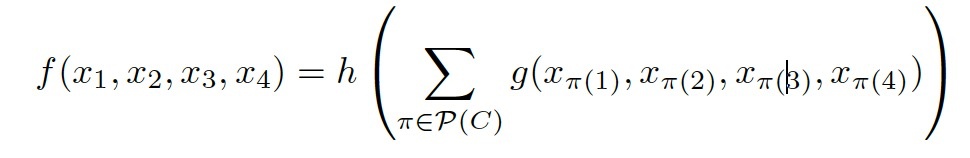
在实际应用中,手性组的排序被分为表格1所示的两种。表格中数字的为按照排序搜索的原子排序结果。
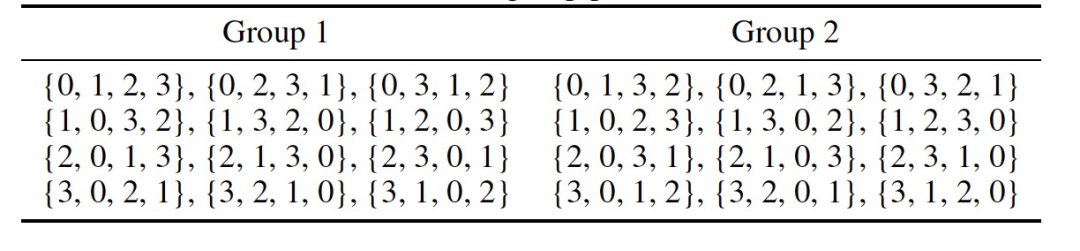
表 1
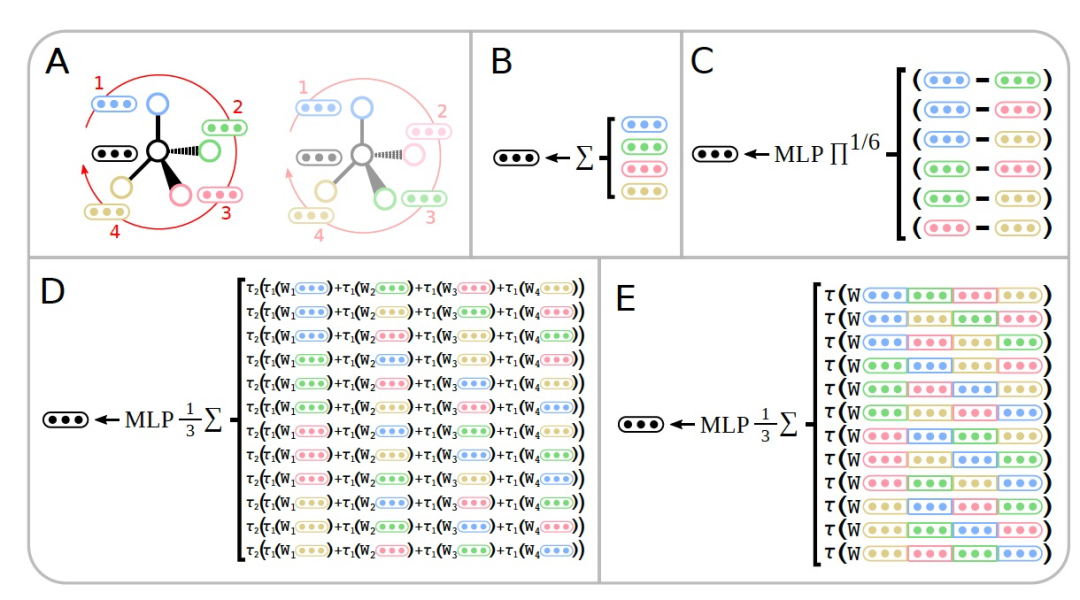
图 3
为了满足设计要求,作者提出了两种聚合方式。第一种方式为排序方式(PERM,图3D为具体计算方式)。这种方式将表格2中所有相同手性组的排序结果均计算一遍,从而保证相同手性组所得结果一定相同,不同手性组结果不同。第二种方式为聚合方式(PERM_CAT,图3E为具体计算方式),思路与第一种并无不同。
实验
大多数属性预测数据集缺乏干净的立体化学信息,无法对支持立体化感知模型进行基准测试。为此,作者从D4多巴胺受体蛋白-配体对接筛选数据集中提取了一部分数据。立体异构体在与感兴趣的蛋白质结合时可能表现出不同的相互作用能,这是由于它们可以达到的构象/姿态不同。数据集(D4DCHP)将原始的1.38亿分子缩小为单个1,3-二环己基丙烷骨架的立体异构体对。我们作者进一步定义了两个额外的子集:一个子集中,对映异构体的对接得分相差超过5 kcal/mol(DIFF5),另一个子集中,分子具有单个四面体中心(CHIRAL1)。作者还在“Lipo”数据集上评估了性能,其中4200个分子中有1127个分子至少包含一个四面体中心;然而,与D4DCHP数据集不同,此数据集没有完整的对映异构体对。
为了确保新聚合方法能够理解四面体立体化学,作者首先在将分子从CHIRAL1分类为R或S的简单任务上对它们进行评估。这个任务测试了MPNN是否能够(1)区分对映异构体,(2)学习CIP规则来分配R/S标签。由于对映异构体对的输入原子/键特征和图连接是相同的,作者预期使用求和聚合器的准确率将达到50%。实证结果(表2)支持了这一假设。MPNN体系结构中的求和聚合器表现不如随机分类器,而所有三种体系结构的自定义聚合器在将分子分类为R或S时准确率接近完美。
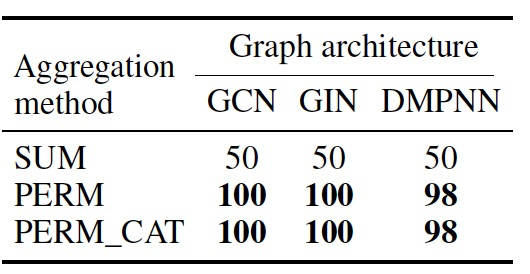
表 2
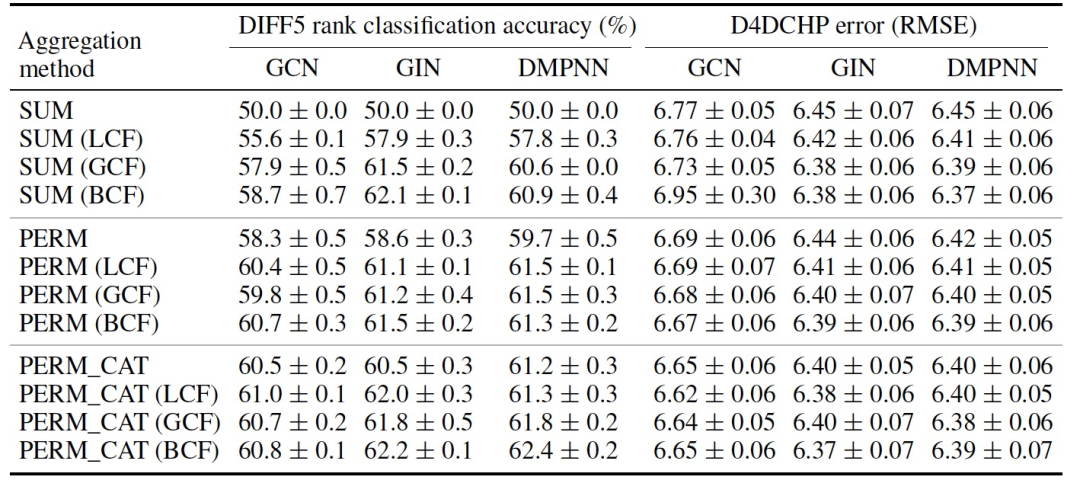
表 3
接下来,作者在完整的D4DCHP数据集上评估实证性能(表3)。在没有原子级别的立体特征的情况下,PERM和PERM_CAT的非对称聚合方法相比于SUM聚合器基线具有显著优势。在存在立体特征的情况下,不同的聚合方法的性能取决于体系结构。自定义聚合函数在GCN体系结构中在排名分类和RMSE方面均显示出可衡量的改进,但对于GIN和DMPNN体系结构来说,这些改进并不明显。对于GIN,包括原子级立体特征就足以捕捉到在缺乏这些特征时由自定义聚合器捕捉到的趋势。对于DMPNN来说,自定义聚合器在排名分类方面提供了改进,但在RMSE方面没有。原子级立体特征的强烈影响令人惊讶。通过只包括两个额外的原子级特征,SUM聚合器在排名分类方面的准确率约提高了10%,而不管图形体系结构如何。更具表现力的图形体系结构可以更有效地使用这些立体信息,以至于自定义聚合器提供的显式等变性不再提供显著的优势。然而,最高观察到的62%的排名分类准确率仍然有改进的空间,这激发了进一步研究能够更好地处理四面体立体化学的表示学习方法。
结论
作者开发了两种聚合函数来学习具有四面体手性的分子的性质。自定义聚合器可以在玩具R/S分类问题上完全区分对映异构体,并且在新提出的D4DHCP数据集上,根据MPNN体系结构和原子级别立体特征的包含与否,表现出与基准SUM聚合器相当或适度改进的性能。
参考资料
Pattanaik, L., Ganea, O. E., Coley, I., Jensen, K. F., Green, W. H., & Coley, C. W. (2020). Message passing networks for molecules with tetrahedral chirality. arXiv preprint arXiv:2012.00094.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢