设计出具有某些期望化学性质的化合物是药物研发领域的核心工作。在计算机辅助药物设计领域中,从头设计(de novo design)一直是计算化学家们关注的重点方向之一。近年来,由于人工智能技术的不断发展,机器学习引入计算机辅助药物设计领域,彻底改变了从头配体设计的任务,各种从头生成药物分子的深度学习模型层出不穷。然而,新领域的出现往往会伴随着诸多新的挑战,目前药物分子生成模型性能的评估方法主要还是通过计算生成分子的简单性质对模型进行评估,在药物设计这种复杂场景中显然是不合理的。因此,该领域的挑战之一是缺乏评估分子生成模型实际性能的基准测试方法。为了解决以上问题,雅盖隆大学Tobiasz
Ciepliński团队提出了一套基于分子对接的基准测试方法,通过将生成分子的对接打分和结构多样性分析结合,对分子生成模型进行真实且有效的评估,同时还发掘了目前分子生成模型的局限性,该项研究工作发表在美国化学会出版的计算化学核心刊物Journal of Chemical
Information And Modeling(J. Chem. Inf.
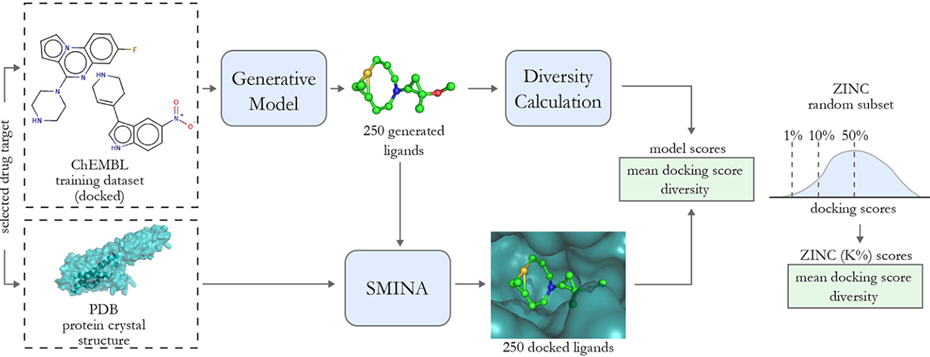
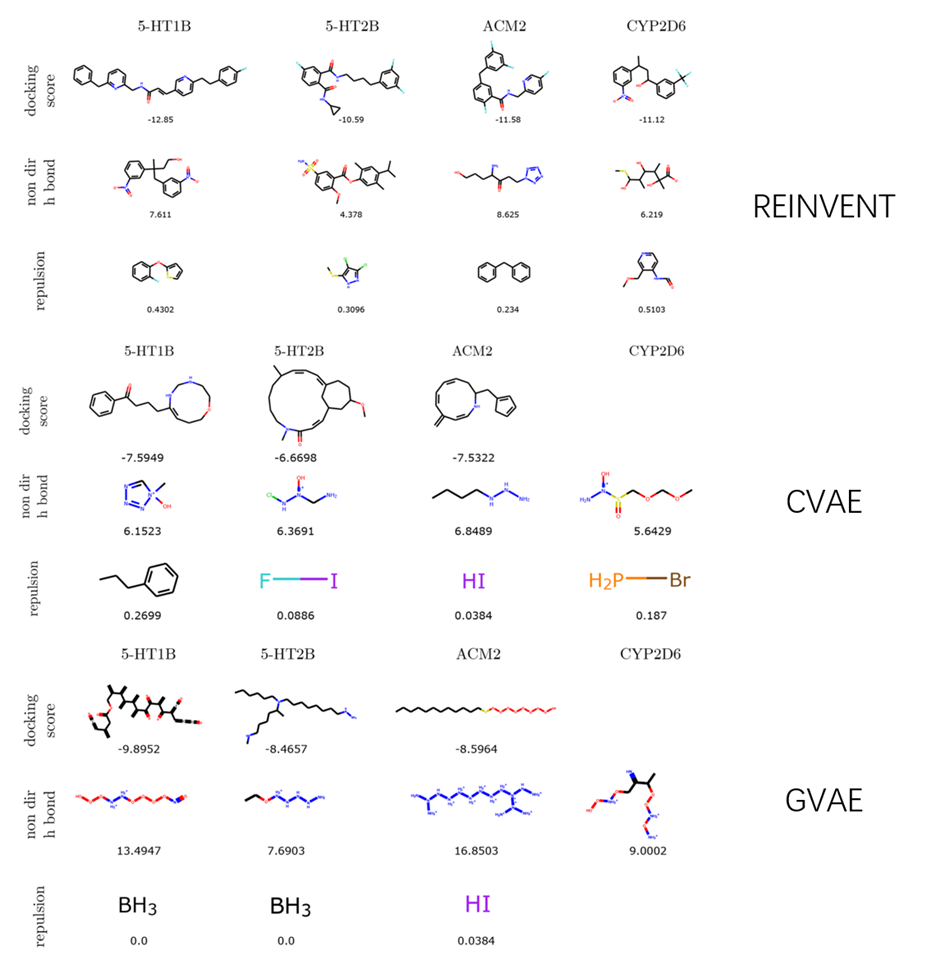
Model. 2023, 63, 3238−3247)[1]。本研究中基准测试方法的设计主要分为三个部分:①分子对接软件,用来生成给定化合物在结合部位的对接构象。本研究中选择的分子对接软件为被广泛使用并且可免费获取的SMINA。②对接构象评分函数,本研究选择vina docking score作为评分函数的基础(公式1),取其中的repulsion和non_dir_h_bond项作为更简单的评估标准与总对接分数进行对比。③已经计算出对接得分的训练集化合物,本研究选择ChEMBL数据库中针对5-HT1B,5-HT2B,ACM2, CYP2D6四个靶点测试活性的化合物和ChEMBL数据库外针对ADRB1,MOR,A2A,D2四个靶点测试活性的化合物,从PDB数据库中下载相对应的蛋白使用SMINA进行分子对接,只有对接成功的分子才会被保留,保留下来的分子会记录其活性标签和对接得分。Dockingscore = - 0.035579 * gauss – 0.005156 * gauss + 0.840245 * repulsion – 0.035069 * hydrophobic – 0.587439 * non_dir_h_bond(公式1)该方法测试分子生成模型共分为5个步骤(图1):①使用课题组提供的链接下载与所选药物靶点相关的结构数据和活性数据,结构数据包括蛋白与化合物的结构,活性数据包括化合物的活性标签(有活性或无活性)、对接得分。②将已经训练完成的生成模型优化目标修改为总对接分数(或其他优化目标),使用对应靶点的化合物训练集对调整优化目标后生成模型进行微调,微调完成后的模型各生成250个非重复分子。③使用Lipinski规则过滤生成的化合物,并确保每个分子的分子量大于100。④对过滤后的化合物进行对接并打分,每个化合物分子取对接得分排名前5的对接构象,将这5个构象的对接得分去平均值即为该化合物的最终得分,挑选出模型生成的·250个化合物中打分的最大值与作者设计的基线进行对比,同时对生成分子的多样性进行评价。⑤重复上述流程,分别使用训练集中的全部八个靶标进行测试,综合八个靶点的打分和多样性评价结果对模型性能进行评估。本研究选择了两个方案作为模型评估的基线:①从ZINC数据库中随机抽取9204719个分子,对这些分子的多样性进行评估,并将这些分子与基准测试方法中8个靶点进行对接打分,得到的对接分数按前1%,前10%和前50%进行记录。②将训练集化合物的分子进行多样性评估,并将对接分数按前1%,前10%和前50%进行记录。在确定了基线之后,本次研究选取了CVAE,GVAE,REINVENT三个分子生成模型进行评估,前两个模型为变分自编码器模型,最后一个为强化学习模型,首先将三个模型各复制3份,再将复制的三份模型的优化目标分别设置为总对接得分、repulsion项得分、non_dir_h_bond项得分,最后按上述的工作流程对三个模型进行测试。在总对接得分优化任务中,在除去ACM2的所有靶点上,三个模型生成分子的最高得分都低于ZINC数据库前10%的分子得分,并且REINVENT模型在该任务中表现最好(在5HT1B靶点中,ZINC前10%分子的得分为-9.894,GVAE与CVAE分别为-4.995和-4.647,REINVENT为-9.774),这意味着目前的生成模型仍然无法生成满足要求的高亲合性化合物。在repulsion项的优化任务中,REINVENT模型的表现远不如CVAE和GVAE,并且所有模型都无法超过在ZINC数据集中找到的排名前10%的分子,但相比于总对接分数优化任务,三个模型的表现都有提升。在non_dir_h_bond项的优化任务中,GVAE和REINVENT的得分与ZINC数据集前1%的分子不相上下。对三个模型生成分子的多样性进行评估,发现REINVENT模型生成分子的多样性最差。本研究还将三个模型在三个优化任务中生成的得分最高的分子挑选出来进行分析(图2),发现以repulsion为优化目标的模型更偏向于生成分子量小的分子,以non_dir_h_bond为优化目标的模型更偏向于生成有较多氮氧原子的分子,只有以总对接得分为目标的模型生成的分子更贴合实际。综上所述,作者认为以总对接得分为优化目标的测试方法更加具有挑战性,并且更能反应生成分子的实际性能。小结: 分子生成模型的开发逐渐成为计算机辅助药物设计领域内的核心研究方向,近年来,大量的药物分子生成模型不断涌现,但模型的评估标准还停留在对分子简单性质的计算上,阻碍了该方向的发展。本研究提出了一个新的生成模型基准测试方法,使用分子的对接得分作为评价指标,从而为各类生成模型提供了更加接近于真实环境的基准测试方法,为评价体系的发展提供了新的思路。[1] Tobiasz Ciepliński,
Tomasz Danel, Sabina Podlewska, and Stanisław Jastrzȩbski. Generative Models Should at Least Be Able to Design Molecules That
Dock Well: A New Benchmark. Journal of Chemical Information and Modeling 2023
63 (11), 3238-3247.
内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢