蛋白质与蛋白质之间相互作用是维持生物体正常功能的基础之一。通过相互作用,蛋白质可以形成稳定的复合物结构并发挥其功能。例如,酶与底物之间的结合就是通过蛋白质与蛋白质之间的相互作用来实现的。这种相互作用可以使酶与底物之间形成稳定的结合,从而促进酶催化反应的进行。此外,蛋白质与蛋白质之间的相互作用还可以参与到信号传导、细胞黏附和免疫应答等生物过程中。因此,探索蛋白质与蛋白质之间如何相互作用对于揭示生命体执行机器-蛋白质作用机制以及基于靶标复合物的药物设计具有重要意义。随着深度学习技术的发展,蛋白质人工智能结构预测方法取得了突破性的进展。在单链蛋白质,特别是单域蛋白质结构预测问题基本解决的后AlphaFold2时代,通过深度学习方法预测蛋白质复合物结构,揭示蛋白质-蛋白质相互作用机理成为当前生物信息学领域迫切需要解决的前沿、热点科学问题。DeepMind团队基于AlphaFold2开发的AlphaFold-Multimer,通过对模型部分调整以满足复合物结合面结构的特殊需要,实现了蛋白质复合物结构的端到端建模。然而,端到端方法往往需要大量的硬件资源,而且高度依赖于共进化信息,在对复合物结构建模时,有可能忽略或过度估计一些相互作用,无法揭示复合物结构域间的相互作用机制。因此,围绕结构域相互作用关系,开发轻量级的高精度蛋白质复合物结构建模新方法,有望应对复合物接口存在处于不同单链中若干不连续结构域相互缠绕这一挑战性问题。近日,浙江工业大学张贵军教授课题组在Nature旗下期刊《Communications Biology》上在线发表了题为《Multi-domain and complex protein structure prediction using inter-domain
interactions from deep learning》的研究论文(图1)。研究团队在之前开展的蛋白质单域结构建模方法PAthreader(Communications Biology,2023),理化模型副本交换采样多域建模方法DEMO(PNAS, 2019)、深度学习辅助计算智能空间采样多域建模方法SADA(Bioinformatics, 2022)、深度学习端到端多域建模方法E2EDA(JCIM, 2023)的工作基础上,进一步提出了以结构域为单位实现蛋白质复合物组装的深度学习方法DeepAssembly。DeepAssembly尝试通过从多域蛋白质中学习到的域间相互作用来实现蛋白质复合物结构预测,提供了一种以结构域为单位来组装复合物结构的轻量级方法,进而缓解算法对计算机硬件的要求。该论文通讯作者为浙江工业大学信息工程学院张贵军教授,课题组夏瑜豪博士生、赵凯龙博士生为论文共同第一作者。图1.《Communications biology》发表论文
一、研究思路
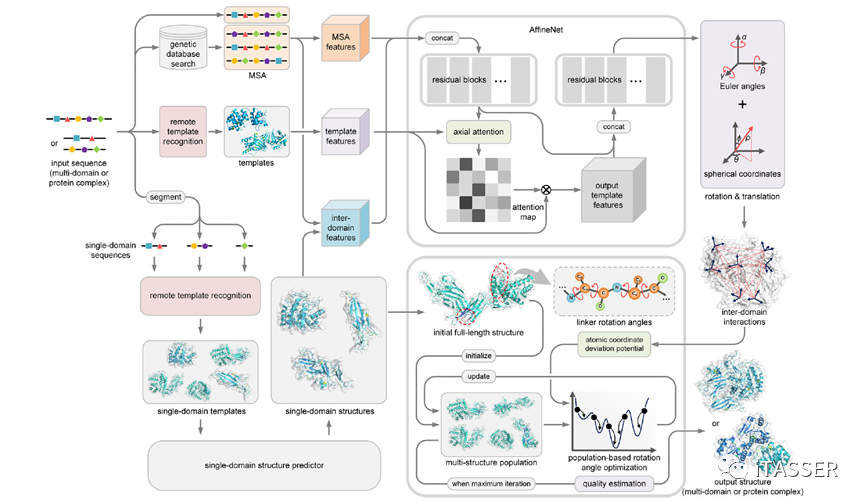
DeepAssembly研究方案如图2所示。对于输入的多域蛋白质或者复合物序列,首先生成多序列比对,并使用课题组先前开发的远程模板识别方法PAthreader(Communications Biology,2023)搜索远程同源模板,利用课题组开发的域分割工具DomBpred(TCBB, 2022)将输入序列分割成单域序列,然后通过PAthreader预测单域蛋白质结构,进一步将从多序列比对、远程同源模板和域边界信息中提取的特征输入到深度神经网络中预测域间相互作用。最后,在预测的域间相互作用约束下,设计基于计算智能的群体优化方法将单域结构组装成全链或复合物结构,最后采用课题组开发的复合物模型质量评估模型GraphCPLMQA(Briefings in Bioinformatics, 2023)选择最佳模型作为输出结构。图2.DeepAssembly流程图
二、实验结果
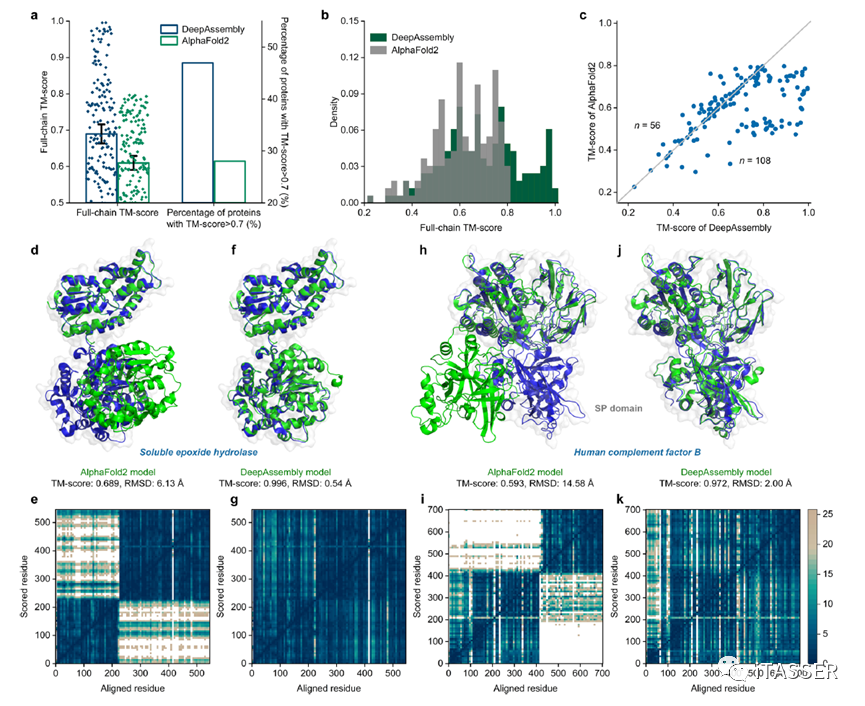
在多域蛋白质全链建模方面,作者从AlphaFold DB数据库中构建了164个人类基因组测试蛋白,并与目前最主流的端到端全链建模方法AlphaFold2进行了比较。实验结果表明,DeepAssembly的平均TM-score比AlphaFold2高出了13.1%(图3)。DeepAssembly可以有效纠正AlphaFold2预测模型中错误的域间方位,这表明可能为AlphaFold DB提供一种可行低碳的更新方式。图3. DeepAssembly与AlphaFold2在多域结构建模上的比较
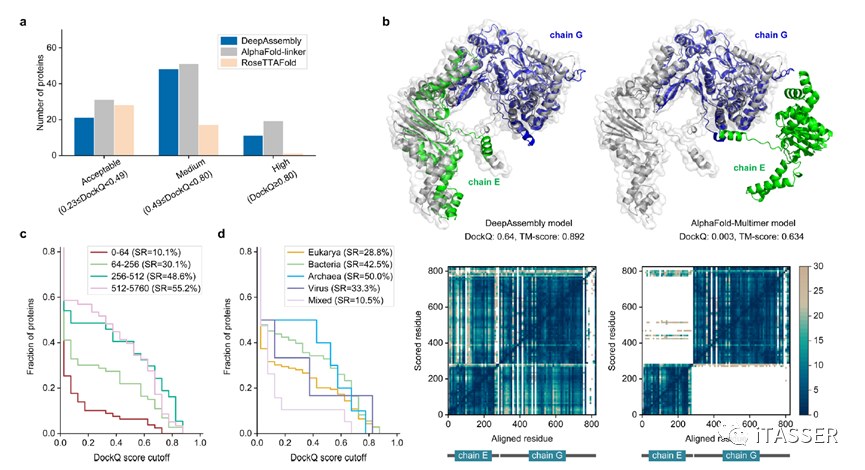
进一步,通过对融合蛋白和相应复合物序列-结构观测,考虑到单链中多结构域相互作用与复合物中多链的所有结构域相互作用机制可能存在等同性,进一步扩展至基于结构域(而非单链)的复合物组装。实验结果表明,在247个异源二聚体测试集上,DeepAssembly在32.4%的测试蛋白上成功预测了正确的链间相互作用(DockQ>0.23)。在21%的测试集上,预测得到了比AlphaFold-Multimer质量更高的复合物模型,如图4中显示的由病毒RNA聚合酶E链和G链组成的异源二聚体的案例,DeepAssembly的预测结果明显优于AlphaFold-Multimer,这表明DeepAssembly的计算模型与之具有一定互补性。另一方面,通过以结构域为单位进行复合物结构组装,在一定程度上缓解了算法对计算机硬件的高需求,提供了一种轻量级的复合物结构组装新思路。图4. DeepAssembly在异源二聚体上的性能分析
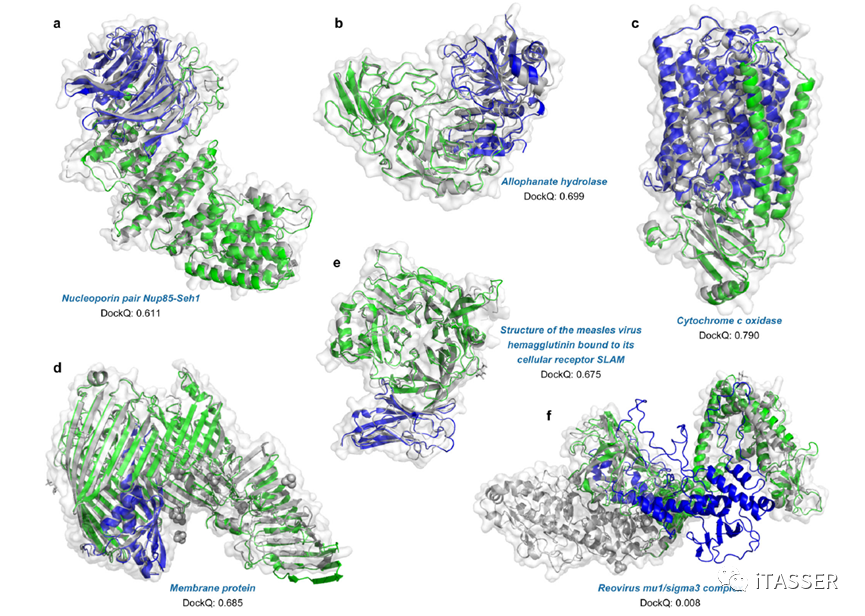
图5显示了DeepAssembly在测试集上的预测案例。作者分析了共进化信息与远程同源模板对该方法的影响。分析表明,共进化信息与模板具有互补性。在没有远程模板的情况下,共进化信息是主要的驱动因素。而当缺乏共进化信息时,远程模板则起到了关键的引导作用。例如图5e中显示的麻疹病毒血凝素与其细胞受体SLAM结合的蛋白质结构,其几乎没有配对的共进化信息,这意味着仅使用这种不足的共同进化信号来预测链间相互作用是相当困难的。而DeepAssembly通过远程模板特征学习到了一定的链间相互作用,正确预测了其的链间界面,在一定程度上减少了对共进化信息的依赖。三、结论
通过以结构域为组装单元,DeepAssembly从多域蛋白质中学习域间相互作用并应用于蛋白质复合物结构预测,这表明通过域间相互作用的方式可以提供一种轻量级的复合物组装方式,从而为复合物的预测,进而探测蛋白质-蛋白质相互作用以及作用通路提供了一种可行、轻量级的思路。该工作也在一定程度上表明,结构域间相互作用虽然对多域蛋白质和复合物而言有一定的共性,但对复合物而言,当结构域处于不同的单链时,这一作用更为柔性,更加难于捕获。目前,蛋白质动态特性研究成为热点,这些新的问题也会给现有AI建模方法带来挑战,虽然通过修改输入、参数可以发现一定的动态现象,但可能并不够系统、优雅,新的问题的解决,未来变革性的技术突破值得期待。原文链接:
https://www.nature.com/articles/s42003-023-05610-7
服务器链接:
http://zhanglab-bioinf.com/DeepAssembly/
内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢