
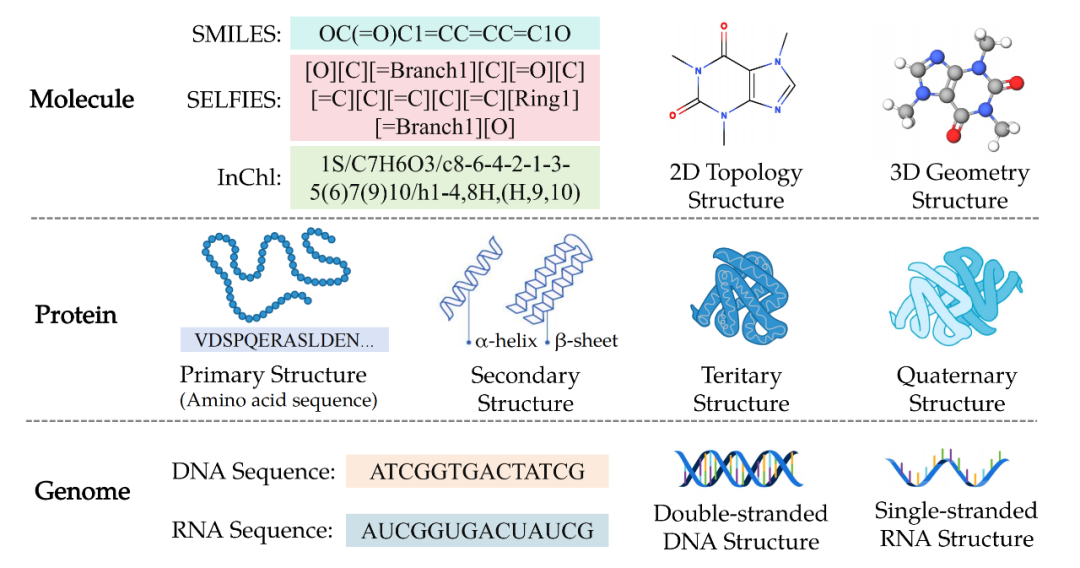
大型语言模型(LLMs)已成为推动自然语言理解能力变革的关键力量,标志着人工智能通用性方面的重大突破。LLMs 的应用已超越传统自然语言的范畴,覆盖了各类科学学科中开发的专用科学语言系统,从而推动了科学语言大模型(Sci-LLMs)的诞生。
作为科学人工智能领域的崭新方向,Sci-LLMs 值得深入研究。然而,目前关于 Sci-LLMs 的进展调查尚属不足。本综述从生命科学和物质科学等视角,围绕生物和化学两个领域全面回顾和梳理了 Sci-LLMs 的最新进展,着重分析了针对文本科学知识、小分子化合物、大分子蛋白质、基因组序列以及多模态科学数据的 LLMs,共计 75 页,引用了 300 余篇参考文献。
综述由浙江大学杭州国际科创中心生物与分子智造研究院 AI 交叉中心团队组织撰写,参与人员来自浙江大学计算机科学与技术学院、浙江大学化学与生物工程学院、浙江大学药学院、之江实验室等多个跨学科单位。

文本科学大语言模型 分子大语言模型 蛋白质大语言模型 基因组大语言模型 多模态科学大语言模型
每个主题都分别从模型、数据集、评估、总结四个方面展开描述。
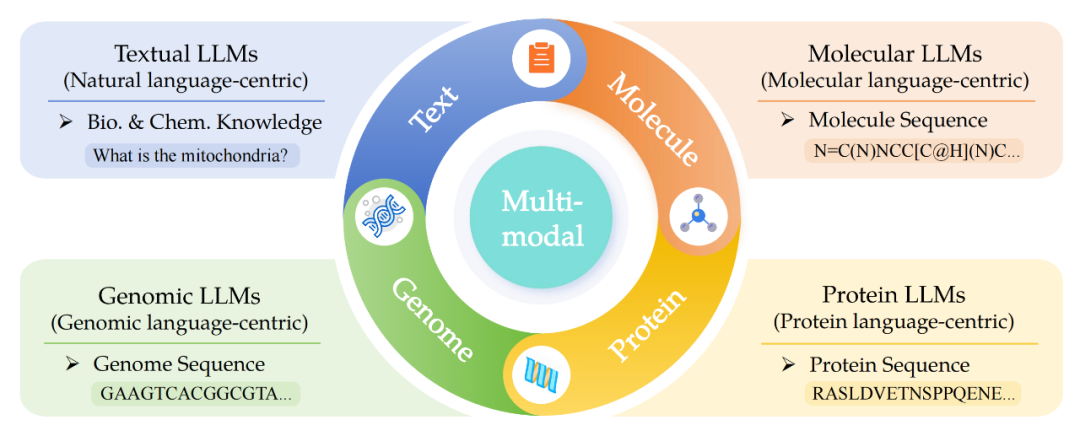
▲ 图2. 本综述的Sci-LLMs研究范围
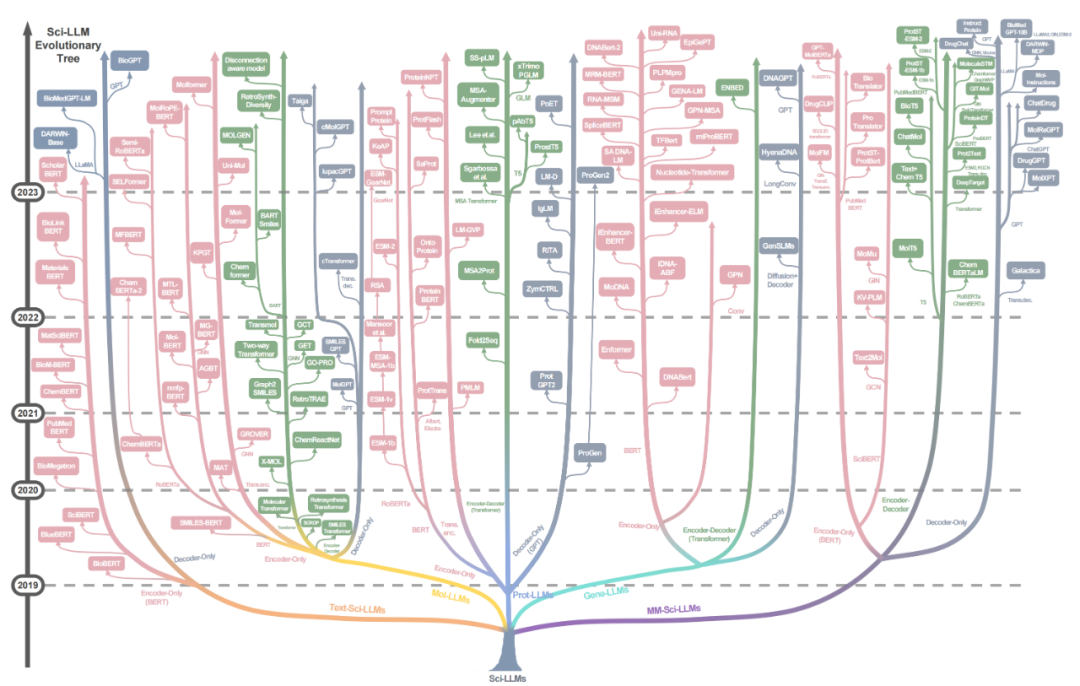
▲ 图3. 本综述的科学大语言模型的进化树

文本科学大语言模型
该综述首先深入探讨了文本科学大语言模型(Text-Sci-LLM),如图 4 所示。所谓文本科学大语言模型,是指使用专业文本语料库(如科学文献)训练得到的模型。该章节主要从生物和化学领域出发,描述了多个相关模型,数据集和评估方法,并采用了一种名为 KnowEval 的评估方法,考察在基础知识,专业知识和创新知识方面的能力。
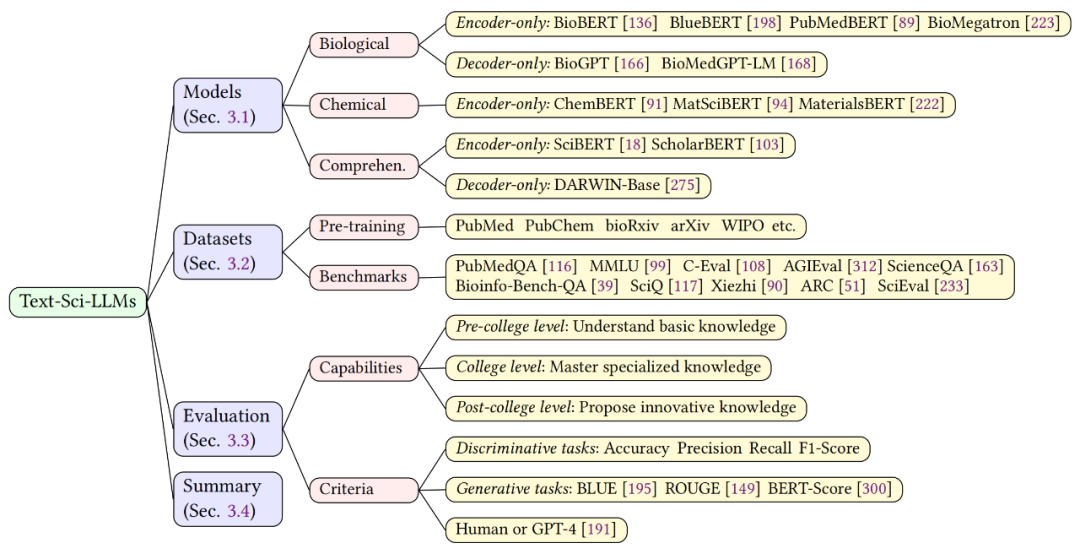
▲ 图4. 文本科学大型语言模型概述

该综述提供了对分子语言大模型(Mol-LLM)的概述,如图 5 所示,包括各种模型及功能,使用的数据集和评估标准。模型方面列举了诸多基于不同架构的分子语言模型,重点关注这些模型如何解释和处理化学语言,然后从预训练和 benchmark 两个方面对数据集进行整理,此外还通过分子性质预测、相互作用预测、反应预测和分子生成对模型进行评估,尤其是对分子生成领域提供了关键的评估指标。
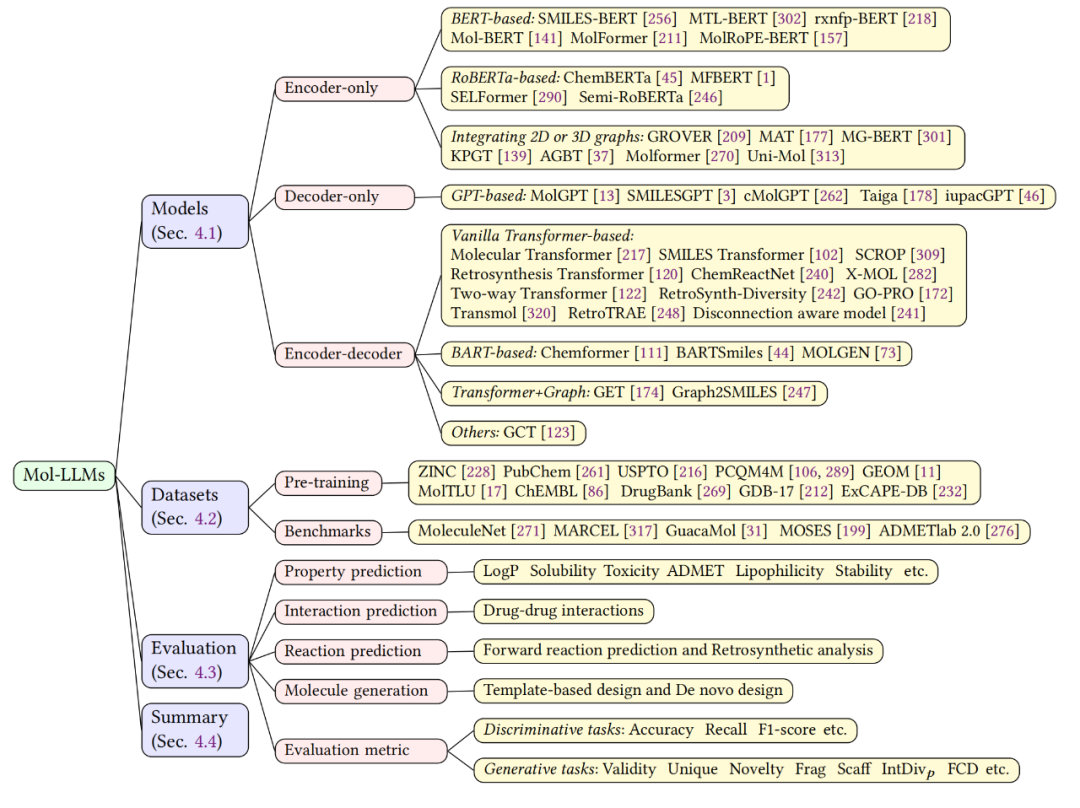
▲ 图5. 分子大语言模型概述

该综述回顾了蛋白质语言大模型(Prot-LLM)的进展,如图 6 展示。在过去数年里,大语言模型在蛋白质研究领域取得了重要突破,为深入理解及操控蛋白质提供了新的技术。本章节首先整理并分析了基于不同架构的蛋白质语言模型,然后列举了广泛使用的数据集,最后阐述了评估方法,包括结构/功能预测和序列设计,为蛋白质模型的进一步发展提供重要的技术支持。
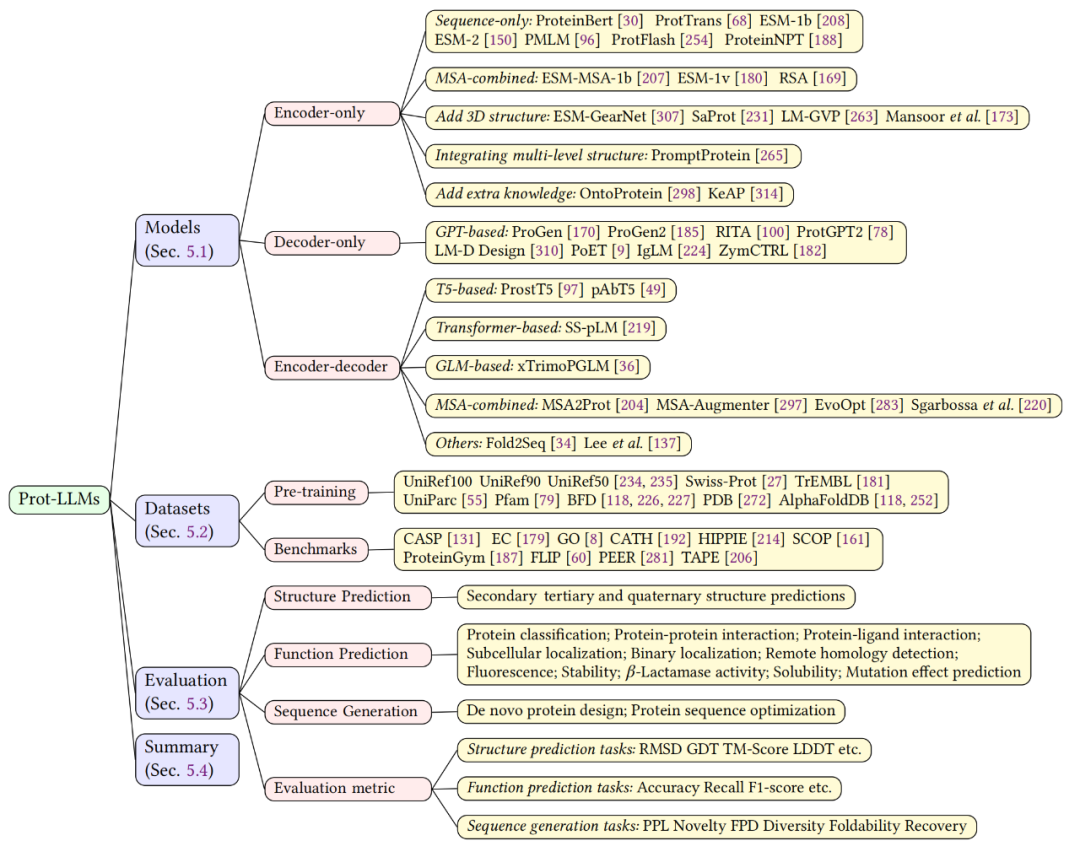
▲ 图6. 蛋白质大语言模型概述

基因大语言模型
该综述中还探讨了基因语言大模型(Gene-LLM)在计算生物学领域的发展,如图 7 所示。本章节对 Gene-LLM 的模型架构、数据集和评估方法进行了详细介绍。文中深入探讨了这些模型在计算生物学中用于分析 DNA 和 RNA 序列的应用,强调了这些模型在理解基因功能、预测染色质谱、剪接位点、结合位点及结构的重要性,并阐述了它们在序列生成、分析序列变异和进化方面的能力。
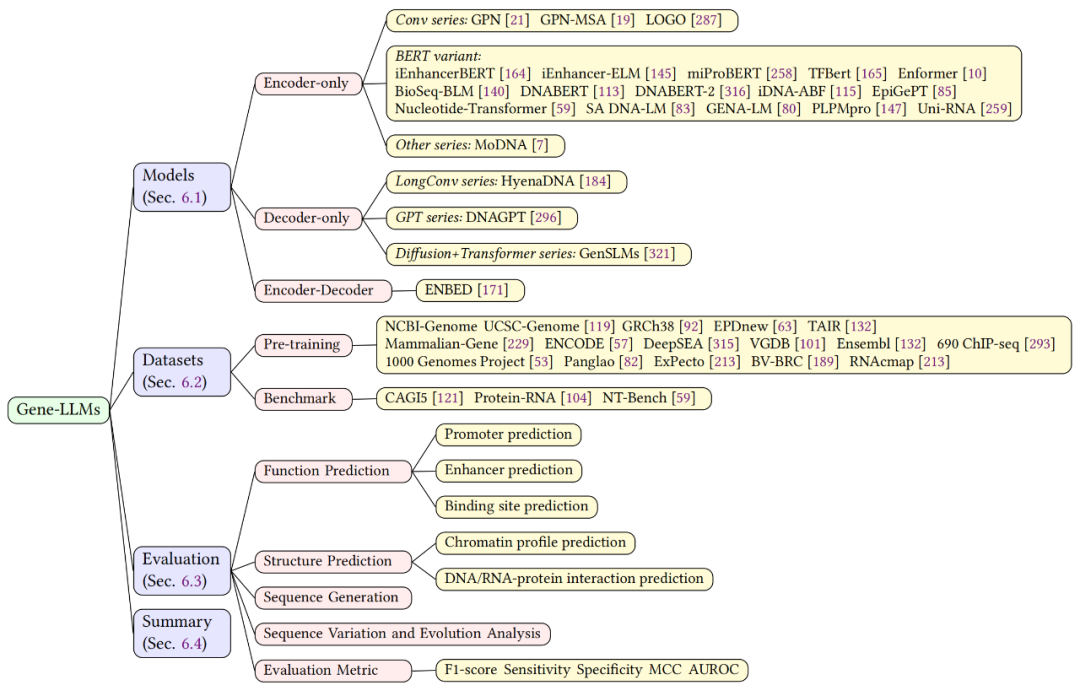
▲ 图7. 基因组大语言模型概述

最后,该综述深入探讨了集成多种科学语言的多模态科学大模型(MM-Sci-LLMs),涵盖文本、分子、蛋白质及基因等多种模态。本章节对这些模型的架构设计和能力进行了详细分析。同时,审视了训练和评估模型所需的数据集,并指出了模型评估的关注要点。多模态科学大模型融合了多种科学语言的交互,是当前 Sci-LLMs 领域的研究热点。
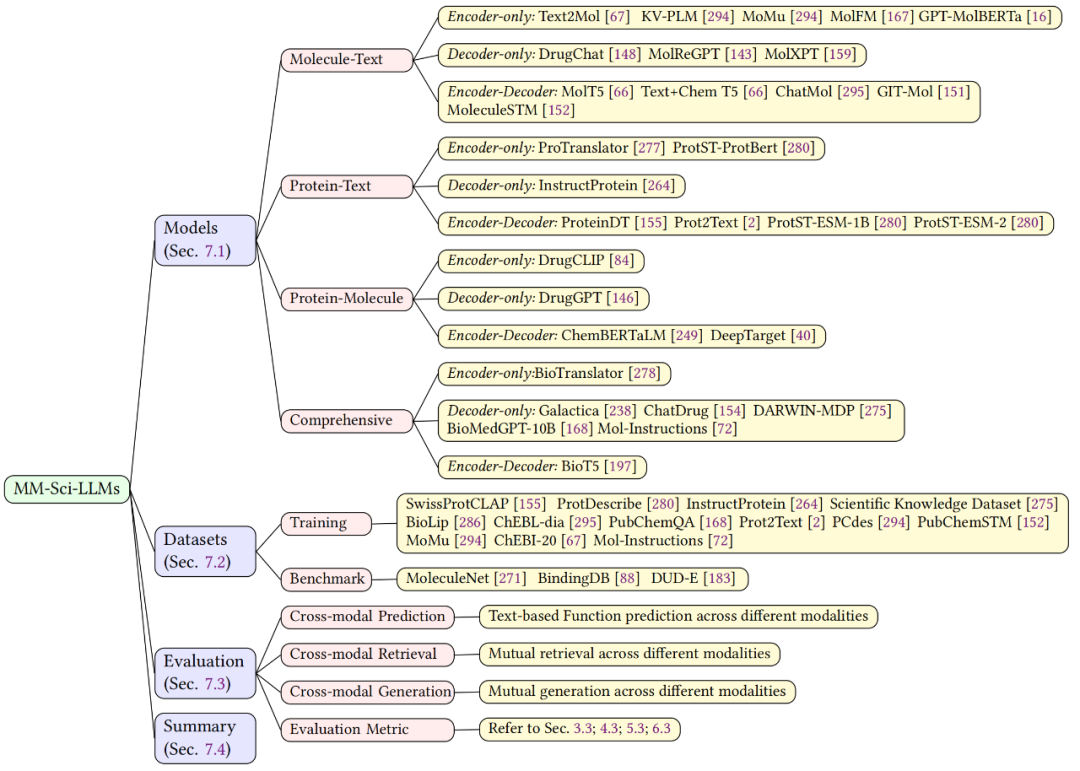
▲ 图8. 多模态大语言模型概述

尽管过去的研究在科学大语言模型(Sci-LLMs)领域取得了显著成果,但不可否认的是,这一领域仍处于初级阶段。本文对现有研究进行了深入审查,提出了尚待解决的几个关键挑战。
1. 训练数据:数据是人工智能模型开发的基础。本文认为,预训练数据集的规模、微调数据集的质量和跨模态数据集的匮乏是影响该领域增长和有效性的关键因素。
2. 模型架构和学习目标:尽管大多数 LLMs 依赖基于 Transformer 的架构来学习语言中的语义相关性,但这种方法对 Sci-LLMs 可能不是最优的。首先,科学语言通常包含比常规自然语言句子更长的序列;其次,当前的语言模型难以明确包含关键的 3D 结构信息;最后,自然语言通常以自回归方式生成,然而,Sci-LLMs 的理想学习目标应允许模型从整个序列中捕获语义信息来生成。
3. 模型评估:对科学大模型的有效评估一直是研究难点。尽管本文列举了诸多计算指标,可以为生成的蛋白质或分子提供质量评估。然而这些指标并不是决定性的,实验室中的湿实验仍然是验证的黄金标准,但它们通常超出许多 AI 研究团队的范围和能力。这一差距凸显了迫切需要更强大、更可靠的计算评估和基准测试系统。
4. 伦理问题:在开发和应用 Sci-LLMs 时,考量伦理问题至关重要的,特别是在处理敏感的生物数据时,需要严格的控制措施。此外,与常规的大语言模型类似,Sci-LLMs 也可能会加剧其训练数据中所存在的偏见,从而导致科学研究结果产生偏差。

7. Sci-LLMs 与人类价值观的超级对齐(Super-alignment)。
这篇综述对科学大语言模型在生物化学领域的发展进行了系统回顾。文章阐述了科学语言的基本概念,包括科学文本、分子、蛋白质和基因,并深入研究了各类科学语言特点,分析了 Sci-LLMs 的最新进展,包括深入探讨模型架构、功能、数据集和评估方法。最后,确定了四个关键挑战,并提出了多个具有前景的研究方向。本综述旨在为人工智能和生命科学、物质科学等交叉领域提供全面的资源,促进合作,推动“人工智能助力科学”(AI for Science)的发展。
AI交叉中心团队简介
团队GitHub主页:
https://github.com/HICAI-ZJU
近期部分论文:
● Knowledge graph-enhanced molecular contrastive learning with functional prompt, Nature Machine Intelligence, 2023.
● Direct prediction of gas adsorption via spatial atom interaction learning, Nature Communications, 2023.
● Learning Invariant Molecular Representation in Latent Discrete Space, NeurIPS, 2023. ● Newton-Cotes Graph Neural Networks: On the Time Evolution of Dynamic Systems, NeurIPS, 2023.
● Multi-level Protein Structure Pre-training with Prompt Learning, ICLR, 2023.
● Graph Sampling-based Meta-Learning for Molecular Property Prediction, IJCAI, 2023.
● Knowledge-graph-based cell-cell communication inference for spatially resolved transcriptomic data with SpaTalk, Nature Communications, 2022.
● De novo analysis of bulk RNA-seq data at spatially resolved single-cell resolution, Nature Communications, 2022.
● OntoProtein: Protein Pretraining With Gene Ontology Embedding, ICLR, 2022.
● Molecular Contrastive Learning with Chemical Element Knowledge Graph, AAAI, 2022.
更多阅读


内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢