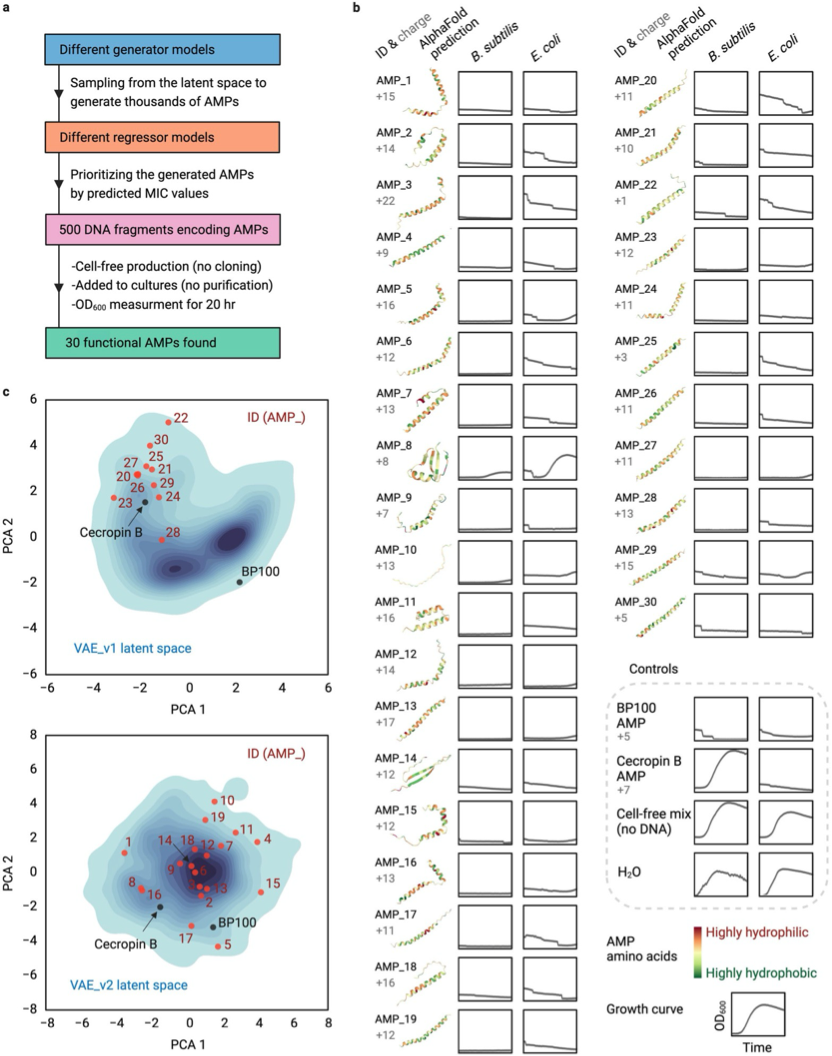
为了从头设计抗菌肽,研究者采用了此前研究中报道的两个损失函数不同的深度生成变分自编码器(VAE)。他们首先使用来自UniProt的约150万个多肽序列作为通用数据集对VAE进行预训练,然后使用约5000个已知AMP的数据集对预训练VAE进行迁移学习(图1a)。为了减少实验测试的抗菌肽数量,他们建立了一种根据预测的生物活性(如最低抑制浓度,MIC)来选择潜在候选多肽的方法,采用CNN/RNN混合的神经网络进行线性回归拟合,并用约5000个已知AMP的序列和对应的最低抑制浓度进行训练(图1b)。他们最初通过采样生成了约50万条新多肽,之后根据长度及可行性筛至约5万条,最后优选了500条用于后续的生物活性试验(图2a)。
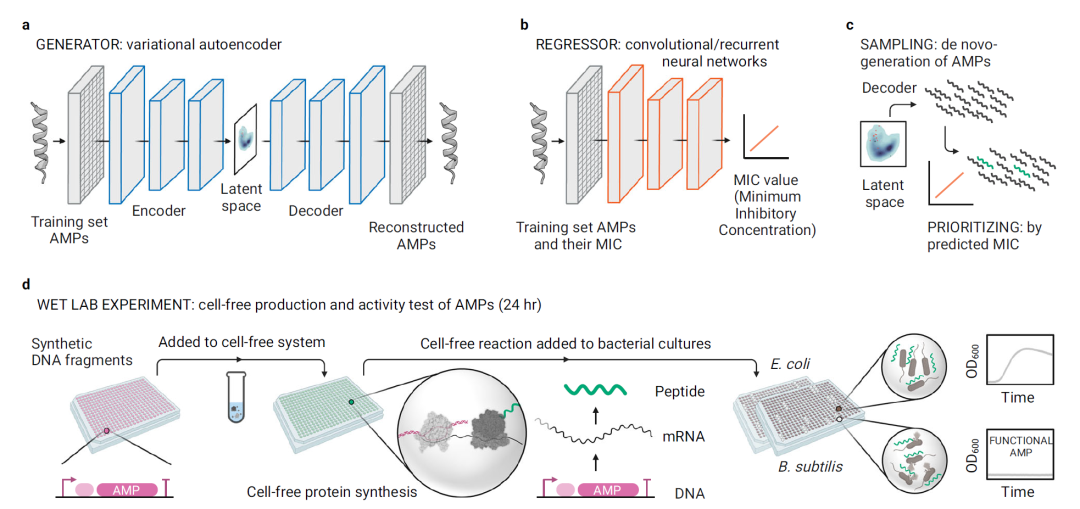
为对深度学习生成的抗菌肽进行高通量活性验证,研究者建立了基于无细胞蛋白质合成的抗菌肽高通量筛选系统。作者首先合成了线性的DNA模板,依次包含T7启动子、核糖体结合位点(RBS)、抗菌肽编码区以及T7终止子。然后将DNA模板直接加入10uL的无细胞转录翻译系统,抗菌肽即在4小时内产生。为测试抗菌肽的活性,研究者将4uL的无细胞合成体系混合物分别于大肠杆菌(革兰氏阴性菌)和枯草杆菌(革兰氏阳性菌)混合,并持续监测混合之后20小时内的菌液OD600的变化情况。
他们首先验证了两个已知的抗菌肽BP100和Cecropin B,随后在五轮实验中筛选了500个候选抗菌肽,得到了30个有效的从头合成抗菌肽。这五轮实验的成功率从0%提高到12.7%。因为翻译起始效率(TIR)会影响蛋白的产量,所以他们还计算了所有设计的序列的TIR,以确认他们筛选方法对翻译起始效率低的序列没有偏,结果显示500条候选肽和30条功能肽间的TIR没有显著性差异。
他们对从头设计的序列进行了进一步的分析。Alphafold预测显示30条序列中,有27条序列形成了螺旋结构。而且,不同的VAE设计的序列显示出不同的结构多样性(图2b)。抗菌肽的主要特征之一是两亲性,这是由抗菌肽核心中阳离子和疏水氨基酸交替产生的。天然抗菌肽主要以脂肪族氨基酸为主,而他们的新抗菌肽的疏水核心则主要是芳香族的。尽管训练肽和生成肽,生成肽和优选肽中存在这样的性质差异,但是测试肽和有效肽之间则不存在这种差异。
接着,研究者们使用分子动力学(MD)模拟来研究他们的AMPs与带负电荷的细菌内膜(IM)和人质膜(PM)模型的相互作用。根据他们的模拟,所有AMPs与IM界面的结合都比与PM的结合强得多,这主要是由于这些阳离子AMPs与带负电荷的细菌膜的相互作用。
研究者们还化学合成了有效的抗菌肽,并表征了它们的最低抑制浓度(MIC),以及溶血(HC50)和细胞毒性(CC50)。30条抗菌肽中,他们成功合成22条,其中20条对枯草芽孢杆菌的MIC≤6 μM, 15条对大肠杆菌的MIC≤25 μM。大多数抗菌肽的HC50和CC50都很高,15条对新鲜人红细胞的HC50 > 100 μM(13个> 250 μM), 13条对HCT116人结肠细胞的CC50 > 100 μM(6个> 250 μM)。
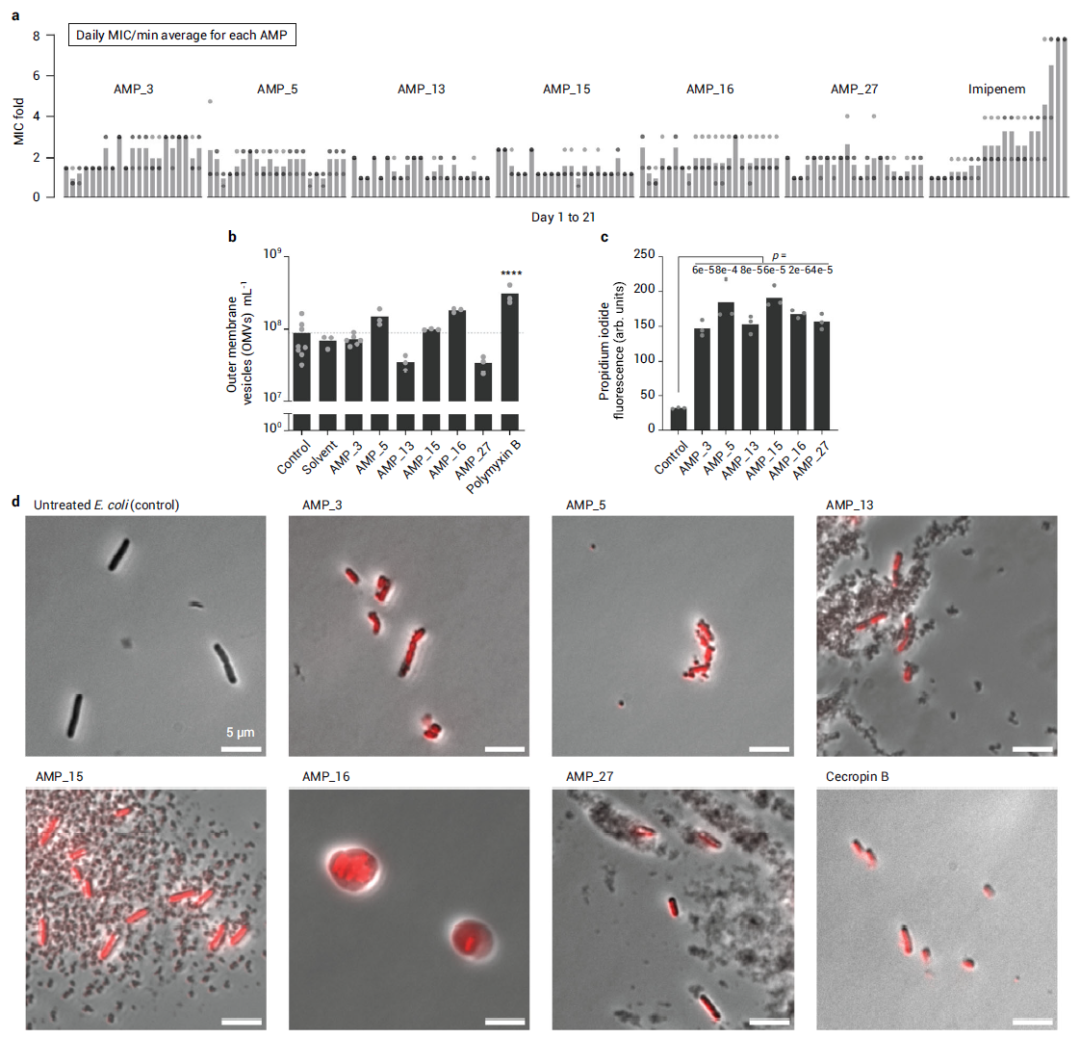
研究者们还对抗菌肽进行了光谱活性和耐药性进行了表征。通过连续传代实验,研究者发现,在21天的连续传代中,抗菌肽的MIC没有显著增加,而亚胺培南MIC逐渐增加至8倍,超过了EUCAST定义的临床相关耐药的敏感性断点(图3a)。作者还通过碘化丙啶染色和显微镜观察对这一结果进行了验证(图3c-d)。
最后,作者还研究了抗菌肽处理对大肠杆菌外膜囊泡(OMVs)释放的影响。OMV由革兰氏阴性菌自然释放,可作为对抗表面攻击剂的反应,中和膜靶向抗生素的作用。结果显示,与未经处理的大肠杆菌相比,六种广谱抗菌肽都没有显著增加OMV的释放(图5b)。这些实验表明,作者发现的的六种光谱抗菌肽能够逃避细菌的耐药性发展和自卫反应。
综上,在本研究中作者介绍了30条从头设计抗菌肽的设计和验证过程,并发现其中6条在体外具有光谱的抗菌活性。相比于其他深度学习方法发现和设计的抗菌肽验证所需的高成本和长周期,作者发展了无细胞蛋白质合成系统(CFPS)大大加速了抗菌肽开发中的设计-构建-测试周期。而作者将CFPS和深度学习相结合的做法,也显示出以更高的速度和效率探索多肽序列空间的潜力。这将有望在未来应用于更多基于多肽的候选药物的发现和开发。
Pandi, A., Adam, D., Zare, A. et al. Cell-free biosynthesis combined with deep learning accelerates de novo-development of antimicrobial peptides. Nat Commun 14, 7197 (2023). https://doi.org/10.1038/s41467-023-42434-9
点击左下角的"阅读原文"即可查看原文章。

内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢