
关键词:酶反应网络,复杂网络,动力学建模
论文题目:Iterative design of training data to control intricate enzymatic reaction networks论文期刊:Nature Communications
论文链接:https://www.nature.com/articles/s41467-024-45886-9
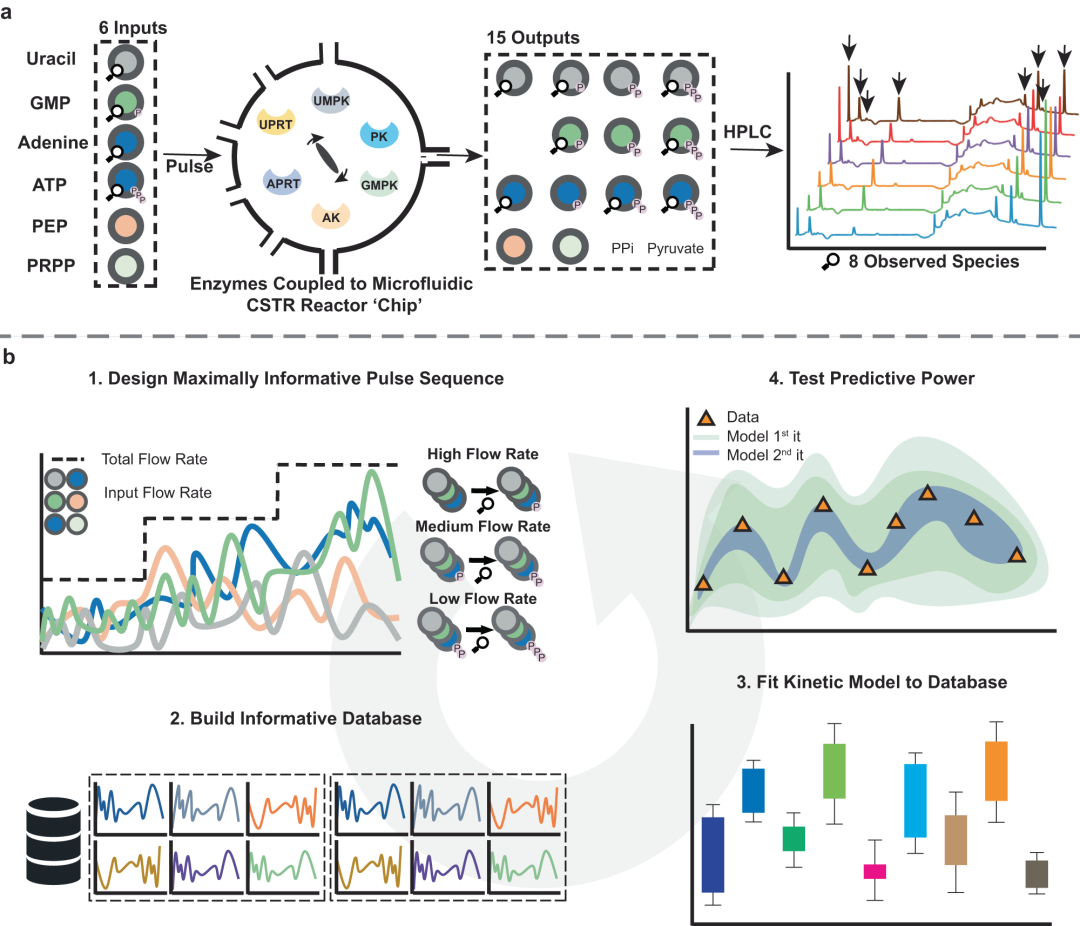
酶反应网络(ERN)是一组在实验室条件下重构的酶催化反应,用于模拟生物细胞内的代谢途径。体外酶反应网络的动力学建模,对于理解和控制内部非线性相互作用产生的复杂行为至关重要。在当前的研究中,科学家们面临着如何有效控制复杂酶反应网络的挑战。尽管近年来在体外重构酶反应网络以模拟细胞功能或生产高价值化学品方面,已经取得了显著进展,但大多数网络未能捕捉到生物酶反应网络的复杂性特征:多个相互连接的子路径同时运作,从而从多个输入生成多个输出。目前难点在于缺乏足够的实验数据集,来训练能够追踪大型酶反应网络动态特性的动力学模型,从而实现按需设计。最新发表于Nature Communications的一项研究,提出了一种迭代设计训练数据的方法,通过在每个优化周期中添加新的、更具信息量的实验来训练动力学模型。这种方法结合了类似主动学习的方法和流动化学,通过最大化信息量的非平衡扰动序列,优化实验设计(OED)算法。基于此,就可以通过一系列有限次的精心设计的实验,收集到高质量的数据,这些数据足以帮助我们理解和描绘出酶反应网络中发生的各种复杂反应动力学,并精确控制酶反应网络(如同操作一个多输入多输出的系统)。研究者在实验室创建了一个化学反应网络,模拟生物体自然的核苷酸回收途径。该途径是通过从RNA和DNA的降解中回收碱基和核苷,再生核苷酸以支持细胞的过程。研究者通过将酶反应网络的反应转化为粗粒度的常微分方程(ODE)模型,就能够追踪所有中间体的时间变化。然而,随着酶反应网络复杂性和规模的增加,选择一组扰动来直接观察和理解这些反应如何发生和相互作用,就变得越来越困难。通过这项研究提出的方法,不仅展示了有效控制酶反应网络的可能性,而且还强调了在化学反应网络控制和优化方面,机器学习和动力学模型的结合使用所具有的巨大潜力。图1 用于训练动力学模型的实验流程设置和训练数据迭代设计的概述

了解读书会具体规则、报名读书会请点击下方文章:
生命复杂性系列读书会:从信息和物理视角探索生命的内在逻辑
点击“阅读原文”,报名读书会
内容中包含的图片若涉及版权问题,请及时与我们联系删除







评论
沙发等你来抢