今天为大家介绍的是来自Robert Hoehndorf团队的一篇论文。基因本体论(Gene Ontology, GO)是一个包含超过100,000条公理化理论,这些公理描述了蛋白质在三个子本体中的分子功能、生物过程和细胞位置。利用GO预测蛋白质的功能既需要学习能力也需要推理能力,以保持一致性并利用GO中的背景知识。虽然已经开发了许多方法来自动预测蛋白质的功能,但有效利用GO中的所有公理进行知识增强学习仍然是一个挑战。

蛋白质的功能是通过基因本体论(GO)来描述的,GO是生物学中最成功的本体论之一,包含了描述单个蛋白质的分子功能(MFO)、蛋白质可以贡献的生物过程(BPO)以及蛋白质活跃的细胞组分(CCO)的三个子本体。研究人员基于实验确定蛋白质的功能,并生成科学报告,这些报告随后由数据库管理员采集并添加到知识库中。这些注释通常会被传播到同源蛋白质上。因此,UniProtKB/Swiss-Prot数据库包含了成千上万种生物和超过550,000个蛋白质的手工策划GO注释。蛋白质功能预测方法依赖于不同的信息源。这些方法可能使用序列域注释,还可能通过知识图嵌入结合蛋白质-蛋白质相互作用。此外,应用于科学文献的自然语言模型在自动功能预测方面也取得了成功。
许多功能预测方法的一个主要局限性是它们依赖于序列相似性来预测功能。虽然这种方法对于具有已知功能的相似蛋白质有效,但对于与已知功能域序列相似性较小或没有相似性的蛋白质来说可靠性较低。分子功能大多来源于结构,具有相似结构的蛋白质可能具有不同的序列。重要的是,具有相似序列的蛋白质根据它们的活性位点和所在的生物体,可以具有不同的功能集合。因此,对所有三个GO子本体使用相同信息源的方法是有限的;虽然可以通过蛋白质序列或结构预测MFO子本体的功能,但BPO和CCO的功能在很大程度上依赖于多个蛋白质的存在和以特定方式相互作用;因此,预测BPO和CCO注释需要与预测MFO注释不同的信息源。
本体论是预测蛋白质功能中很少利用的另一信息源。本体论不仅仅是类的集合;它们是使用基于逻辑的语言明确某些类意图含义的正式理论。GO的公理中包含的背景知识可以被一些机器学习模型利用,通过知识增强的机器学习来改进预测。通过将正式公理纳入机器学习模型,就有可能在学习或预测过程中利用先验知识,对参数搜索空间施加限制,这可以提高学习过程的准确性和效率,最终做出更好的预测。只有少数功能预测方法利用GO中的正式公理。例如GoStruct、DeepGO、DeePred、SPROF-GO和TALE等预测蛋白质功能的层级分类方法利用了包含级别关系的下位公理,但忽略了GO中其他可以用来减少搜索空间和改进预测的公理。因此作者开发了DeepGO-SE,这是一种蛋白质功能预测方法,它使用预训练的大型蛋白质语言模型结合一个神经符号模型,通过近似语义蕴含来预测功能。作者使用ESM2蛋白质语言模型生成单个蛋白质的表示。类似于DeepGOZero,作者将ESM2嵌入投影到由GO的公理生成的嵌入空间(ELEmbeddings)中。ELEmbeddings基于几何形状和几何关系对本体论公理进行编码(作者称为世界模型),在这个模型中可以确定语句的真假。与DeepGOZero不同的是,作者使用这些世界模型来执行“语义蕴含”:如果在所有理论T成立的世界模型中,某个声明ϕ都是真的,那么我们就说理论T蕴含了声明ϕ。虽然一般来说,对于理论T或语句ϕ有无限多这样的世界模型,作者的模型学习有限多个这样的模型,并生成作为“近似”语义蕴含的功能预测,在生成的每一个世界模型中测试。使用这种形式的近似语义蕴含,作者展示了GO扩展版本中的公理如何增强预测性能。
模型架构
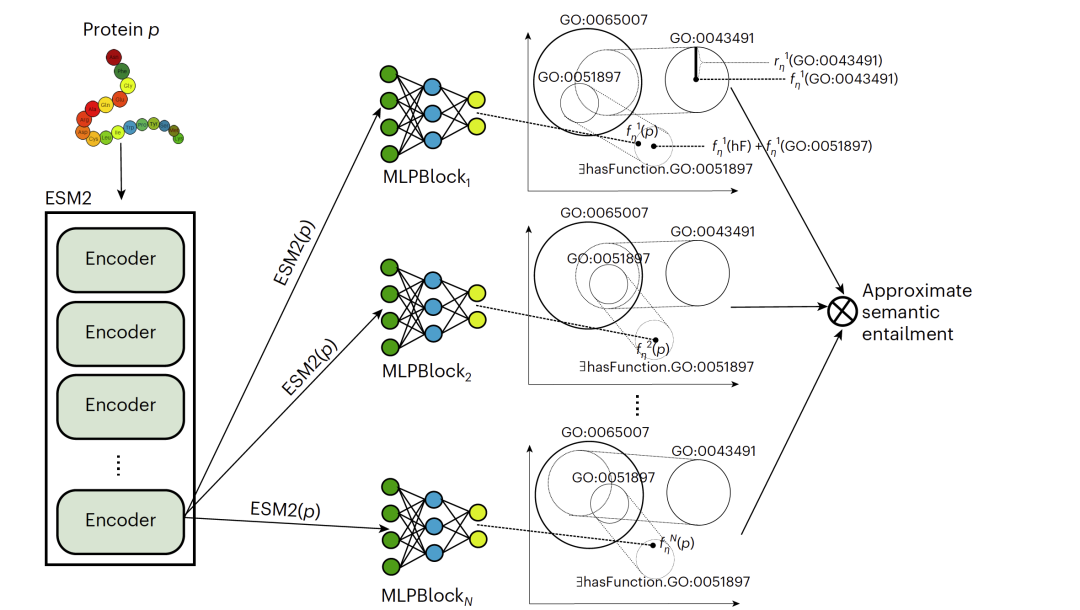
图 1
DeepGO-SE模型通过近似语义蕴含来实现知识增强学习。它的执行分为三个步骤:
生成近似模型:
首先基于逻辑理论生成一个近似模型ℐ,这个逻辑理论由GO中的背景知识(即公理)和关于蛋白质的一组断言(如“蛋白质具有功能C”的声明)构成。
这里使用的ELEmbeddings是根据GO的公理来生成的,旨在捕捉这些公理在几何空间中的表示。
表示蛋白质并优化目标:
然后,作者用ESM2嵌入来表示蛋白质,并将这些表示作为近似模型ℐ中的实例,使得声明“蛋白质具有功能C”在ℐ中为真的可能性最大化,作为一个优化目标。
生成多个近似模型并近似蕴含:
最后重复上述过程,生成k个逻辑理论的近似模型ℐ1, … , ℐk;
蕴含被定义为在所有模型中为真。
通过这k个模型进行近似蕴含。
为了计算蕴含,作者聚合了所有生成模型中“蛋白质具有功能C”的声明的真值。
图1展示了这个过程。
UniProtKB/Swiss-Prot数据集结果评估
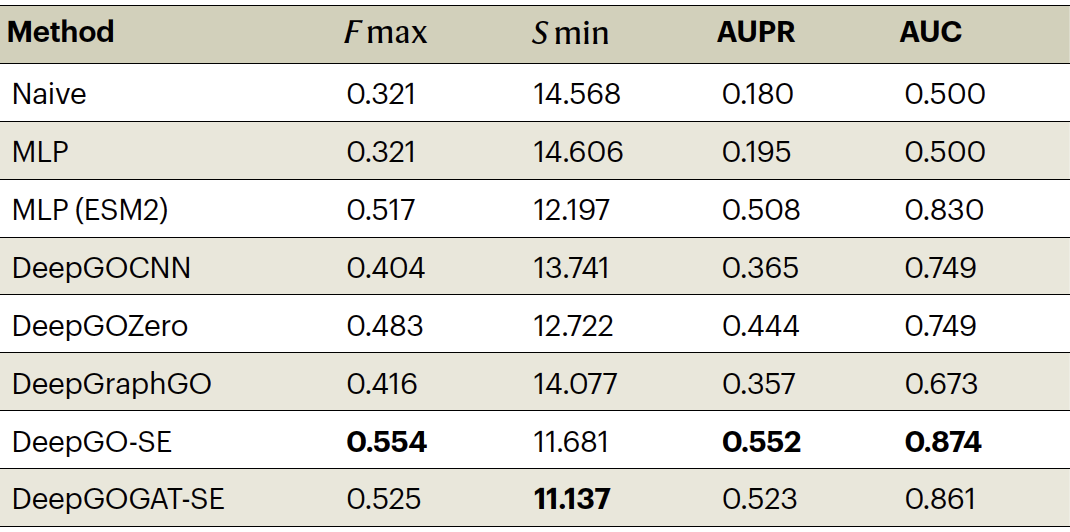
表 1
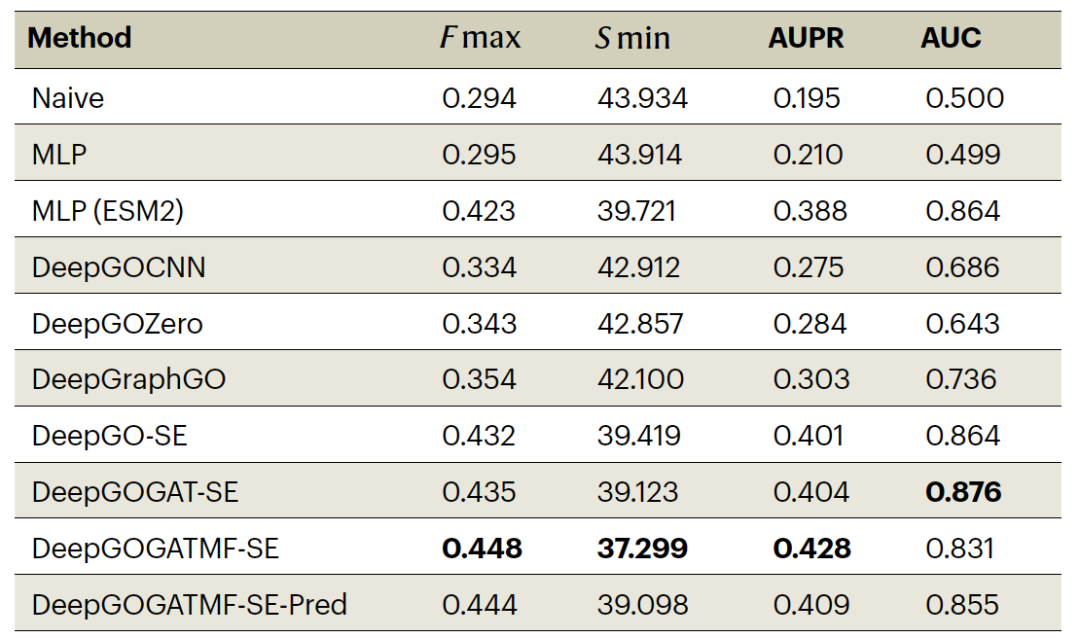
表 2

表 3
在UniProtKB/Swiss-Prot数据集评估中,作者通过基于序列相似度分割的UniProtKB/Swiss-Prot数据集,将作者的方法与基准方法进行了比较和评估。作者分别训练和评估GO的三个子本体,因为它们在类别数量及其关系、蛋白质数量以及它们可以从中受益的信息源方面有不同的特点。与五种基准方法进行了比较。这些方法中没有一个依赖于序列相似度,并且,除了Naïve方法外,所有方法都是基于直接学习或使用从InterProScan等工具派生的特征的序列特征来分配功能。在所有评估中,DeepGO-SE模型在F max、AUPR和AUC方面显著优于所有基准方法。在MFO评估中,DeepGO-SE达到了0.554的F max,比MLP和DeepGOZero方法的结果高出7%(表1)。在预测BPO注释时,该模型的F max达到了0.432,比最佳基准方法DeepGraphGO高出约8%(表2),而在CCO评估中,DeepGO-SE模型的F max达到了0.721(表3)。
在基础DeepGO-SE模型中,蛋白质嵌入是通过ESM2从蛋白质序列生成的;然而可以修改蛋白质嵌入来编码更多关于蛋白质的信息。作者认为,仅从蛋白质序列无法预测生物过程和细胞组分注释,因为即使是序列相同的蛋白质也可能因为其他蛋白质的存在或缺失而合理地参与不同的过程。因此,作者使用蛋白质嵌入也编码关于蛋白质组及其相互作用(蛋白质-蛋白质相互作用,PPIs)的信息。作者使用这个嵌入函数并改变输入向量到DeepGO-SE来进行三个实验。首先,在DeepGOGAT-SE中,作者使用ESM2嵌入作为每个蛋白质的输入。其次,在DeepGOGATMF-SE中,输入包括使用大小为6,851的二进制向量对蛋白质进行其分子功能的实验注释。第三,在DeepGOGATMF-SE-Pred中,使用DeepGO-SE模型对分子功能的预测得分作为输入。作者训练并评估这三个模型,以确定纳入相互作用的效果。在DeepGOGAT-SE模型中结合PPIs和ESM2嵌入降低了MFO预测性能到一个F max的0.525,但稍微改善了S min。纳入PPIs在BPO预测中提高了性能,到一个F max的0.435。当使用实验MFO注释作为特征时(DeepGOGATMF-SE),BPO的总体最佳性能被实现,其次是由DeepGO-SE预测的MFO注释(DeepGOGATMF-SE-Pred)(表2)。对于CCO,在DeepGO-SE模型中纳入PPIs将F max从0.721提高到0.736(DeepGOGAT-SE)(表3)。
编译 | 曾全晨
审稿 | 王建民
参考资料
Kulmanov, M., Guzmán-Vega, F.J., Duek Roggli, P. et al. Protein function prediction as approximate semantic entailment. Nat Mach Intell 6, 220–228 (2024).
https://doi.org/10.1038/s42256-024-00795-w
内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢