今天为大家介绍的是来自华南理工大学崔巍团队的一篇论文。计算机辅助的启动子设计是合成启动子工程中的一大发展趋势。各种深度学习模型已被用来评估或筛选合成启动子,但关于从头设计启动子的研究还很少。为了探索生成模型在启动子设计中的潜力,作者在大肠杆菌中建立了一个基于扩散的生成模型。该模型完全由序列数据驱动,能够研究自然启动子的基本特征,从而生成在结构和组分上与自然启动子相似的合成启动子。作者还改进了FID指标的计算方法,使用卷积层来提取启动子序列的特征矩阵。得到的合成启动子的FID为1.37,这意味着合成启动子的分布与自然启动子类似。作者的工作为从头设计启动子提供了一种新的方法,这表明一个完全数据驱动的生成模型对于启动子设计是可行的。

具有稳健和可预测活动的调控序列被加入合成流程中以满足所需的表达水平和模块化工程。然而,通常选择的调控序列面临着基因表达范围有限、多样性不足和可用成分少的困难。设计具有所需基因表达水平的合成调控序列有望解决这些问题,而转录的关键调控元件——合成启动子的设计已经引起了合成生物学领域的广泛关注。合成启动子的设计方法可分为人工诱变、杂交启动子工程以及计算方法。
人工突变包括侧翼区域的饱和突变(SMFR)和易错PCR(epPCR)。SMFR通过强制侧翼区域发生突变来改变启动子活性,而不改变已知的共识基序,但它过度依赖设计者对启动子结构的先验知识,使其难以工业化。epPCR在共识区和间隔区引入突变,以获得许多新颖的启动子变体,这种方法已广泛用于构建具有广泛基因表达水平的合成启动子库。然而,通过视觉选择菌落所消耗的资源不容小觑。另一种突变技术是杂交启动子工程,通过杂交现有的启动子生成合成启动子。根据以往的研究结果,杂交启动子工程在性能改进方面是有效的,但存在输出低、复杂性高和上下文依赖性的问题。
与上述方法相比,计算方法可以在广阔的潜在序列空间中有效寻找新颖的启动子。在GAN的生成器训练过程中可能发生模式崩溃,其中生成器可能收敛到少数几个主导模式,导致生成样本的多样性和丰富性丧失。当应用于实际场景时,多样性低的合成启动子容易与类似序列发生同源重组,导致基因组不稳定,影响正常的细胞功能。与通过对抗训练优化生成器的GAN不同,扩散模型通过逐步去噪方法生成复杂的目标分布,从而避免了由对抗训练不稳定性引起的模式崩溃问题。为了探索扩散模型在合成启动子设计中的适用性并建立一种全新的从头设计启动子的方法,作者建立了一个基于扩散的生成模型来进行从头启动子设计,该模型在训练过程中很少遭受模式崩溃的问题。并且,生成的启动子能够捕捉到天然启动子的关键特征,如-10和-35基序、GC含量和k-mer频率。作者提出了可以作为设计合成启动子的新策略,并进行了一些体外实验来支持这一点。
模型部分
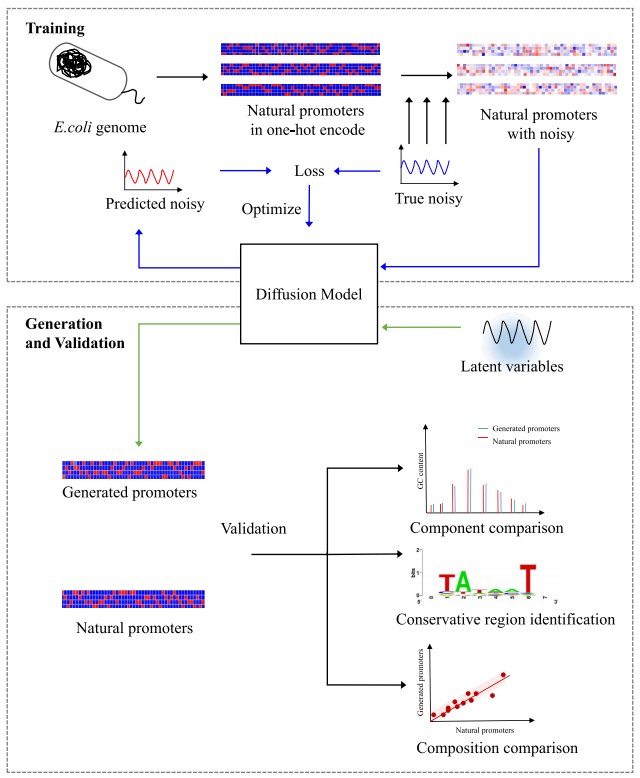
图 1
在合成启动子序列的实验设计中使用了三个数据集。第一个数据集由Thomason等人发布,包含了大肠杆菌K12 MG1655基因组中共14,098个实验鉴定的启动子,启动子序列定义为转录起始位点上游50个碱基对。第二个数据集由Johns等人发布,包含了来自184个原核生物基因组的29,249个调节序列,调节序列定义为基因起始密码子上游165个碱基对。该调节库被克隆到p15A载体中,并转化到大肠杆菌MG1655中。第三个数据集由Lubliner等人发布,包含了来自酿酒酵母中获得的7536个核心启动子序列,这个数据集构建的调节库被转化到酵母细胞(Y8205菌株)中。这三个数据集将分别用来训练三个扩散模型,用于后续实验。
扩散模型的构建主要包含两个阶段:在预定义的前向扩散过程中,高斯噪声逐渐加入到自然启动子序列中,直到完全被噪声取代。接下来的去噪扩散过程中,神经网络被训练以减少噪声并恢复自然启动子序列。在该模型中,神经网络基于U-Net网络,因为它在编码器和解码器之间引入了残差连接,这大大改善了梯度流。U-Net的所有构建块包括网络助手、位置嵌入、ResNet块、注意力模块和组归一化。在这项工作中,ResNet块是U-Net的核心构建块,它是一个更深层次的残差学习框架。
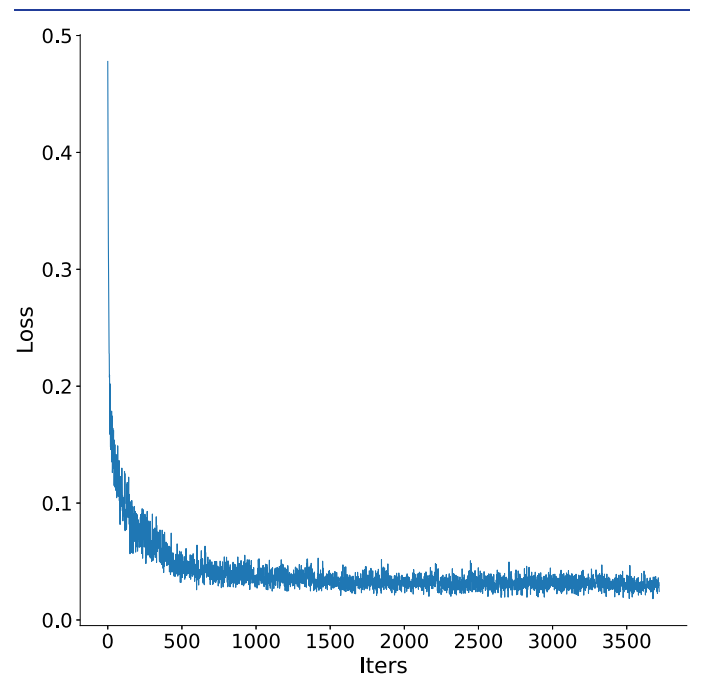
图 2
对于训练过程,启动子序列通过单热编码导入模型,然后处理到[-1, 1]的范围内,确保从标准正态先验开始,反向过程操作在一致的尺度输入上。损失函数是预测噪声和实际噪声之间的均方误差损失,优化器使用的是Adam优化器,初始学习率设为1×10-4,批处理大小设为64,大约在3000次优化迭代后损失收敛(见图2)。
扩散模型捕捉到了天然启动子的基本特征
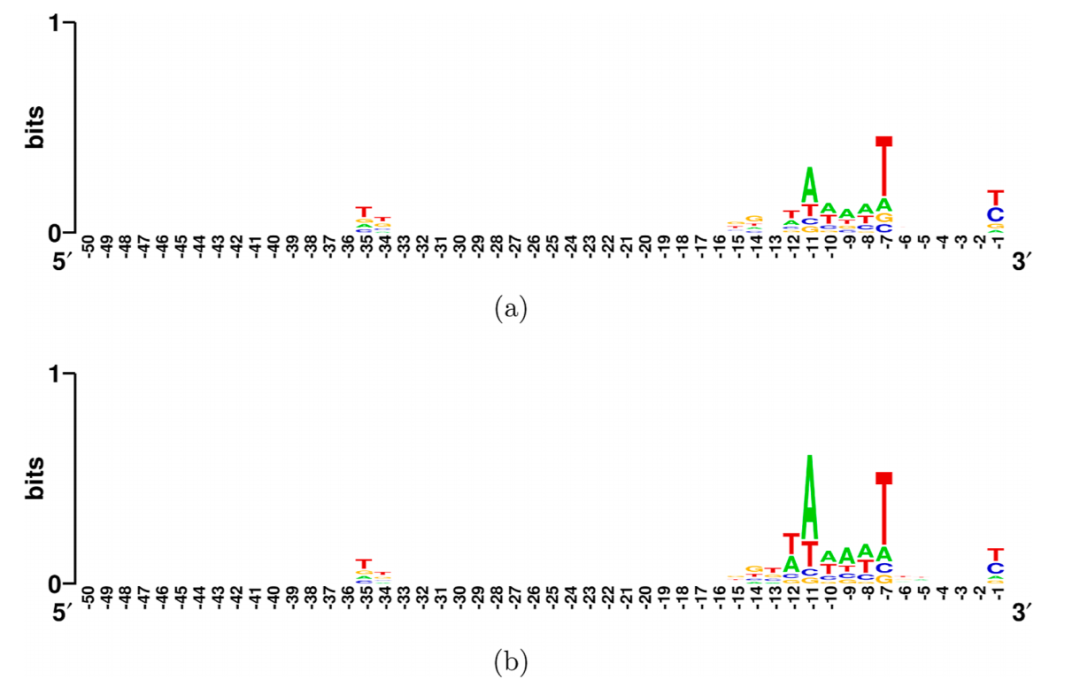
图 3
根据对原核启动子的先验信息,进行了几项外部验证实验,以全面评估合成启动子。首先,-10和-35区域是原核启动子中最重要的保守区域,其结构和间距影响启动子的转录活性。-10区域是一个富含AT碱基的保守序列(TATAAT),位于转录起始位点(TSS)上游大约10个碱基对;-35区域位于-10区域上游16至19个碱基对处,这是RNA聚合酶与启动子之间的结合位点。如序列标志所示(见图3),合成启动子中包含-10和-35区域,这意味着扩散模型能部分学习自然启动子的保守序列特征。此外,本工作严格遵守了保守序列的间距条件,结果大约为18个碱基对。
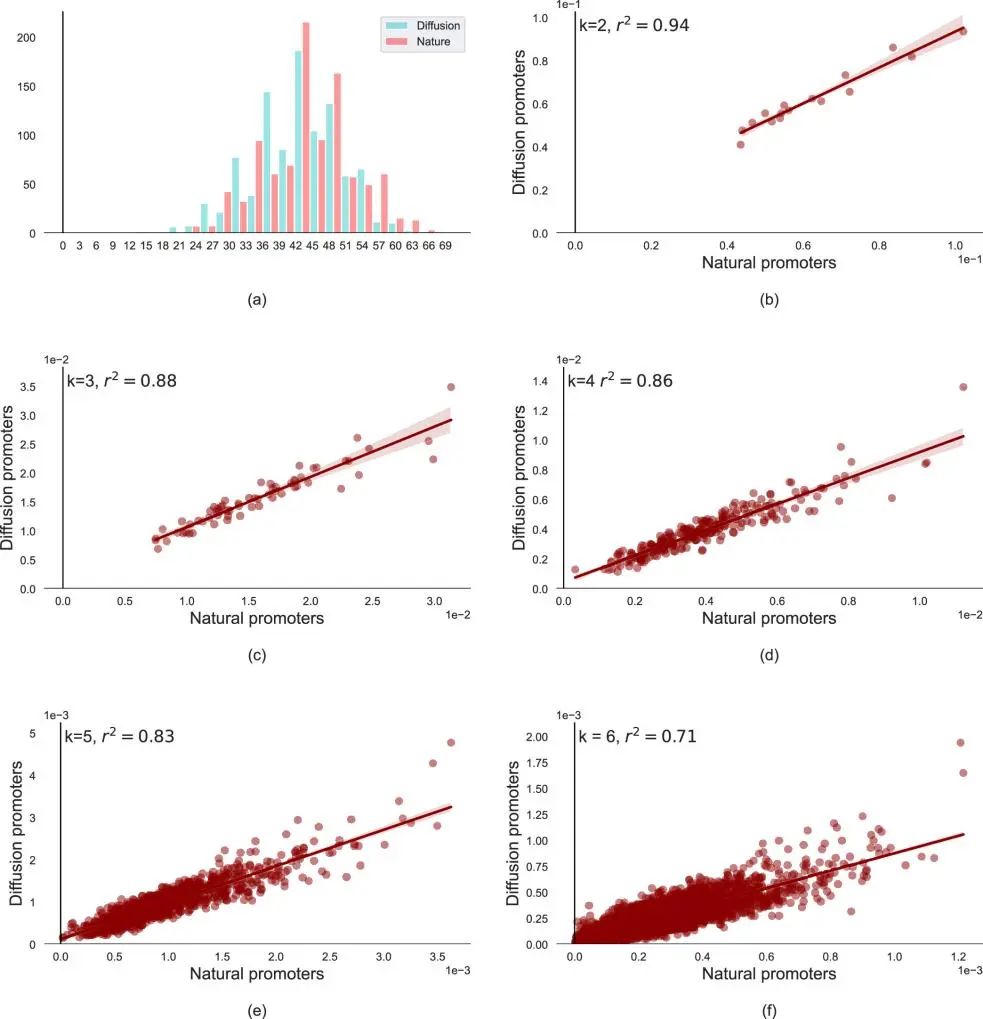
图 4
在分子生物学和遗传学中,GC含量是指基因片段中属于鸟嘌呤(G)或胞嘧啶(C)的碱基所占的百分比。原核生物的生长活性与GC含量密切相关,这也是细菌分类中的一个重要考虑因素。为了比较合成启动子与自然启动子之间组分的相似性,作者计算了1000个随机选取的合成启动子和1000个自然启动子的GC含量,并发现GC含量分布非常相似(见图4a),所有数据都集中在30%到60%的范围内,其中45%的GC含量的启动子序列最为常见。
在生物信息学中,k-mer是指生物序列中长度为k的子串。它们已被用于翻译效率控制、细菌属的区分、物种的DNA条形码以及其他领域。作者计算了一些常见k-mer的频率(见图4)以及合成启动子和自然启动子之间k-mer频率的皮尔逊相关系数的平方(R2MF)。R2MF越高,表明合成启动子的分布越接近自然启动子,这表明合成启动子的质量越高。如图4所示,尽管随着k的增加,相关性有所下降,但仍保持在0.8的较高水平。
生成的启动子展现出令人满意的质量和多样性
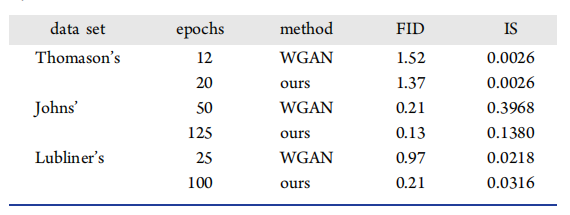
表 1
Wang等人使用WGAN模型从头设计启动子,并进行了体内验证实验,这是生成模型在合成启动子中的首次应用。为了比较,作者计算了该扩散模型和WGAN模型的FID与IS。作者对WGAN模型进行了160个训练周期,对该扩散模型进行了130个周期的训练。根据他们的工作显示的先前结果,WGAN模型在12个周期内表现最佳。作者收集了那些迭代产生的序列,并从120个周期中采样序列,以分析WGAN模型训练过程中是否发生了模式崩溃。与WGAN模型不同,扩散模型在训练过程中很少发生模式崩溃。然后,从最佳周期中选择了1000个生成的序列进行FID计算和与WGAN模型的比较(见表1)。FID和IS越小,表明合成的分布与自然的分布越接近。
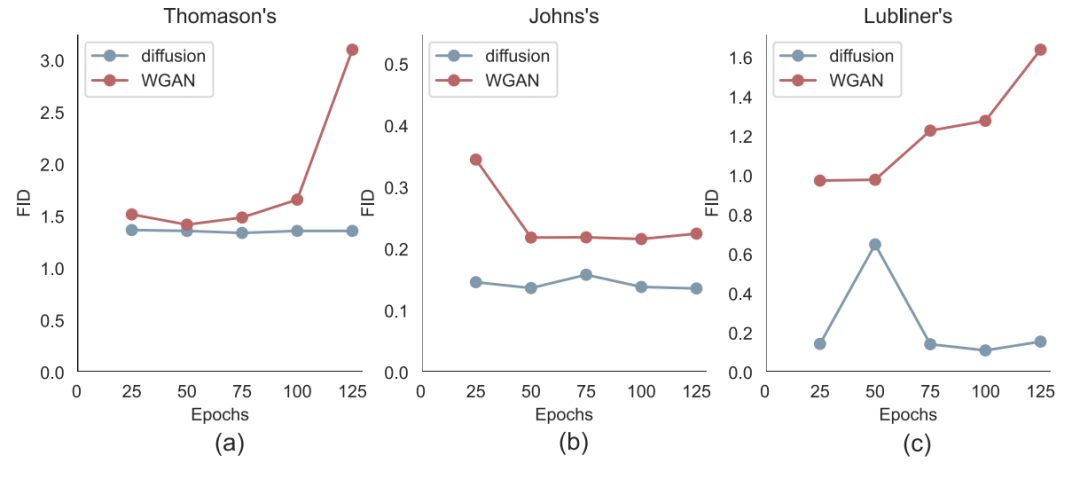
图 5
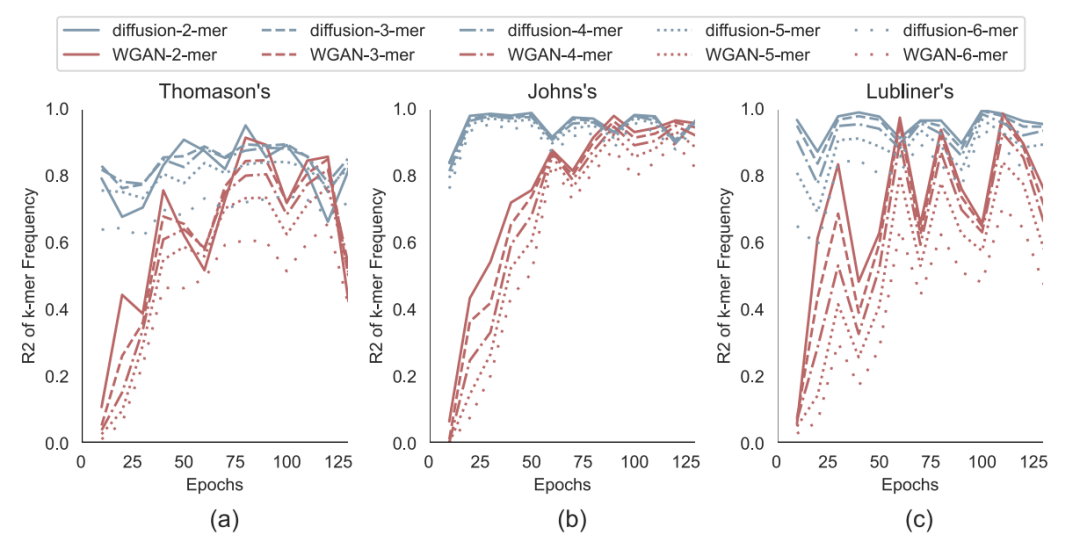
图 6
作者绘制了WGAN和扩散模型在不同训练周期的R2MF曲线。从图6中可以看出,两种模型的R2MF性能在所有周期中都相对稳定,持续保持在较高水平(>0.7)。相比之下,WGAN模型的R2MF随着周期的变化而显著波动,其性能始终低于扩散模型。
此外,通过结合图5和图6的数据,作者发现尽管使用早停方法(训练周期<50)有效避免了WGAN中的模式崩溃问题,但在这一阶段WGAN模型的R2MF仍然保持在较低水平(<0.5),这表明WGAN模型无法同时兼顾生成序列的多样性与其与自然序列的相似性。相比之下,扩散模型能够同时保持良好的FID性能和高R2MF。这进一步强调了扩散模型在训练稳定性和合成启动子质量方面的优势。
编译 | 于洲
审稿 | 王建民
参考资料
Lin J, Wang X, Liu T, et al. Diffusion-Based Generative Network for de Novo Synthetic Promoter Design[J]. ACS Synthetic Biology, 2024.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢