精准的评估蛋白质-配体相互作用对药物发现至关重要。然而,开发可靠的评估方法一直是学术界和工业界的长期挑战。近年来,人工智能技术在该领域已经取得了显著进展,以AlphaFold为代表的深度学习方法在蛋白质三维结构,蛋白质-配体复合物结构预测方面表现卓越。但是,在新靶标的药物虚拟筛选场景中,高精度的活性预测评分方法仍然非常匮乏。许多研究表明,深度学习模型倾向学习数据中的分布偏差,对分布内的数据可以给出很好的性能指标。但在实际应用中,尤其面对训练集未见的新靶标和化学多样性空间,却无法展现出良好的泛化效果。
最近,中国科学院上海药物研究所郑明月团队提出了一种通用蛋白质-配体相互作用评分方法EquiScore,用于解决新靶标的药物虚拟筛选问题。该方法利用等变图神经网络来整合蛋白质-配体相互作用相关的物理先验知识,并且使用多种数据增强、数据去冗余策略来避免模型过拟合潜在的数据分布偏差。在药物虚拟筛选场景和先导化合物优化场景中,EquiScore对训练集未见的新靶标表现出了良好的泛化性能。当EquiScore与不同的分子对接或复合物结构预测方法联合使用,均可显著增强这些方法的虚拟筛选能力。此外,EquiScore还能够通过注意力权重分析来捕获关键的分子间相互作用,从而为合理的药物设计提供有价值的线索。相关研究论文 “Generic protein–ligand interaction scoring by integrating physical prior knowledge and data augmentation modelling” 于2024年6月6日在Nature Machine Intelligence在线发表。

1 背景
随着蛋白-配体相互作用数据的爆炸式增长,基于机器学习的相互作用评分取得了显著进展。通过采用不同的机器学习算法和神经网络架构,这类数据驱动的方法在虚拟筛选和亲和力预测的基准测试上显示出优势。然而,最近研究发现,基于机器学习的蛋白-配体相互作用评分方法对新靶标体系难以实现良好的泛化,甚至无法超越一些传统的评分方法(如Schrödinger公司的Glide SP)。究其原因,一方面可能是机器学习模型复杂度提升,使其能够可能过拟合整个训练数据;另一方面,训练和测试数据集普遍存在的数据冗余和泄漏问题,可能导致我们高估了模型的真实性能。这些问题极大的限制了数据驱动的评分方法在实际药物研发场景中的应用潜力,尤其是针对新靶标的药物虚拟筛选。
除了数据因素,影响基于机器学习的评分方法性能的另一个关键因素是模型是否能够有效学习蛋白-配体相互作用的物理先验。许多工作在这个方向上进行了有益的尝试。例如,通过显式地添加损失项来引导模型学习,或对模型输入进行约束(只输入相互作用信息构建的图),或将物理公式引入到模型架构设计中来实现隐式的正则化。近年来,等变模型开始在预测分子间相互作用(如氢键相互作用)上展示出了良好的潜力。然而,对一些重要的相互作用,如芳香环系统之间的相互作用(如π-π相互作用),现有的神经网络架构中并不能进行很好的表征。对深度学习模型应用显式约束或隐式正则化可能会限制其固有的表达能力。因此,需要研究新的等变神经网络架构,使其能够更有效的结合物理先验知识与数据驱动建模,从而更好的学习蛋白-配体相互作用。
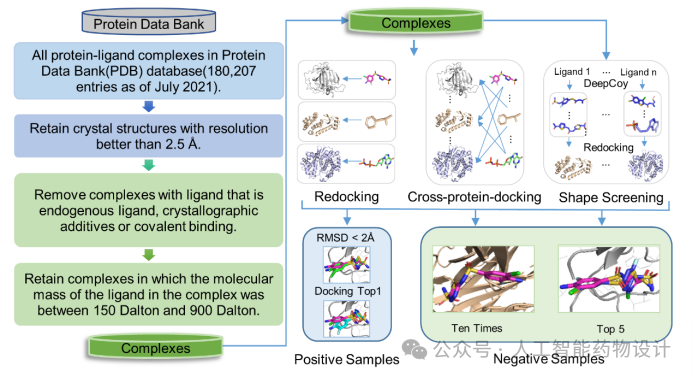
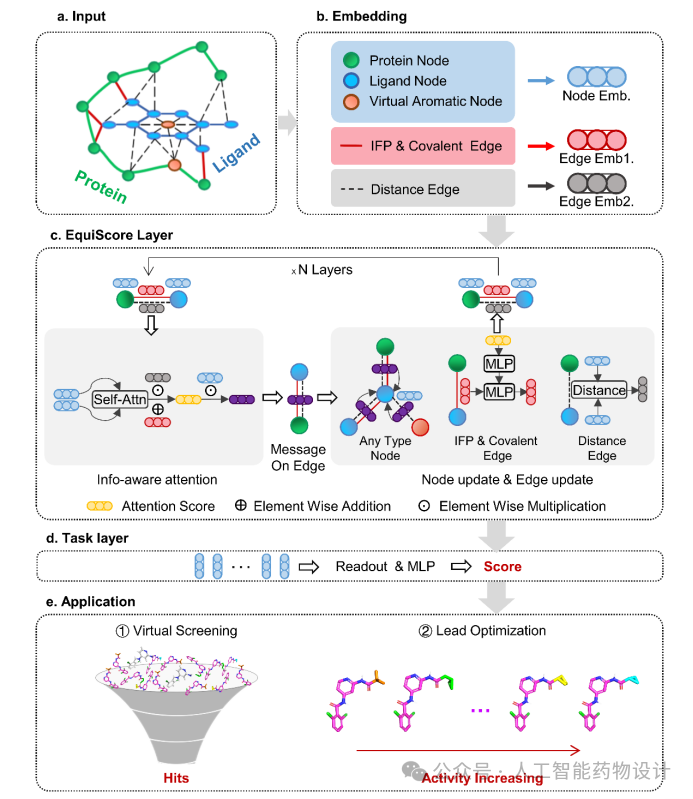
该研究主要从两个方面来提高深度学习评分函数对新蛋白的预测能力。首先,作者收集了更多的阳性样本,并使用重对接来生成更多样的阳性样本。同时,使用交叉蛋白对接,分子生成模型来生成更多具有欺骗性和多样性的诱饵分子,以减少构建训练数据集时可能出现的类似物偏差(analog bias)、数据分布偏差(data distribution bias)以及人工富集偏差(artificial enrichment bias)(见图1)。其次,作者提出了一种异质图构建流程(见图2),可以通过引入新的节点和边来整合分子间相互作用的物理先验信息。同时,作者提出了一种信息感知注意力机制,用于整合不同信息中的相互作用,这些信息包括包括:(1) 等变几何信息,(2) 化学结构信息,(3) 经验相互作用信息。
作者使用新构建的数据集和等变异质图网络来训练最终的评分模型,并命名为EquiScore。为了进行严格的评估,(1) 作者将EquiScore与最近新报道的深度学习评分方法在两个外部的虚拟筛选测试集(DUD-E和DEKOIS2.0)上进行比较,以评估其对新靶点的筛选能力; (2)并在外部的先导化合物优化数据集上比较EquiScore与其它方法对结构类似物的活性排序能力; (3) 使用不同的对接方法生成蛋白-配体复合物结合构象,进一步评价EquiScore作为评分方法的鲁棒性。最后,作者还分析了模型的可解释性。
2 结果与讨论
2.1 EquiScore 可以提升对新靶标蛋白的虚拟筛选能力
在训练集中未见过的蛋白质上的虚拟筛选能力能够更好的反映评分方法在实际应用中的泛化性能。为了进行充分的比较,作者选择了21种不同的评分方法作为基准。最近报道的所有方法都是基于PDBbind数据集训练得到的,该数据集与外部测试集具有高度的“软重叠”,即很多蛋白都是训练过程模型已经见过的。为了进一步检查这种数据泄漏是否会导致性能高估,作者将外部测试集涉及“软重叠”的数据进行了去重,并对所有方法的结果进行重新评估。
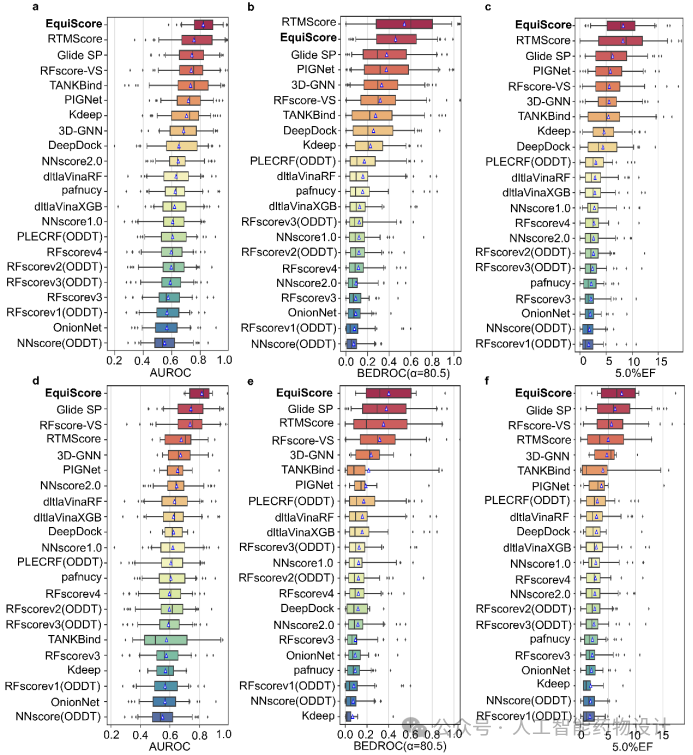
首先,作者验证了EquiScore作为一种评分方法的有效性,该方法使用薛定谔软件中的Glide SP模块生成的对接构象进行重打分。在DEKOIS2.0测试集上的分析结果显示(图3),EquiScore的AUROC得分最高,为0.821,显著高于第二名RTMscore的0.756(图3a)。为了比较不同方法在高排名分子中的活性化合物占比,作者也计算并比较了所有方法的BEDROC指标(图3b)。EquiScore优于RTMscore以外的所有基线模型。值得注意的是,当只考虑训练过程中未见的蛋白时,RTMScore的性能显著下降,从0.541(图3a)降至0.352(图3d),远低于EquiScore的0.401。在图3中,作者还观察到基于PDBbind训练的其他方法的性能均出现了同样的现象。以上结果表明,在严格的测试下,EquiScore的综合排序能力超过了现有的方法。此外,EquiScore对新蛋白的富集能力超过了传统评分方法和深度学习方法。
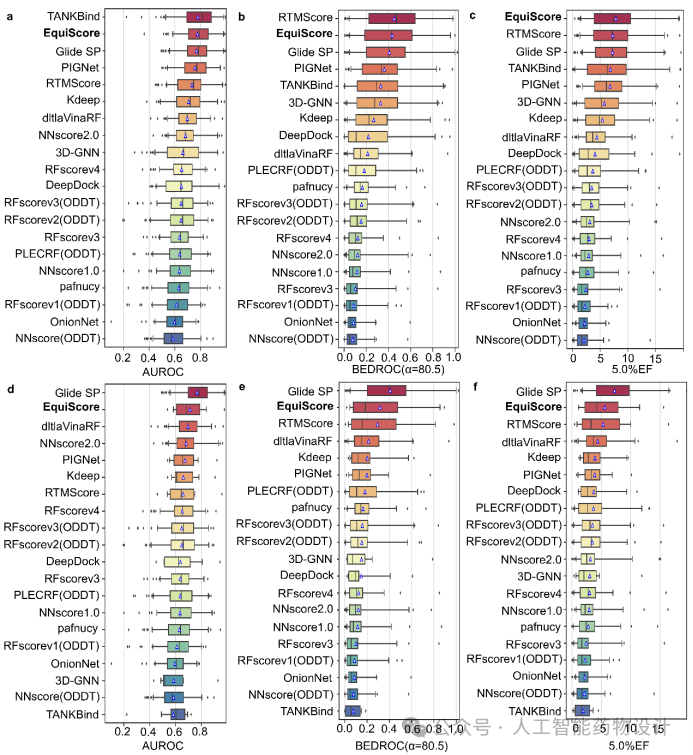
随后,作者将评估扩展到DUD-E数据集,该测试能够进一步验证EquiScore在更大规模虚拟筛选场景中的性能。总体而言,DUD-E结果与DEKOIS2.0结果一致。即使使用严格去重的训练数据,EquiScore的表现仍然可以在所有结果中排名前二。当只考虑新蛋白时,基于PDBbind训练的其他方法均表现出明显的性能下降。有趣的是,Glide SP使用更简单的函数形式和更少的参数却在去除重数据的DUD-E测试集上表现最好,EquiScore排名第二(图4d,e,f)。这一结果表明基于力场或经验函数的传统评分方法可能在训练集中未见的蛋白质体系上表现良好。然而,基于机器学习的方法可能会过拟合训练数据,从而对新体系缺乏泛化能力。
2.2 EquiScore 对结构类似物具有一定的排序能力
与虚拟筛选不同,先导化合物优化涉及具有相似结构或共同骨架的活性分子。在这种情况下,一个好的评分方法需要区分这些结构类似物之间活性的细微差异。目前,很少有方法可以同时展示良好的虚拟筛选和类似物排序能力。为了进一步验证EquiScore在先导化合物优化场景中的潜力,作者从文献中收集了一个外部数据集,其中包含8组类似物及其活性数据,用来测试EquiScore以及其他深度学习评分方法和传统方法的排名能力(结果见表1)。在这个测试集上,EquiScore(0.54)排名第二,仅次于FEP+(0.73),高于Glide SP。这一结果表明EquiScore显示了一定的区分类似物活性的能力,但它在性能方面与高精度的自由能计算方法还有一定差距。
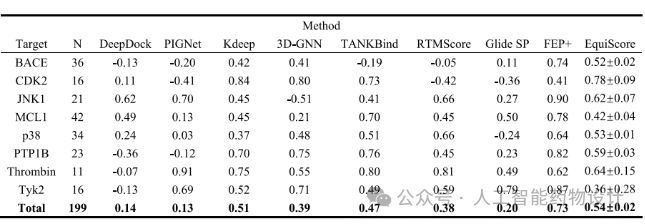
2.3 EquiScore展示出稳健的重打分能力
为了研究EquiScore的性能是否依赖于复合物结构的质量和生成方法,是否可以适配不同对接方法生成的对接构象,作者收集了不同对接软件(AutoDock Vina, GOLD CHEMPLP, Surflex-Dock, LeDock, Glide SP)在DEKOIS2.0上产生的对接构象,并使用EquiScore进行了重打分。结果显示,EquiScore显著提升了所有对接方法的虚拟筛选性能。尽管EquiScore是基于Glide SP生成的对接构象进行训练,但与不同对接方法结合使用时,EquiScore仍能有效增强这些对接方法的筛选能力,展现出了良好的通用性和稳健性,使其能够与各种分子对接方法进行集成。
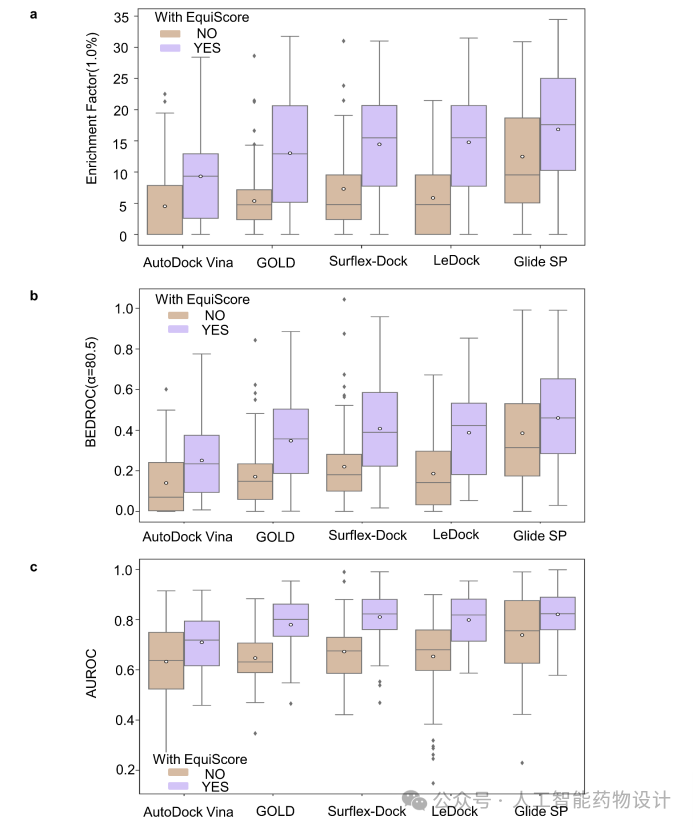
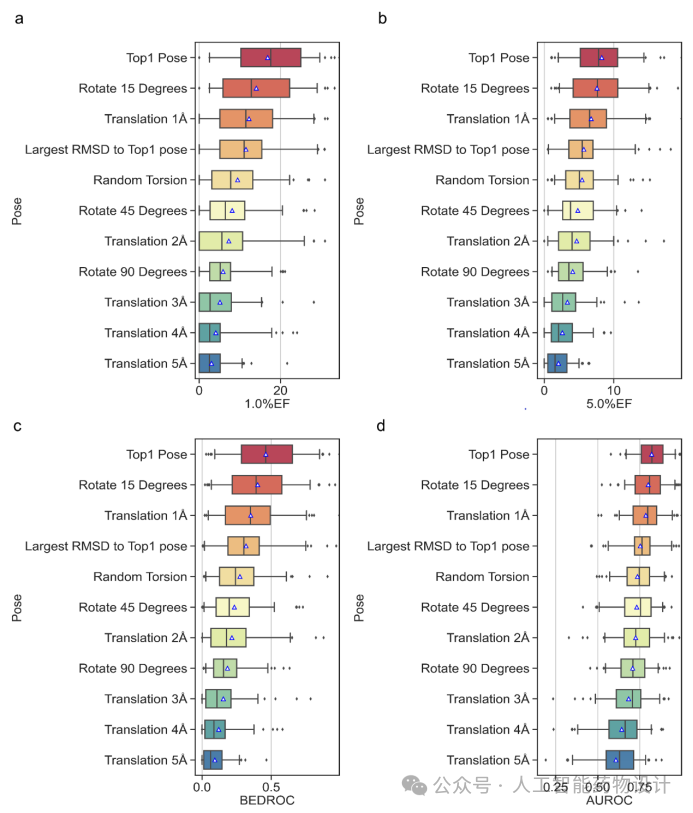
为了进一步研究构象扰动对EquiScore打分的影响,作者进行了更深入的分析。结果表明(图6),随着对构象扰动幅度的增大,EquiScore的性能逐渐下降,直至为零。这表明EquiScore的预测是依赖于蛋白-配体复合物的相互作用信息,而不是记住了数据分布偏差。
2.4 EquiScore可以解释构效关系
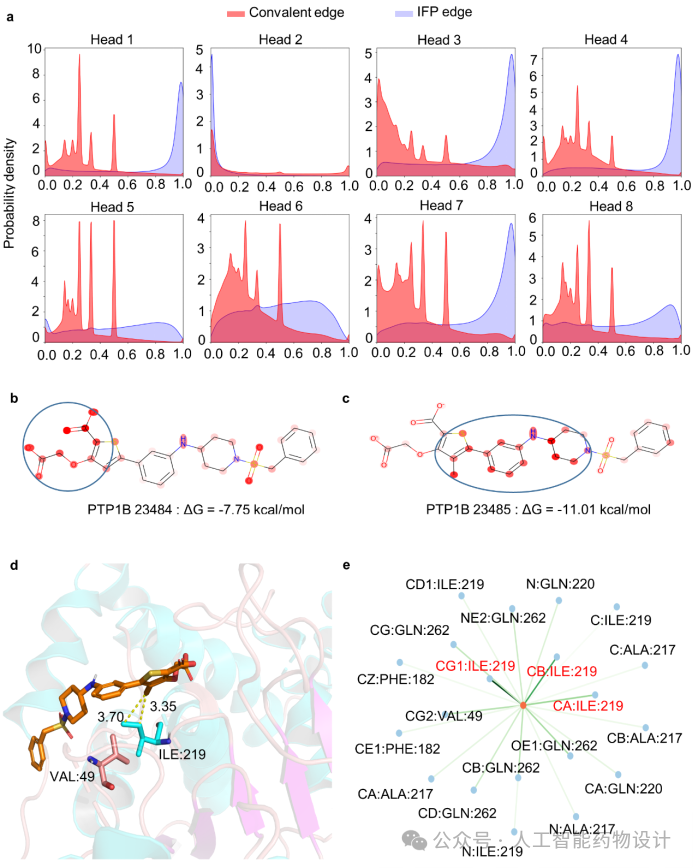
在注意力权重分布分析中,作者观察到不同注意力头在IFP边和共价边上的注意力权重分布存在一定程度上的差异(图7)。其中两种边的注意力权重分布在注意力头2中是相似的,但在其他注意力头中却有显著不同。注意力头之间分布的多样性可能解释了为什么EquiScore在虚拟筛选和类似物排序场景中都表现出色。
在图7b-e中,作者使用了一个先导化合物优化案例展示了EquiScore的多级可解释性。有文献表明,甲基化后的噻吩环与附近疏水氨基酸残基ILE219之间的疏水相互作用是这一对同系物产生活性悬崖的主要原因。在图7b-c中,作者注意到引入甲基确实显著地改变了模型在原子级别的注意力权重,而且这种变化不仅局限于噻吩环的局部区域,还影响了整体的权重分布。同时,在图7d中,可以观察到甲基的引入使配体和蛋白质口袋在形状上更具互补性,使分子与口袋内疏水氨基酸残基ILE219和VAL49的碳原子距离更近。如图所示,碳-碳原子对之间的距离在3.5Å左右,可以形成良好的疏水相互作用。图7e中蛋白-配体相互作用的进一步可视化显示:该模型将较高的权重分配给了引入的甲基和ILE219上的碳原子,这表明该模型可以通过EquiScore层很好地捕获配体原子和受体原子对之间的相互作用。总之,基于注意力的可解释性分析可以帮助我们定位蛋白质上的关键氨基酸和配体官能团,为合理的药物设计和结构优化提供指导。
3 结论
在该工作中,作者通过整合物理先验知识和数据驱动建模来构建了一个通用的蛋白配体评分函数EquiScore。EquiScore在新靶标的药物虚拟筛选任务上展现出了良好的性能,同时还展示出一定的先导化合物优化的潜力。此外,EquiScore可以与不同的对接方法联用,均可显著提升不同对接方法的虚拟筛选能力,具有良好的兼容性。EquiScore还具有不同级别的可解释性,可以为基于结构的药物设计提供指导。
浙江大学与上海药物研究所联合培养博士研究生曹端华,国科大杭州高等研究院硕士研究生陈庚为本文的共同第一作者。上海药物研究所郑明月研究员为本文通讯作者。本研究得到了国家自然科学基金、国家重点研发专项、上海药物所与上海中医药大学中医药创新团队联合研究项目、中国科学院青年创新促进会会员项目、上海市科技重大专项资助。
https://www.nature.com/articles/s42256-024-00849-z
(点击下方“阅读原文”跳转)

内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢