
点击蓝字 关注我们

微末生物
Nanomega BioAI
民间有俗语:“龙生龙、凤生凤,老鼠的儿子会打洞”,“种瓜得瓜、种豆得豆”。高中生物遗传课本上又提到:“一猫生九崽,连母十个样”。这些谚语都体现了人们对遗传现象的理解和观察,说明了亲代和子代之间在特征、性格和能力上的相似性或差异性,也就是遗传的稳定性和变异性。
从生命的角度出发,遗传确保了父母的基因信息能够传递给后代,保证了种族的延续。基因变异导致生物多样性,提供了适应环境变化的能力。DNA作为遗传物质的载体,负责存储、传递和表达遗传信息,是生物遗传性状和进化发展的关键,它在细胞分裂中复制和传递遗传信息,通过基因表达调控细胞的生理过程,同时也是遗传变异和进化的基础。
对DNA结构和功能的深入理解,推动了遗传学、分子生物学和生命科学领域的不断进步,为人类探索很多生命的奥义提供了重要的科学依据。

DNA复制涉及前导链的连续复制和滞后链冈崎片段的不连续合成。DNA复制体是一种多蛋白质机器,可在两条链上执行协调的亲本链分离和RNA引发的DNA合成。解旋酶通过将DNA解旋与前导链聚合酶和滞后链引物酶的催化耦合起来,成为所有复制体的核心。滞后链聚合酶通常在冈崎片段合成开始时连接到复制体上。前导链和滞后链合成虽然方式不同,但在复制过程中以相似的速度进行。复制体不可避免地会遇到各种障碍,需要修复和同源重组来挽救受阻碍的复制叉。
尽管复制体很重要,但它从未在原子分辨率下被可视化。美国国立卫生研究院杨薇教授团队与加州大学洛杉矶分校的周正洪教授等合作在SCIENCE上发表文章Structures and operating principles of the replisome,使用噬菌体 T7 作为模型系统,以高达 3.2 Å的分辨率确定了沿着 DNA 易位的解旋酶的冷冻电镜结构,以及参与两条 DNA 链合成的解旋酶-聚合酶-引物酶复合物。螺旋形六聚体解旋酶的每个结构域沿着 ssDNA 线圈依次手拉手易位,类似于 AAA+ ATP酶展开肽的方式。两条滞后链聚合酶连接到引物酶上,准备串联进行冈崎片段合成。
来自前导链聚合酶的β-发夹将两条亲本DNA链分开成 T 形叉,从而使紧密耦合的解旋酶能够垂直于下游 DNA 双链体前进。这些结构揭示了复制体的分子组织和操作原理。
一、“初来乍到”的解旋酶
所有解旋酶都是由六个亚基组成的ATP酶,属于RecA样或AAA+家族,并且能够沿5'至3'或3'至5'的方向移动。尽管它们的ATP酶位点都位于亚基之间,并且单链DNA都通过中央通道结合,但这些解旋酶的整体结构和DNA结合方式却有所不同。例如,乳头瘤病毒E1解旋酶呈平面环形,并围绕狭窄的6ntDNA线圈进行结合,类似于包裹RNA的大肠杆菌Rho转录终止子。然而,无论有无DNA,E1解旋酶的结构都保持不变。
相比之下,细菌DnaB解旋酶则呈锁垫圈形状,围绕14ntDNA线圈进行结合,并且在解旋DNA时,每个亚基可以从锁垫圈的一端移动到另一端。真核生物的CMG解旋酶与DNA的结合方式与DnaB相似,但只与半螺旋中的6-8nt与六个亚基的一个子集接触,其具体工作机制尚不明确。
此研究探索了噬菌体T7中相对简单的DNA复制系统。T7解旋酶是一种类似于RecA的ATP酶,能够在滞后链上移动,并且能够使用dTTP,从而提高持续加工能力。确定了T7 DNA聚合酶-Trx-DNA-dGTP复合物和gp4片段(包括锌结合域的引物酶、单独的解旋酶和带有解旋酶的引物酶)的结构,此外还报道了gp4-gp5复合物的结构。这项研究获得了T7 DNA复制体的作用快照,并探讨了其与大肠杆菌和酵母复制体的相似之处,这有助于我们更深入地理解DNA复制过程中解旋酶的工作原理和机制。
二、gp4-DNA复合物在冷冻电镜下的“真面目”
5'端附近具有引物酶识别位点的25-nt ssDNA被用作gp4的解旋酶和引物酶的底物。已知E343Q突变体的gp4缺乏NTP水解活性并具有增加的DNA亲和力,在dTTP存在的情况下很容易在ssDNA上形成六聚体。
纯化后,在提供ATP、CTP和Mg2+的环境下,gp4-DNA-dTTP复合物能够在DNA链上合成RNA引物。使用3'-脱氧-CTP产生终止的pppACd二核苷酸。
随后添加CTP并没有产生更长的产物,而是生成了稳定的gp4-DNA/pppACd-CTP/dTTP复合物或gp4-DNA复合物,最终这种gp4-DNA复合物的冷冻电镜结构以3.2Å分辨率测定。
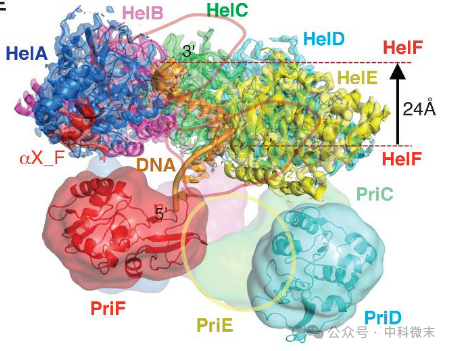
图1: gp4-DNA 复合物的组装和结构
(A、B、C、D、E 和 F 亚基和 DNA 采用颜色编码。显示的半透明冷冻电镜密度没有任何解旋酶结构域过滤器,并且对于引物酶结构域低通过滤至 8 Å。HelF 的易位由其起始位置 (gp4-DNA-I) 和结束位置 (gp4-DNA-III) 的半透明红色轮廓表示。PriE 用黄色轮廓表示。)
三、解旋酶与引物酶的构象及作用
引物酶和解旋酶形成的双层结构中,解旋酶构成了一个锁垫圈,围绕着右手单链DNA螺旋。进一步的三维分类发现解旋酶的第六个亚基HelF有三种不同的构象(gp4-DNA I-III)。在一个构象中,HelF在HelE后面结合到DNA的5'端,而在另一个构象中,HelF向前移动并在HelA之前结合到DNA的3'端。
这表明解旋酶亚基之间可能存在一种“手拉手”的易位机制。引物酶的催化结构域连接在不同的密度区域,其中与DNA 5'端结合的PriF区域的电镜密度最大。这可能是因为PRS/pppACd/CTP和gp4的ZBD与PriF结合,导致体积增大。每个解旋酶结构域都包含一个dTTP·Mg2+,其碱基由Y535和R504的侧链夹持,与ATP的腺嘌呤类似,而WalkerA基序与三磷酸相互作用。解旋酶结构域界面的R522残基接触dTTP的γ-磷酸盐,Mg2+则与dTTP的β-和γ-磷酸盐以及S319和E343Q的侧链配位。D424来自WalkerB基序,可能与Mg2+有水介导的相互作用。
有趣的是,在HelDE界面处的dTTPγ-磷酸附近观察到亲核水分子,它可能有助于推动HelE的易位。这个水分子由Q494主链和H465侧链协调,与DNA结合的LoopD1和LoopD2上的残基相邻。H465突变消除了dTTPase活性的DNA刺激。因此,gp4-DNA结构揭示了DNA结合和NTP水解之间的直接联系。
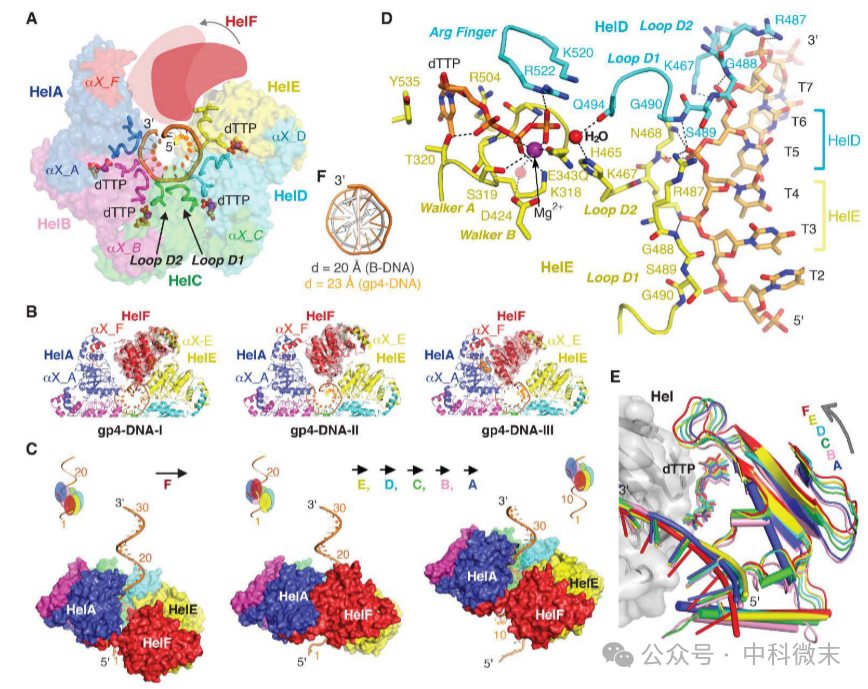
图2: 解旋酶易位机制
(A)从 DNA 5' 端观察 Gp4-DNA 复合物。显示了 HelF(浅红色)的开始位置(深红色)和结束位置。 (B) HelF 易位。gp4-DNA-I至III的结构从左到右显示。HelF 和 HelE 连接体的冷冻电镜密度显示为半透明表面。 (C) 解旋酶易位图。dTTP 水解并从 HelEF 界面释放,在 5' 端释放 HelF。HelF 向 3' 端易位后,新形成的 HelFA 界面获得新的 dTTP。E 亚基依次易位至 A 亚基,每个亚基均朝向 3' 端,完成 12 个核苷酸的螺旋转动。 (D) HelDE 接口处的 NTPase 活性位点及其与 LoopD1 和 LoopD2 与 DNA 结合。 (E) 解旋酶亚基界面的逐渐变化。Hel 二聚体由其 3' 亚基叠加(显示为灰色表面和带状),5' 亚基用不同颜色表示变化顺序,对 (A) 中做了细化。(F) gp4及其上的ssDNA(橙色,直径更大)和 标准B 型 DNA(青色)复合的叠加。
四、滞后链聚合酶与gp4引物酶的结合
为了捕捉滞后链DNA合成的起始阶段,研究人员设计了含有44个核苷酸的单链DNA和短的DNA/RNA混合底物。他们通过冷冻电镜技术获得了四种gp4-gp5 DNA/RNA复合物(LagS1-4)的结构,分辨率大多在4埃(Å)左右。
在这些结构中,聚合酶-DNA/RNA-dTTP三元复合物均以相似方式连接到两个相邻的引物酶结构域EF、AB或BC,最多能连接三个滞后链聚合酶。在LagS4结构中,观察到两种DNA聚合酶附着在引物酶结构域上:一种附着在含有DNA/RNA底物的PriAB上,另一种附着在没有底物的PriCD上,似乎在等待下一个引物的到来。
在没有DNA的gp4-gp5结构中,也观察到邻近引物酶结构域的两个DNA聚合酶,尽管它们被认为分别参与前导链和滞后链的合成。在3.7 Å分辨率的LagS1-gp5-DNA结构中,gp5聚合酶与PriE和PriF相互作用。其中PriE在没有gp5聚合酶情况下参与引物合成。PriE与聚合酶拇指结构域上的Helix H形成广泛的极性和疏水接触,而聚合酶与PriF之间的相互作用较弱,涉及聚合酶的螺旋E'尖端和PriF的R77和T82。gp4-gp5相互作用稳定了两个引物酶结构域,并使得引物酶和解旋酶之间的柔性铰链变得可追踪。
此外,gp4的N末端ZBD通过20个无序残基与引物酶结构域连接,支撑RNA引物的5'端并被聚合酶包围。ZBD的Y37侧链与第一个DNA/RNA碱基对堆叠,H14侧链也接近该碱基对。PRS中的第一个核苷酸dC被隔离在口袋中并与H33接触。ZBD的突变会阻碍引物合成和聚合酶的延伸,而H33A突变则改变了PRS的偏好序列。这些结构不仅揭示了ZBD如何结合PRS并稳定短DNA/RNA杂交体,还展示了柔性ZBD如何作为DNA模拟物促进引物合成、传递和延伸,同时不妨碍解旋酶的运动。类似的机制也可能出现在真核生物的引物酶和聚合酶之间,尤其是在前导链合成的开始和重新引发时。
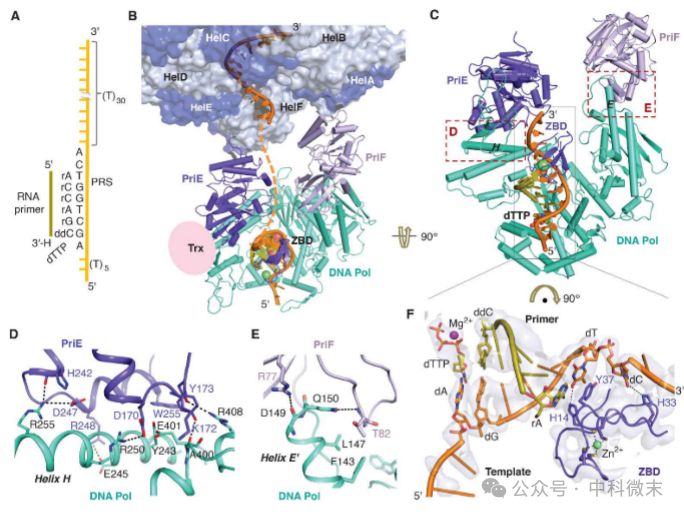
图3: 滞后链 gp4-gp5 复合物的结构
(A) DNA/RNA 底物。(B) gp4的结构。(C) gp4 引物酶、gp5 聚合酶、DNA/RNA 杂交体和 dTTP 复合物的放大视图。(D和E) (C) 中的聚合酶和引物酶界面的放大视图。可能的相互作用如虚线所示。(F) DNA 模板 (PRS) 和 RNA 引物的 5' 端由 gp4 ZBD 稳定。冷冻电镜密度叠加为灰色半透明表面。ZBD 和 DNA/RNA 之间的相互作用用虚线表示。
五、DNA解旋和前导链合成是如何协调一致的?
本研究首先利用冷冻电镜技术深入探究了T7噬菌体复制其DNA时,两个主要蛋白——gp4解旋酶和gp5聚合酶——是如何协同工作的。这一发现对于理解DNA复制机制具有重要意义。
在DNA复制过程中,gp4解旋酶负责解开DNA双螺旋结构,使得gp5聚合酶能够读取其中的遗传信息并合成新的DNA链。研究发现,这两个蛋白在DNA分叉处紧密结合,形成一个高效的复制体。通过冷冻电镜分析,研究人员获得了不同分辨率的复合物结构,揭示了gp4和gp5的精确位置和互动方式。这些结构显示,gp4形成一个六聚体,与DNA叉紧密结合,而gp5聚合酶则以不同方向接触解旋酶结构域。这种动态的相互作用使得复制体能够灵活地移动并适应DNA的形状变化。
同时,研究人员还发现了一些有趣的细节,比如某些氨基酸残基在与DNA结合时的特定位置和构象。此外,研究还表明,这两个蛋白之间的相互作用对于DNA复制的效率和准确性至关重要。通过改变某些氨基酸残基或干扰蛋白间的相互作用,可以影响复制体的功能和DNA合成的结果。这为未来设计针对病原体DNA复制过程的药物或基因编辑工具提供了潜在的靶点。
总之,本研究不仅揭示了T7噬菌体DNA复制过程中解旋酶和聚合酶的协同工作机制,还为理解其他生物体中DNA复制和修复机制提供了重要线索。这些发现对于发展新的治疗方法和技术具有重要意义。
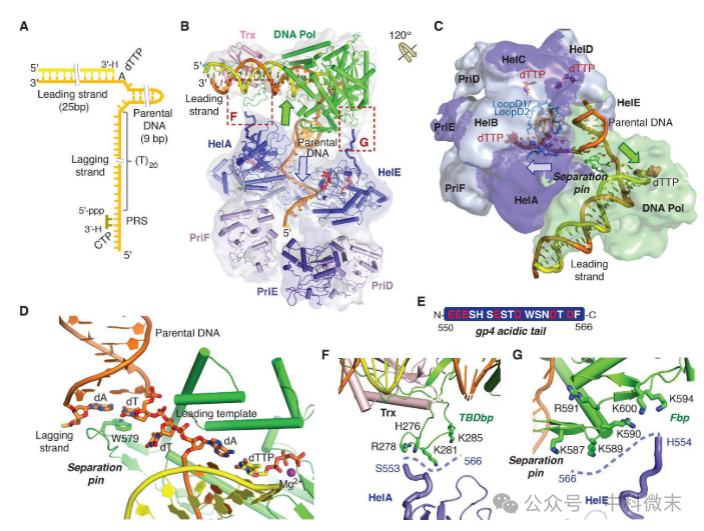
图4: 前导链 gp4-gp5 复合物的结构
(A) DNA 叉底物。(B 和 C) gp4-gp5-DNA 叉结构。聚合酶(绿色)、Trx(浅粉色)和解旋酶(深蓝色和浅蓝色交替)的卡通图以冷冻电镜密度(半透明)显示。为了清楚起见,省略了 (B) 中 HelF 的蛋白形态和 (C) 中所有蛋白质的结构细节。dTTP 和 DNA 结合环分别在 C 中显示为红色和蓝色棒。绿色和蓝色箭头表示聚合酶和解旋酶解旋 DNA 的方向。(D) 被前链聚合酶结合的 DNA 叉的放大视图。交界处的核苷酸显示为球棍结构。分离销(separation pin)和 W579 与亲本 DNA 的第一个碱基对堆叠。(E) gp4 酸性尾的序列。(F 和 G) gp4 引物酶和 gp5 聚合酶界面的放大视图,如 (B) 中所示。
六、复制体的组织架构
研究人员使用相同的DNA叉底物,还成功获得了四个滞后链复合物(LagL1-4)。这些复合物显示滞后链聚合酶连接到引物酶的不同结构域。与短RNA/DNA杂交体相比,长DNA底物下持续因子Trx更有序,Helix H引物酶的相互作用也增强了。LagL复合物可能由两种蛋白形成,分别与不同的DNA叉底物结合。冷冻电镜图像显示,完整的复制体颗粒中,前链聚合酶与解旋酶结构域接触,引物酶一侧有一个或两个后链聚合酶。尽管颗粒数量有限,且组合多样,但仍未能获得从头开始的复制体结构。
然而,通过叠加保守的gp4解旋酶部分,研究人员构建了一个复制体模型。在这个模型中,gp4夹在前导链和滞后链聚合酶之间,复制叉位于前导链聚合酶和解旋酶之间,由两者共同推动。滞后链DNA首先被引物酶捕获,然后转移到滞后链聚合酶。多个聚合酶与引物酶一侧的结合确保了冈崎片段的平行合成以及前导链和滞后链合成的相似速度。这些发现揭示了T7噬菌体复制体的组织原则,与细菌和真核生物的DNA复制细节相似。
尽管在细菌中解旋酶-引物酶关联是分子间的,并由Tau蛋白介导,而在真核生物中,CMG解旋酶沿前导链易位,但CMG解旋酶仍然是复制体的中心组织者。当CMG和Pol都沿前导链DNA移动时,复制体可能忽略并跳过滞后链上的损伤。最新数据表明,CMG帮助DNA聚合酶穿越病变和障碍,并共同保持复制叉向前移动。
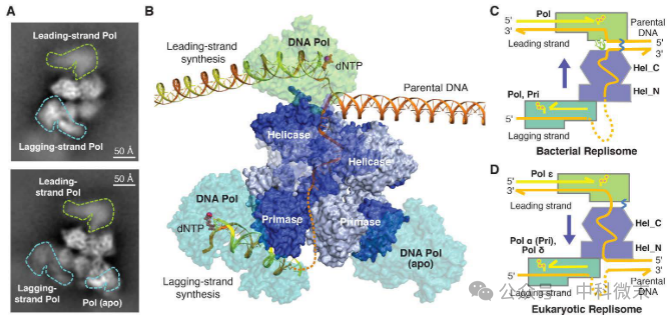
图5: T7 复制体的架构
(A) 代表性的二维分类复制体结构。聚合酶以绿色和青色勾勒出轮廓。(B) 通过重叠保守的 gp4 解旋酶结构域获得由前导链 (Lead1) 和滞后链复杂结构 (LagL1 和 LagS4) 组成的复制体模型。(C 和 D) 分别在滞后链 (C) 和前导链 (D) 上带有解旋酶的细菌和真核复制体图。在每种情况下,解旋酶易位的方向都用蓝色箭头标记。
小结:
总结而言,本研究展示了简单复制体的原子分辨率结构,说明了复制解旋酶如何在垂直于 DNA 叉的方向上易位,以及前导链和滞后链 DNA 合成如何在解旋酶的前侧和后侧发生。复制体组件之间观察到的灵活连接描绘了一种高度动态的组织,但具有细菌和真核复制体中保守的操作原理,它不仅复制基因组DNA,而且还检测DNA损伤并协调应激反应。详细的复制体结构解释了许多先前发表的结果,并为理解DNA复制、重组和修复之间的协调提供了基础。
中科微末团队在生物大分子及其复合体的三维结构解析方面技术成熟、经验丰富,能够在原子级分辨率解析多种生物大分子材料的三维结构。我们致力于为药企和高校医院科研工作者提供一站式、自动化、原子级分辨率的结构解析服务,助力创新药物研发和科研成果转化,帮助科研人员实现“原子结构自由”。
内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢