DRUGAI
今天为大家介绍的是来自西湖大学李子青团队的一篇论文。蛋白质-蛋白质结合在多种基本生物过程中起着关键作用,因此预测氨基酸突变对蛋白质-蛋白质结合的影响至关重要。为了应对注释突变数据稀缺的问题,利用大量未标注数据进行预训练已经成为一种有前景的解决方案。然而,这一过程面临一系列挑战:(1) 尚未完全捕捉到多个(不止两种)结构尺度之间复杂的高阶依赖关系;(2) 很少研究突变如何改变周围微环境的局部构象;(3) 预训练在数据规模和计算负担方面成本高昂。在本文中,作者首先构建了一个分层提示代码簿(hierarchical prompt codebook),独立记录不同结构尺度下常见的微环境模式。然后,作者开发了一种新颖的代码簿预训练任务,即掩码微环境建模(masked microenviroment modeling),用于模拟每个突变与其残基类型、角度统计和微环境中局部构象变化的联合分布。通过构建的提示代码簿,作者将每个突变周围的微环境编码为多个分层提示,并将它们结合起来,灵活地为野生型和突变蛋白复合物提供关于其微环境差异的信息。这种分层提示学习框架在突变效应预测和针对SARS-CoV-2优化的人类抗体的案例研究中,表现出优于最新预训练方法的卓越性能和训练效率。

蛋白质通常通过与其他蛋白质相互作用来执行特定的生物功能,这对于所有生物体都是必不可少的。一个主要的例子是抗体,这是一类由免疫系统产生的Y形蛋白,用于识别、结合和与病原体表面的蛋白质相互作用。因此,如何开发方法来调节蛋白质-蛋白质相互作用已成为一个关键问题,而最普遍的策略之一是突变相互作用界面的氨基酸。考虑到超过2030种氨基酸突变的巨大组合空间和突变结构的高变异性,在实验室中测试所有潜在突变是不现实的,这需要计算方法通过预测突变后蛋白质复合物的结合亲和力变化来筛选出理想的突变。这一问题,也被称为结合自由能变化(∆∆G)预测,是蛋白质复合物设计中的核心挑战。
用于∆∆G预测的计算方法已经经历了从基于生物物理和统计技术到深度学习技术的范式转变。尽管基于深度学习的方法取得了巨大进展,但注释实验数据的稀缺性和突变复合物结构的不可用性仍然是有效监督学习的两个主要挑战。因此,利用大量未标注数据进行预训练正成为∆∆G预测中最普遍的策略之一。
尽管取得了丰富的进展,现有基于预训练的方法仍面临几个关键问题。首先是忽视了对多种结构尺度及其依赖关系的建模。蛋白质在执行特定功能时可以聚焦于不同的结构尺度,每种结构尺度都有其独特的优势,无法相互替代。此外,不同结构尺度之间的依赖关系多种多样,仅使用现有预训练任务聚焦于单一或成对的结构尺度,无法完全捕捉它们复杂的高阶依赖关系。第二个障碍是缺乏突变后的复合物结构。尽管Alphafold2(AF2)和ESMFold在蛋白质结构预测方面取得了巨大进展,但它们在预测氨基酸细微突变导致的精确构象变化时仍存在困难。而且,研究发现,用实验结构训练的模型在测试预测的AF2结构时性能显著下降。此外,现有预训练任务中由于大量数据而产生的计算成本过高,甚至远超∆∆G预测任务本身。例如,SKEMPI v2.0数据集中只有7k标记的突变数据,但RDE用于预训练的PDB-REDO数据集包含超过143k的数据。
在本文中,作者提出了一种简单而有效的微环境感知分层提示学习框架,用于高效的∆∆G预测(Prompt-DDG)。Prompt-DDG的核心理念是避免计算量大的预训练,而是直接以轻量且高效的方式为每个突变生成简明的提示。这些提示旨在表征野生型和突变复合物之间突变周围的微环境差异。为了使生成的提示能够充分覆盖微环境的不同结构尺度的多样性,作者构建了一个分层提示代码簿,分别记录不同结构尺度的常见微环境模式。然后提出了一种新颖的代码簿预训练任务,即掩码微环境建模,用于模拟每个残基突变及其异质特性(包括残基类型、角度统计和微环境中局部构象变化)的联合分布。使用分层提示代码簿,作者将每个突变周围的微环境编码为多个提示,并通过一个轻量模块传递这些提示,为野生型和突变复合物灵活提供关于其微环境的多尺度结构信息。最终,Prompt-DDG在效果和效率方面均优于其他领先方法,如图1所示。
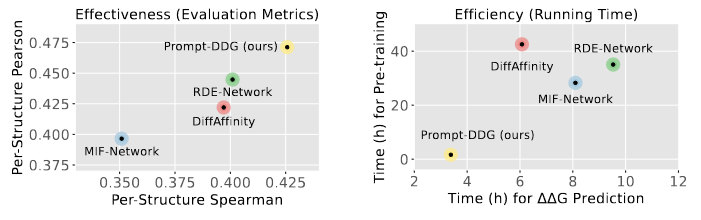
图 1
模型部分
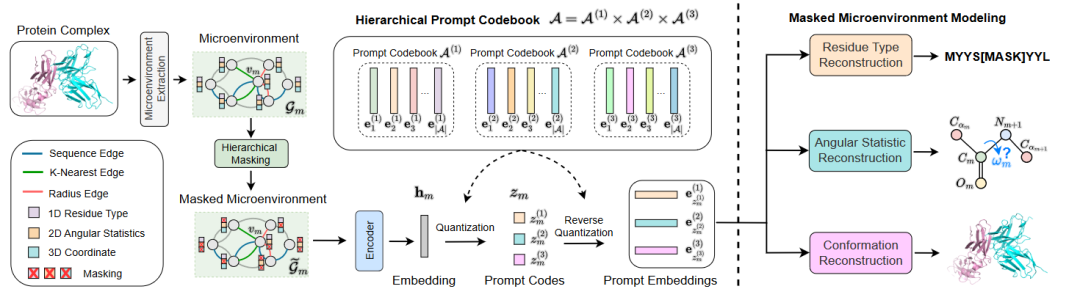
图 2
作者提出了一个用于∆∆G预测的Prompt-DDG框架,该框架包含三个新颖的组成部分。其流程如图2所示。具体来说,第一个组成部分构建了一个分层提示代码簿,将每个突变周围的微环境编码为不同结构尺度的提示;第二个组成部分通过掩码微环境建模对提示代码簿进行分层预训练;第三个组成部分采用轻量提示适应模块,结合不同尺度的提示,以提供微环境的差异。
残基的微环境描述了其周围的序列和结构背景。作者遵循Wu等人的方法,将每个突变的微环境定义为蛋白质复合物图G的一个-ego子图,其中其节点集合定义如下。其中,和是距离阈值,和为中心碳原子的3D坐标,而是残基空间中的k-hop邻域。
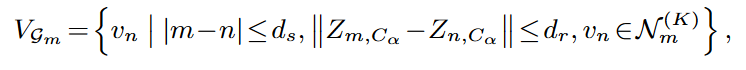
为了充分捕捉微环境的不同结构尺度,作者构建了一个分层提示代码簿,其中,,分别对应三种微环境(残基种类、角度、局部构象)。每个子代码簿由|A|个可学习的prompt表征组成。为了生成不同结构尺度的微环境感知提示,作者首先使用基于自注意力的图神经网络,将微环境编码为一个对旋转和平移不变的隐向量。接下来,使用矢量量化,将|M|个突变的微环境标记为离散提示码,通过在分层代码簿A中查找最近邻居进行量化。
为了解决矢量量化的不可微性问题,作者通过直通估计器施加了一个约束,以桥接代码簿A和微环境表示,其定义如下所示。其中η是一个可权衡的超参数,sg是截断梯度操作。
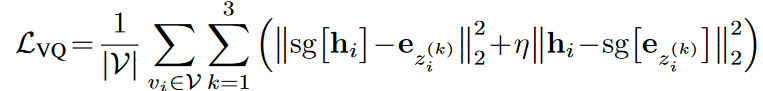
上式中的第一项为代码簿损失(codebook loss),用于更新代码簿,使微环境表示接近最相似的提示嵌入。第二项是承诺损失(commitment loss),仅训练编码器,鼓励编码器输出保持接近所选的提示嵌入。
接下来,作者聚焦于如何在构建的分层代码簿A中预训练可学习的提示嵌入。为此,作者采用三种不同的数据重建任务,同时学习分层代码簿A并训练微环境编码器。我们通过个体重建任务对每个子代码簿进行分层训练,使其专注于特定的结构尺度。此外,为了充分捕捉各种结构尺度之间的高阶(超过单一和成对的)依赖关系,我们在一个预训练任务中统一分层训练,即掩码微环境建模。具体而言,我们通过随机翻转、归零和高斯噪声独立地掩码微环境中的残基类型、几何角度和构象坐标,然后通过三种不同的重建任务从掩码后的微环境中重建输入。这三个结构尺度的掩码残基集是独立的,分别表示为,和。对应的三个训练损失函数如下所示。
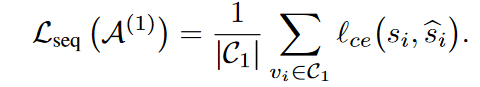
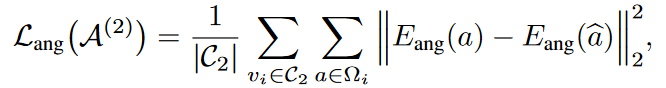
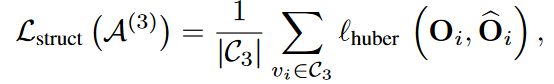
最终,层次训练提示代码簿的损失函数定义如下所示。
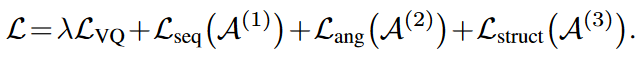
利用预训练好的代码簿A,可以将每个突变m的微环境编码为三个离散的提示代码。由于不同的子代码簿记录了微环境的不同结构尺度,作者灵活地结合获取的提示嵌入,以弥合预训练提示与下游任务之间的差距。提示组合由一个轻量级的提示适应模块实现,定义如下。
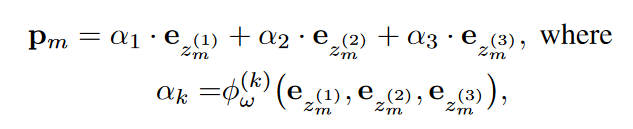
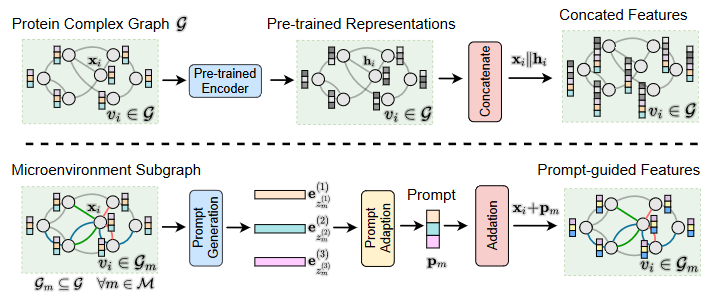
图 3
之前基于预训练的方法为每个残基学习一个预训练表示,并将其与对应的残基拼接(或相加)。相比之下,作者将每个突变周围的微环境编码为一个提示嵌入,然后将其添加到微环境内的每个残基上,如图3所示。接下来,作者使用与微环境编码器,相同架构的网络来转换提示引导的输入,并应用最大池化以获得全局结构表示。然后将突变体表示减去野生型表示,将其输入到一个MLP中以预测∆∆G,并计算其与真实值∆∆G之间的均方误差(MSE)损失作为最终目标函数。
在SKEMPI v2数据集上的实验性能
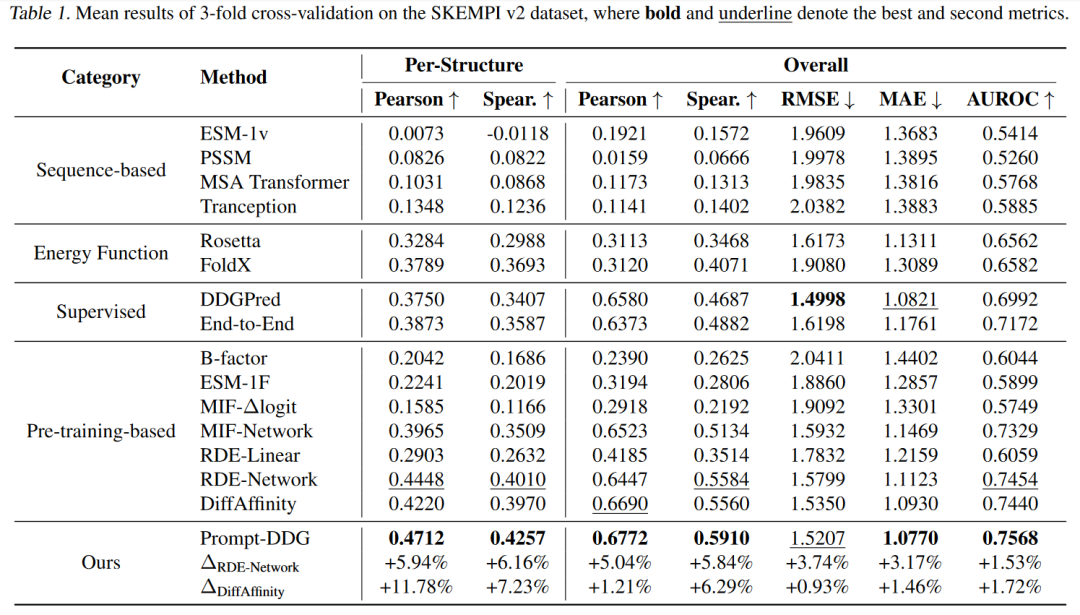
表 1
表1报告了SKEMPI v2.0数据集上15种方法的7个评估指标,以及Prompt-GPT相对于两个领先方法RDE-Network和DiffAffinity的相对改进情况。可以观察到:(1) Prompt-DDG在7个评估指标中的6个上表现优于所有基准方法。此外,它在RMSE指标上仅次于最先进的监督方法DDGPred,接近其水平。(2) 尽管未使用任何额外数据进行预训练,Prompt-DDG在所有7个指标上均超越了所有基于预训练的方法,这表明专门的微环境提示比从蛋白质预训练中获得的一般知识更有效。(3) 尤其值得注意的是,Prompt-DDG在两个最关键的指标上,即每个结构的皮尔逊和斯皮尔曼相关性,取得了最显著的改进,展示了其在实际应用中的更大潜力。
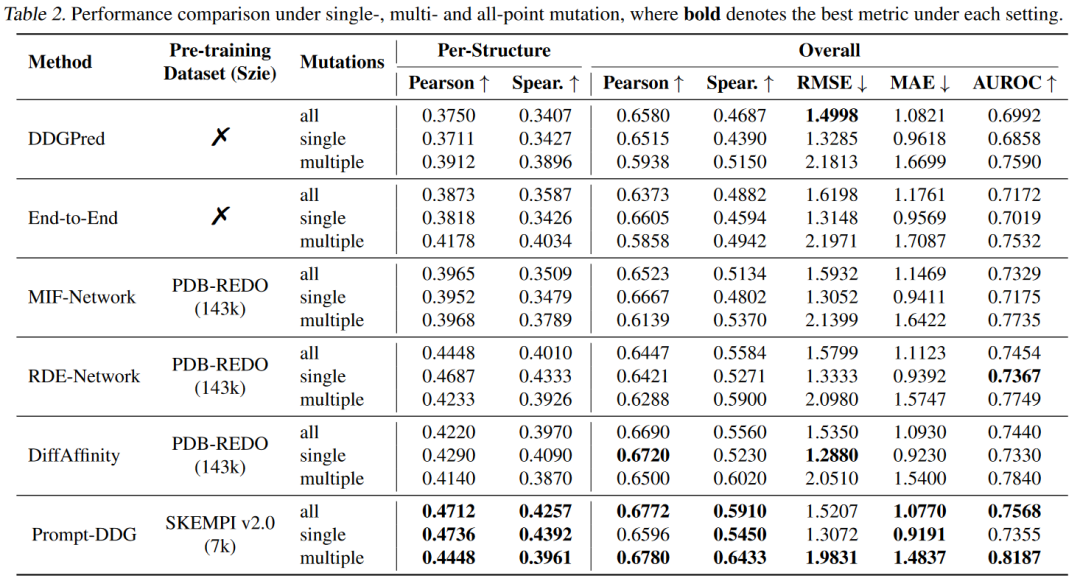
表 2
作者基于对7个指标的综合考虑,从表1中选择了五种表现较优的方法,并在单点、多点和全点突变下与Prompt-DDG进行比较。表2中的结果显示,在单点突变设置下,Prompt-DDG在7个指标中有4个排名第一,整体表现最佳。在实践中,通常需要突变多个氨基酸以达到所需的结合亲和力,这使得多点突变效应预测非常重要。特别是,在多点突变设置下,Prompt-DDG以较大优势超越了包括RDE-Network和DiffAffinity在内的所有其他基准方法。Prompt-GNN在多点突变上的优越性体现在两方面:(1) 它为每个突变周围的微环境单独生成提示,捕捉到了更细粒度的局部(而非全局)差异;(2) 具有多个突变的复合物的构象比单个突变的更为多变,Prompt-DDG擅长建模每个突变对其局部微环境构象的影响。
相关性分析的可视化

图 4
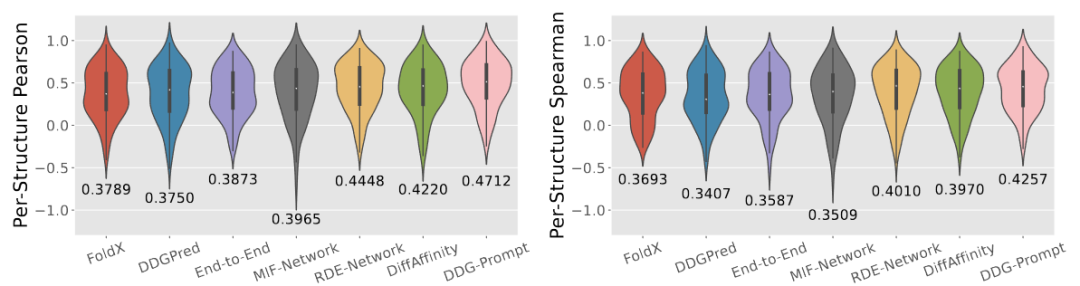
图 5
图4中展示了四种代表性方法(MIF-Network、RDE-Network、DiffAffinity和Prompt-DDG)的实验和预测∆∆G的散点图,以及它们的整体皮尔逊和斯皮尔曼相关性得分。可以看出,Prompt-DDG在定性可视化和定量指标上均优于其他三种方法。此外,作者在图5中提供了每个结构的皮尔逊和斯皮尔曼相关性得分的分布情况,以及所有结构的平均结果。作者发现,Prompt-DDG不仅在平均性能上表现最佳,而且其分布主要集中在高相关性,并且低相关性的结构较少。
编译 | 黄海涛
审稿 | 曾全晨
参考资料
Wu, L., Tian, Y., Lin, H., Huang, Y., Li, S., Chawla, N. V., & Li, S. Z. (2024). Learning to Predict Mutation Effects of Protein-Protein Interactions by Microenvironment-aware Hierarchical Prompt Learning. arXiv preprint arXiv:2405.10348.
内容中包含的图片若涉及版权问题,请及时与我们联系删除



评论
沙发等你来抢