今天为大家介绍的是来自David van Dijk团队和Rahul M. Dhodapkar团队的一篇论文。大型语言模型(如GPT)在自然语言任务中表现出色。在此,作者提出了一种新颖的方法,将这些预训练模型直接应用于生物学领域,特别是单细胞转录组学。作者的方法称为Cell2Sentence,它通过将基因表达数据表示为文本来实现这一点。具体来说,Cell2Sentence方法将每个细胞的基因表达谱转换为按表达水平排序的基因名称序列。作者展示了这些基因序列(“细胞句子”)可以用于微调因果语言模型,如GPT-2。关键的是,作者发现自然语言预训练提升了模型在细胞句子任务上的表现。当在细胞句子上进行微调时,GPT-2在给定细胞类型的情况下可以生成生物学上有效的细胞。相反,当给定细胞句子时,它也可以准确预测细胞类型标签。这表明,使用Cell2Sentence微调的语言模型可以获得对单细胞数据的生物学理解,同时保留其生成文本的能力。作者的方法提供了一个简单、适应性强的框架,可以使用现有的模型和库将自然语言和转录组学结合起来。代码可在以下网址获取:https://github.com/vandijklab/cell2sentence-ft。

大型语言模型(LLMs),如GPT,在自然语言处理任务(包括问答、摘要和文本生成)中展示了强大的能力。然而,将LLMs应用于其他领域(如生物学)仍然是一个开放的挑战。特别是,直接将现有LLMs应用于单细胞转录组学的方法,可以开辟分析、解释和生成单细胞RNA测序数据的新途径。目前,该领域的现有方法依赖于专门设计的神经网络,这些网络没有利用大型语言模型的预训练知识和语言理解能力。
模型部分
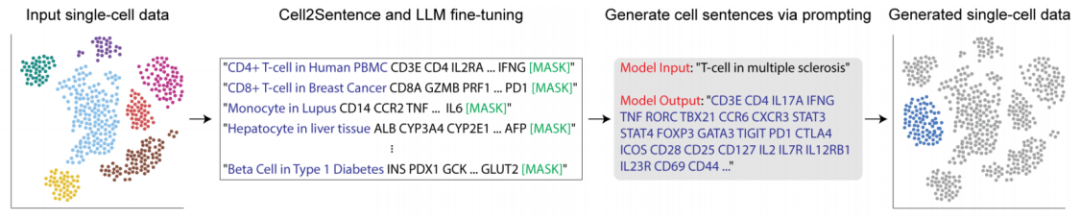
图1:Cell2Sentence整体框架
在这项工作中,作者旨在将大型语言模型(LLMs)的能力扩展到转录组学领域。本文的核心理念是通过一种称为Cell2Sentence(C2S)的方法,将单细胞数据表示为适合语言模型处理的文本格式。C2S将每个细胞的基因表达谱转换为按表达水平排序的基因名称(也称为基因符号)序列(如图1所示)。
数据转换
Cell2Sentence转换的核心是将细胞表达矩阵重新组织成按转录丰度排序的基因名称序列,类似于计数矩阵的排序转换。设C表示一个细胞基因计数矩阵,其中包含n个细胞和k个基因,Ci,j表示在细胞i中观测到的基因j的RNA分子数量。作者遵循单细胞RNA测序数据的标准预处理步骤,包括过滤掉表达基因少于200个的细胞以及在少于200个细胞中表达的基因。然后使用Scanpy Python库根据每个细胞中的线粒体基因计数计算质量控制指标,过滤掉含有超过2500个计数或超过20%转录计数来自线粒体基因的低质量细胞。接下来,将计数矩阵行标准化,使每个细胞的总转录计数为10,000,并进行对数标准化,得到最终的预处理计数矩阵C′。作者总结这一标准化步骤为:
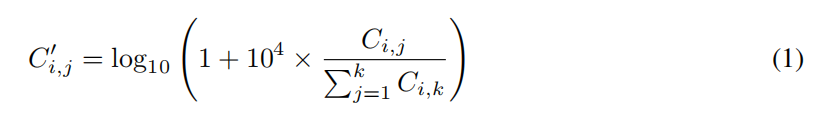
作者将应用于C′的排序转换表示为S,将排序转换后得到的基因名称序列称为每个细胞i的细胞句子si。实际上,作者对每个单细胞数据集分别应用预处理和排序转换S,提供了一种灵活的过程,将传统的单细胞基因表达计数矩阵转换为细胞句子。
作者通过从一个包含人类免疫组织细胞的大型数据集中抽样49,920个细胞来构建数据集。作者应用前述的标准化步骤并转换为细胞句子,然后将生成的细胞句子划分为训练集(39,936个细胞句子)、验证集(4,992个细胞句子)和测试集(4,992个细胞句子),并且附加了每个细胞的类型。为了限制计算成本,作者将每个细胞句子截断为只保留表达最高的100个基因。需要注意的是,这种截断操作最小化了排序的可变性,因为低表达基因的表达值更相似。
模型训练
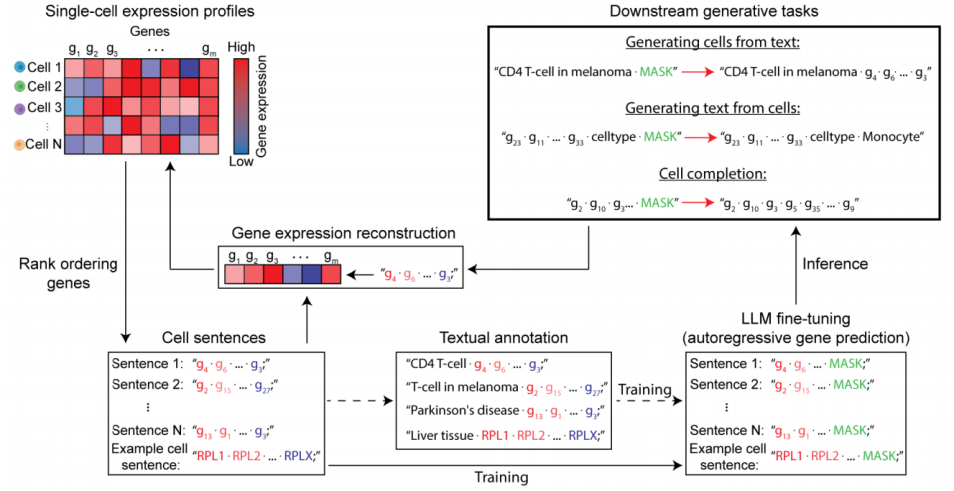
图2:Cell2Sentence的训练细节
作者为这项研究训练了四个GPT-2模型。具体来说,作者对GPT-2小型和中型模型都进行了预训练和微调,并使用细胞句子语料库。在这两种设置中,作者将输入格式化为提示,为模型提供学习的自然语言上下文(如图2所示)。作者遵循标准的训练配置并使用AdamW优化器。为了节省内存,作者采用了半精度浮点(FP16)和梯度累积。作者发现,使用全精度浮点(FP32)几乎没有带来性能改进,但速度却减慢了60%。

图6:在训练和生成过程中使用的三种类型的提示
作者制定了三个下游任务(见图6):
无条件细胞句子生成:生成一个由100个基因组成的序列,无需指定细胞类型标签。
条件细胞句子生成:在给定特定细胞类型标签的情况下,生成一个由100个基因组成的序列。
细胞类型预测:在提供的100个基因序列后生成一个细胞类型标签。
在训练阶段,每次迭代中,作者随机选择一个任务,然后从每个任务对应的大约20个模板中选择一个。尽管这些模板在措辞上有所不同,但语义上保持一致。细胞类型预测的提示结构将提示与细胞句子结合在一起。对于条件细胞生成,则将提示与指定的细胞类型结合。而无条件细胞生成的提示主要是简短的指令。
GPT-2小型模型初始化时有12层和768个隐藏单元,中型模型有24层和1024个隐藏单元。作者使用了6×10-4的学习率和余弦调度器,并采用1%的预热比例。对于GPT-2中型模型,采用累积16步梯度。小型和中型模型的有效批量大小分别为10和48个样本。每个模型在单个A5000 GPU上训练了两天。
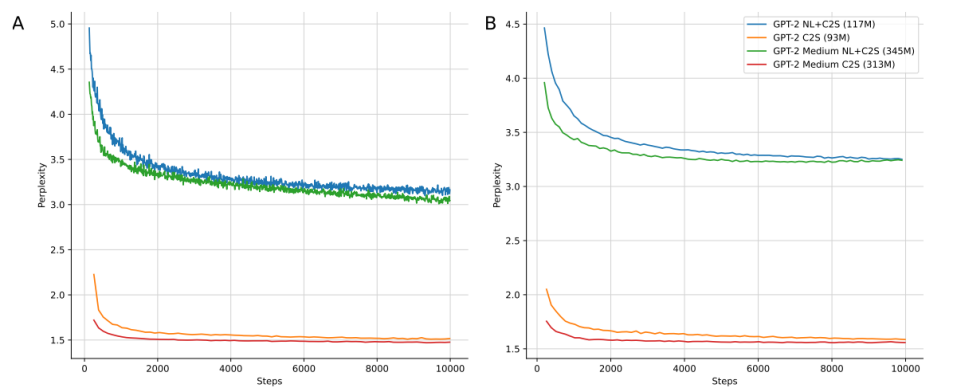
图5:微调模型和预训练模型的复杂度曲线
作者在完整的细胞句子数据集上训练了一个Byte Pair Encoding(BPE)分词器,包括自然语言提示和细胞类型标签,生成了9609个词汇表。训练集包含大约3000万个词汇,平均每个样本740个词汇。由于较小的嵌入空间,初始化模型的参数略少于在50,257个词汇表上预训练的对应模型(小型模型93M,中型模型313M,如图5所示)。由于提示的简短和重复,语料库中的自然语言词汇较为稀疏。尽管指令语料库传统上用于微调预训练模型以进行问答任务,但在预训练期间也采用了这种设置以与微调设置相似。作者假设提示模式中的语义变化可能会隐式地正则化词汇和位置嵌入,自然语言词汇起到了类标记词的作用。
表5:生成输出的质量
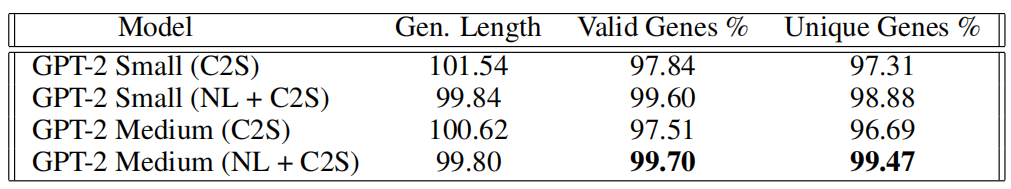
作者强调,损失是对提示和相关标签(即细胞类型)计算的。如果不这样做,会导致提示词汇的嵌入保持随机,从而削弱模型学习提示和标签词汇之间条件关系的能力。如图5所示,作者评估了模型生成有效基因并维持准确序列长度(此处为100个基因)的能力。结果显示两个预训练模型都能生成100个基因的序列且偏差不大,它们在基因有效性和唯一性上均超过97%和96%的准确率。
这两个模型使用Hugging Face模型库中的预训练权重初始化。作者使用5×10-5的学习率和线性调度器。对这两个模型,采用累积16步梯度并使用8个样本的批量大小(有效梯度更新批量大小为128个样本)。每个模型在单个A5000 GPU上训练。虽然作者尝试了应用高效微调技术(如LoRA),但完全微调的模型在基因唯一性和有效性评估中表现优于其他方法。作者特别发现LoRA在生成模式中表现高度可变,生成句子中的基因唯一性仅为70%。与预训练设置不同,作者在微调任务中采用仅对标签计算损失的传统方法。作者使用预训练的GPT-2分词器,平均每个训练样本约233个词汇(总共900万个训练词汇)。
作者使用序列长度以及基因有效性和唯一性来评估生成输出的一致性。作者发现微调后的模型在生成真实基因和句子内基因唯一性方面表现优异,平均生成基因的有效性超过99%,唯一性超过98%。虽然两个模型在这些标准指标上均表现出色,但微调后的模型在生成真实人类基因方面表现更加一致,在细胞句子中极少重复。
实验结果
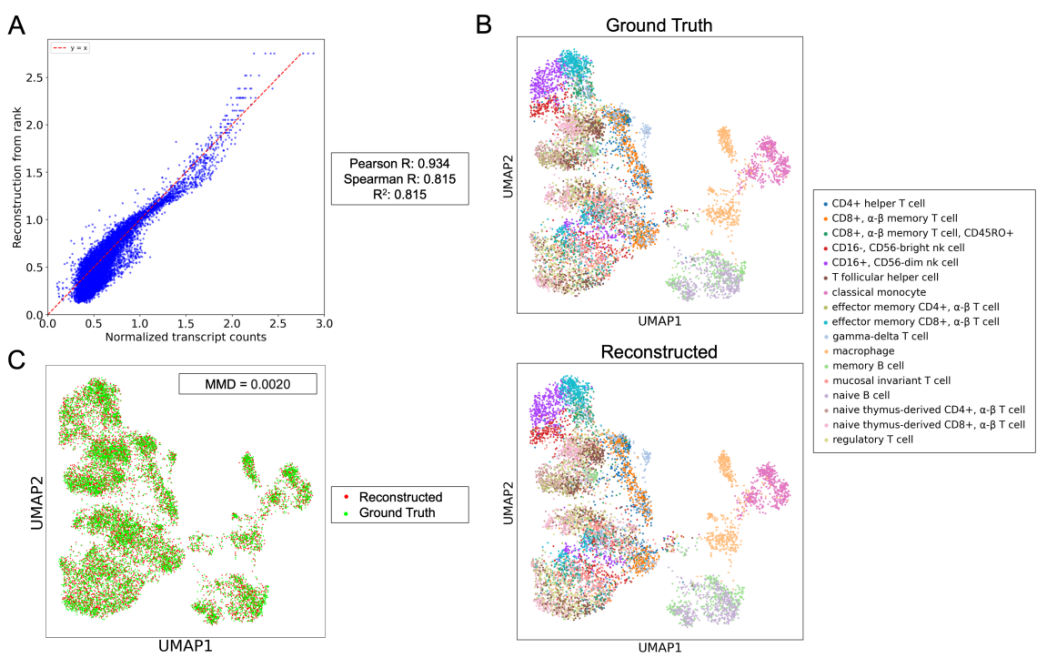
图3:Cell2Sentence转换可以准确地从细胞句子中重建细胞的表达
作者首先评估了C2S转换的有效性,以从细胞句子格式重建细胞的原始基因表达。图3A展示了在一个包含5万个细胞和超过3.5万个基因的免疫组织数据集上拟合线性回归的重建性能。一个简单的线性模型捕捉到了基因表达中超过81%的变异,仅需要该基因的对数排名值即可进行表达重建。这表明,将基因表达数据转换为细胞句子再转回表达空间,保留了单细胞数据中的大部分重要信息。这使得可以在细胞句子的自然语言空间进行分析,并随后准确地转换回表达数据。图3B和3C展示了原始的真实免疫组织数据与从转换后的细胞句子重建的表达数据。这从定性上显示了免疫组织数据中的重要结构以及细胞类型分离得到了保留。
表1:每个细胞类型的平均生成的细胞与原始表达式值的相关性度量
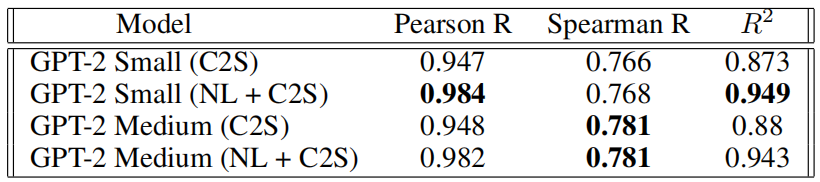
准确生成不同的细胞类型对于单细胞数据的生成方法至关重要,因为这使得可以使用模型进行下游分析。为了评估训练模型生成真实细胞的能力,作者考虑了免疫组织数据集中每种17种细胞类型的平均生成细胞,并在表1中将其与每种细胞类型的平均真实细胞进行比较。在17种不同的细胞类型中,微调后的模型生成的细胞与真实细胞表现出高度相关性,捕捉到了平均细胞表达中超过94%的变量。
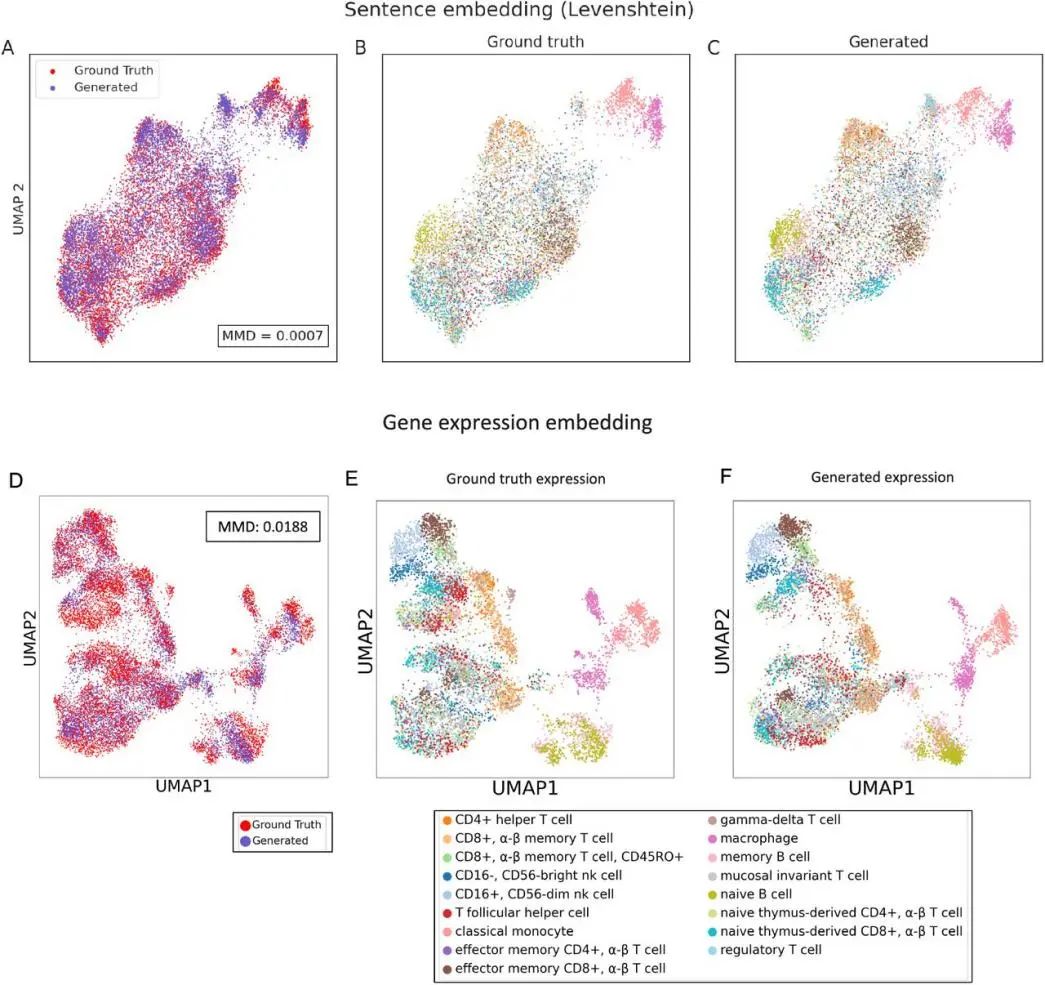
图4:单元格句子和表达式数据的绘图
图4中的UMAP图展示了通过表示具有最高表达的100个基因的细胞,可以实现多少分离。与通过选择高变异基因子集来减少基因表达矩阵大小的方法不同,作者的方法允许用最相关的特定基因对不同细胞进行编码,可能更好地表示稀有细胞类型,同时保持类似的压缩水平。此外,由于免疫数据集中某些细胞类型(例如T细胞)具有许多亚型,当仅限于表达最高的100个基因时,作者预计一些亚型会非常接近。
在图4C和4D中,UMAP图展示了从生成的前100个基因重建的基因表达不仅保持了原始数据的一般结构,还与基线UMAP可视化中的细胞类型特异性区分紧密对齐。这验证了该生成模型能够捕捉宏观和细粒度的表达谱,证明了其在创建具有生物学相关性的细胞表示方面的有效性。
表2:与真实数据相比的KNN分类精度结果
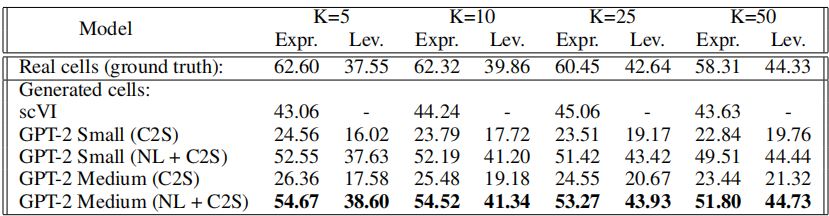
表3:分离不同细胞类型的KNN分类结果
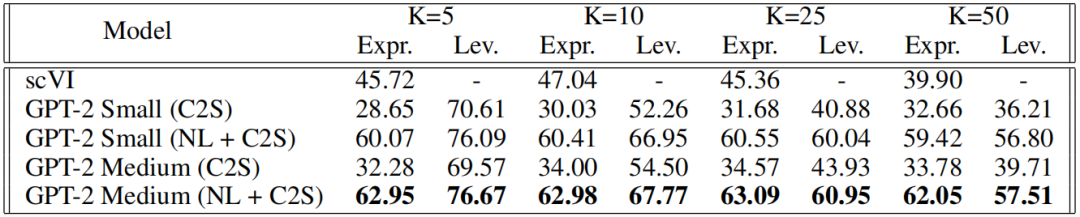
表2和表3显示了不同邻居数量下各模型的KNN准确率。由作者训练的模型生成的细胞在细胞类型上被KNN分类器准确分类,分类准确率最高可达54%。表3进一步量化了生成细胞的独特性,表明该模型能够通过自然语言生成不同的细胞类型。作者将结果与基于变分推断的单细胞数据生成模型scVI进行了比较。在生成质量方面,作者的模型优于scVI。
表4:对未知细胞的自回归细胞类型预测的定量研究
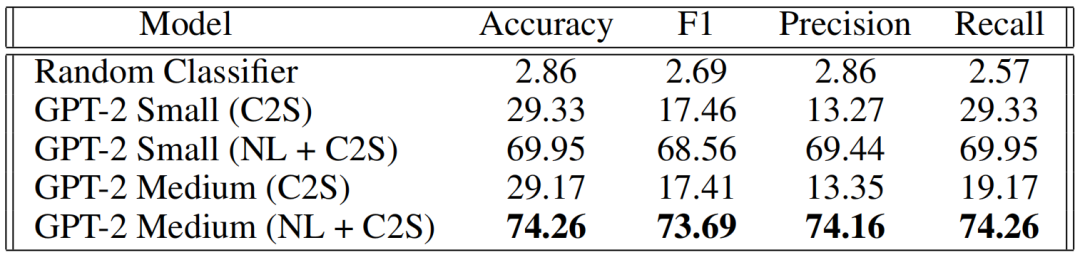
表4强调了自然语言预训练对于准确识别细胞类型的必要性。当使用未经自然语言预训练的模型时,观察到显著的性能下降,这证明了模型不仅仅是在记忆条件文本。尽管相对于预训练语料库,训练提示中的自然语言文本范围有限,并且允许模型在这些自然语言提示上进行训练,但没有进行自然语言预训练的模型未能获得有意义的自然语言嵌入。此外,随着预训练模型规模的增加,性能有适度提升。
编译|于洲
审稿|曾全晨
参考资料
Levine D, Lévy S, Rizvi S A, et al. Cell2sentence: Teaching large language models the language of biology[J]. bioRxiv, 2023: 2023.09. 11.557287.
内容中包含的图片若涉及版权问题,请及时与我们联系删除




评论
沙发等你来抢