前言
分子对接在现代药物开发中越来越重要,它可以高效模拟蛋白与配体的结合模式,提升药物筛选的准确性。为了帮助科研人员推进药物研发进程,英飞智药在其PharmaMind平台上新增了分子对接MolDock模块,让我们一起来探索这个新的工具。
MolDock简介
MolDock性能测评
一、MolDock-Sample对接采样召回率
图1对接采样测试结果显示,随着运行次数的增加,当运行300次时对接构象采样成功率(RMSD<2埃)达到了86.32%,即285个体系中有246个体系能对接成功。该结果表明MolDock-Sample采样方式能够比较好地复现出晶体的结合模式。
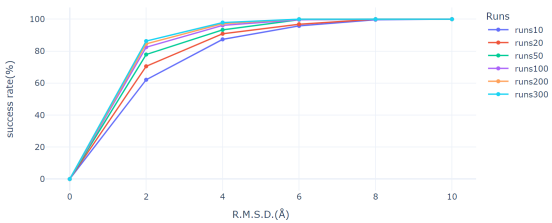
图1. MolDock采样在CASF-2016数据集上的测试结果。
二、MolDock-RefineScore虚拟筛选性能评估
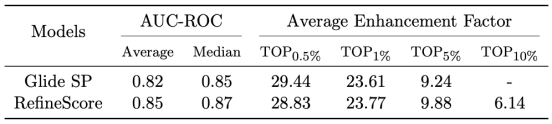
在药物设计领域,筛选能力是评估打分系统实用性的重要指标。通过在多个标准数据集(如CASF-2016、DEIKOS2.0 和 DUD-E)上的测评,我们将MolDock-Score的筛选性能与市场上的顶级软件进行对比,其中包括开源软件Vina以及商业软件Glide SP。表1/2/3数据显示,MolDock-RefineScore在这些数据集上的表现与广受认可的商业软件Glide SP(相关数据均为文献收集数据)相比,具备相当的竞争力,进一步证明了MolDock在药物虚拟筛选中的可靠性,为药物研发人员提供了一个有力的工具,帮助他们加速创新药物的发现与开发过程。
表1. Vina/Glide SP/RefineScore在CASF-2016标准数据集上的虚筛性能评测
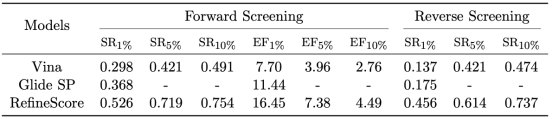
表2. Glide SP/RefineScore在DEKOIS2.0标准数据集上的虚筛性能评测
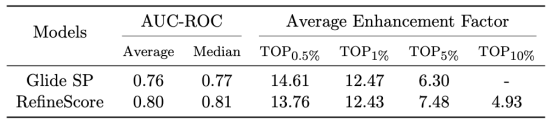
表3. Glide SP/RefineScore在DUD-E标准数据集上的虚筛性能评测
三、MolDock-Score打分能力和排序能力测试评估
CASF-2016评价体系中包含了打分能力(Scoring power)和排序能力(Ranking power)。图2的结果反映了目前MolDock-RefineScore目前也具备了领先的打分能力和排序能力。
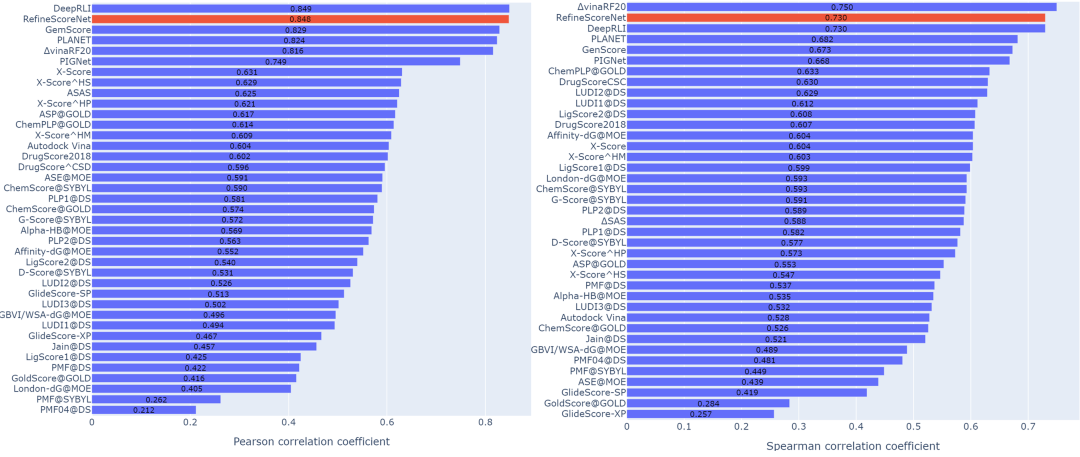
图2. MolDock-Score在CASF-2016数据集上的打分能力评估(左图)和排序能力评估(右图)。
四、案例展示:晶体结构复现
以目前比较热门的GLP-1R靶点的6XOX晶体结构为例,进行小分子结合构象复现。
在我们在PharmMind平台上使用MolDock,直接输入小分子的SMILES字符串:Cc1cc(cc(c1F)C)n2c(c3c(n2)CCN([C@H]3C)C(=O)c4cc5cc(ccc5n4[C@]6(C[C@@H]6C)C7=NOC(=O)N7)[C@H]8CCOC(C8)(C)C)N9C=CN(C9=O)c1ccc2c(c1F)cnn2C。使用默认参数进行重对接复现晶体结构(需要把Filter setting部分的分子量范围提高到900,避免该分子被过滤掉),结果如图3所示。
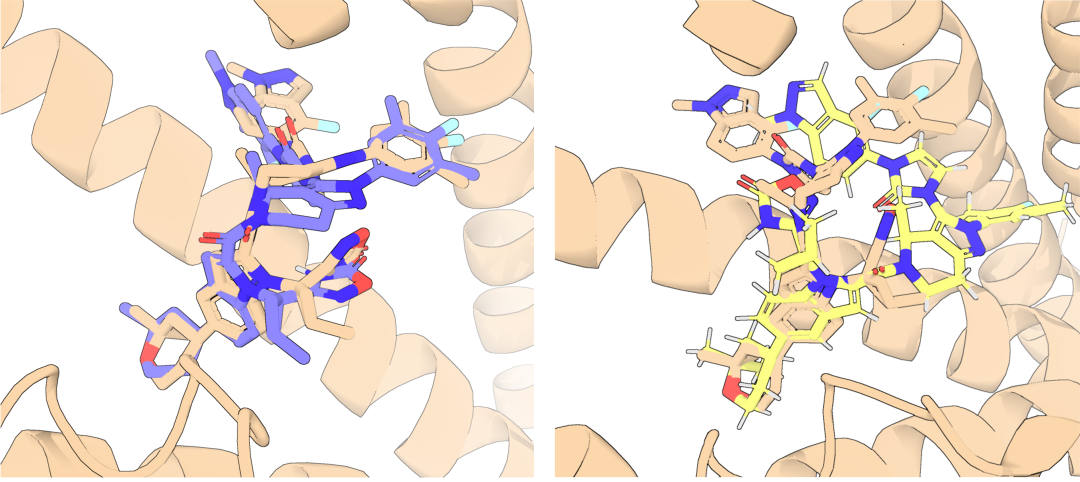
图3. Orforglipron对接结果比较图。
左图是MolDock的对接结果(浅杏黄色是原始晶体构象,紫色是对接最优构象,RMSD = 1.34 Å),右图是其他合作单位用Glide SP的默认参数对接结果(浅杏黄色是原始晶体构象,黄色是对接最优构象,RMSD = 4.90 Å)。
MolDock目前已支持功能
•蛋白准备模块:补全残基,替换非标准残基,加氢,氢键网络优化,能量最小化(需要开通企业账号),自定义口袋;
•配体准备模块:质子化状态、异构体生成、三维构象生成、分子类药性过滤,支持smiles/sdf的分子上传;
•对接配置模块:支持RefineScore,对接构象筛选。
总结
内容中包含的图片若涉及版权问题,请及时与我们联系删除





评论
沙发等你来抢